|
永野 聡一郎 先生 公益財団法人かずさDNA研究所 植物ゲノム・遺伝学研究室 |
QuantSeq 3'mRNA-Seq Library Prep Kit で行う多検体の網羅的遺伝子発現解析
ユーザーレポート
Product
メーカー:Lexogen GmbH メーカー略号:LEX
■ QuantSeq 3' mRNA-Seq Library Prep Kit (FWD)
イルミナ社機器に対応!
網羅的遺伝子発現解析用ライブラリ調製キット

- 網羅的な遺伝子発現解析にお勧め
- 4.5 時間で total RNA から 3' mRNA-Seq ライブラリを調製可能
- マイクロアレイ解析や従来の RNA シークエンス解析と比較し低コスト
- 無料のデータ解析パイプライン「Bluebee®」をご用意
- Globin blockやマルチプレックス解析用インデックス、増幅バイアス確認用インデックス(UMIs)と組み合わせてライブラリ調製可能
- Unique Dual Indices(UDIs)とのセット品もご用意
実験内容
いわゆる”次世代”シーケンサーによるDNA配列解析が身近になり、RNAシーケンシング(RNA-Seq)はマイクロアレイに変わる網羅的な遺伝子発現解析の手法として様々な生物種を対象に行われるようになった。RNA-Seq は、試料から抽出した mRNA を逆転写して得られる cDNA の配列を超並列シーケンサーで解析することにより、発現遺伝子の特異的配列や頻度をデータとして直接取得する手法で、目的や解析プラットフォームに合わせて様々なアプリケーションが開発されている。
我々かずさDNA研究所の植物ゲノム・遺伝学研究室でも、植物の網羅的遺伝子発現解析を進めている。材料はトマト、イチゴ、キクなどの野菜類や花卉類で、先行して全ゲノム解析や遺伝子予測を進めてきたものであるが、生育ステージ別の組織特異的な遺伝子発現や形質にばらつきのある交雑分離集団の遺伝子発現を数十から一千検体の規模で解析するうえで、解析コストの削減が大きな課題となっていた。
そこで、コスモ・バイオ株式会社から発売されているウィーンの Lexogen社の QuantSeq 3’ mRNA-Seq Library Prep Kit を導入した。QuantSeq はオリゴ dT プライマーによる polyA 修飾された mRNA の逆転写と続くランダムプライマーによる二本鎖 cDNA 合成により、発現遺伝子の3’特異的配列の cDNA ライブラリを作成することができるキットである。解析プラットフォームは、イルミナ株式会社(以下 イルミナ社)の各種シーケンサーとサーモフィッシャーサイエンティフィック株式会社の Ion Torrent に対応しており、それぞれ別々のキットとなっている。イルミナ社シーケンサー対応キットの場合、幅広い total RNA インプット量に対応し、検体数が小規模であれば、実験開始から合計4時間半と短時間でライブラリの作成が可能である。QuantSeq の利点は、(1) 理論上1転写産物あたり1分子の断片のみ逆転写され mRNA のカウンティングに特化している点、(2) cDNA ライブラリ中に mRNA の3’側配列のみ含まれるため、シーケンス解析時にサンプルあたりのリード数を節約できる点にある。後者は特に多検体を解析する場合に有益で、これによりシーケンス解析にかかるコストを大幅に節約することができる。96サンプル対応キットの場合、付属する96種類の6塩基のバーコード配列により、フローセルの同一レーンで複数サンプルのマルチプレックス解析が可能である。さらにデュアルインデックスに対応した384サンプル対応キットも発売された。シーケンス解析プラットフォームのハイスループット化が加速するなか、今後の有力な選択肢の一つとなるだろう。
一方、3’特異的 cDNA ライブラリゆえの注意点もある。mRNA の全域を逆転写するライブラリ作成手法(例えば イルミナ社の TruSeq など)では、取得したリードを発現遺伝子の de novo アセンブリやゲノムへのマッピングを経て遺伝子予測を行うなどの目的に用いることができるが、QuantSeq の3’特異的cDNAライブラリには5’側配列が含まれないため、発現遺伝子の全長をカバーする配列取得はできない。このため、QuantSeq を導入する前に予めリファレンスゲノムが構築されている生物種であることを確認する必要がある。また、同じ理由でスプライシングバリアントの検出等の目的にも不向きであろう。リード取得後の発現量の算出の際には、遺伝子長で補正する必要がないため、カウントのみを行うプログラム(HTSeq等)を用いるなど、配列取得後も注意が必要である。
mRNA のカウンティングに特化した QuantSeq は、全ゲノムや遺伝子の情報が明らかになっている材料で、タイムコースや集団規模など多検体の遺伝子発現解析が必要な場合に最も有力な選択肢の一つであると考えられる。
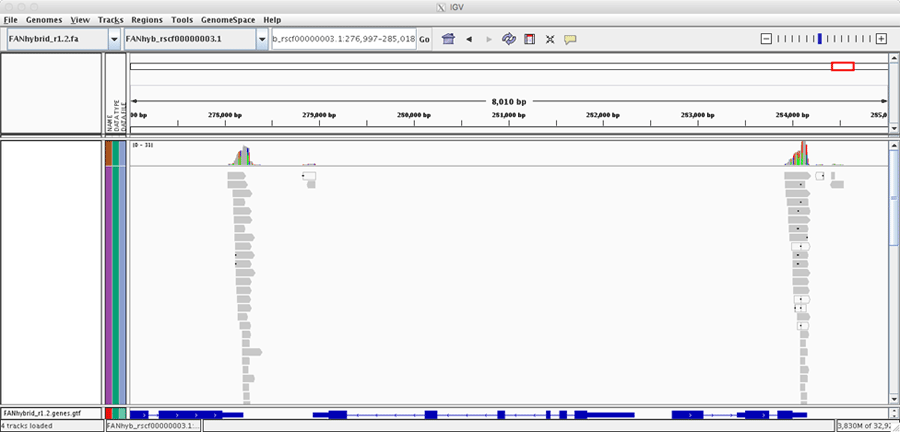
図1 QuantSeq 3’ mRNA-Seq Library Prep Kit で作成したイチゴの cDNA ライブラリの Illumina リード(灰色の横棒)をリファレンスゲノムにマッピングし、IGVで表示した。QuantSeq を用いると、遺伝子領域(青色の横棒)の3’特異的配列を取得することにより、リードの節約を行うことができる。























 このページを印刷する
このページを印刷する