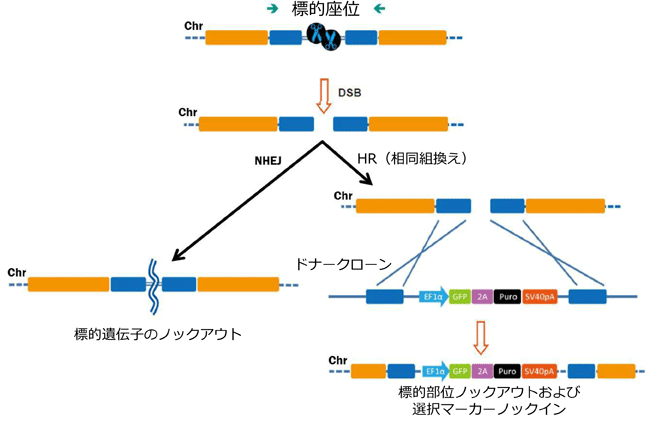
遺伝子ノックアウト
特定遺伝子のノックアウトを行う場合、ドナー(ベクター)は必須ではありません。実際、遺伝子改変動物では薬剤選択ができないことから、ノックアウトドナーはあまり有用ではありません。一方で、培養細胞における特定遺伝子のノックアウトの場合は、ドナー(ベクター)が有用となるケースが多々あります。これらのドナーは非常に単純な構造で、 sgRNA(gRNA、ガイドRNA)の標的部位に隣接する相同性の左腕と右腕(ホモロジーアーム)をもたせ、標的部位の一部が削除されて、薬剤選択マーカーと置換される構造となっています(図.2)。ドナーを利用したノックアウトにより、エキソン全体や遺伝子全体を削除する方法もあります。ノックアウトを目的とする際、多くの場合で2つの対立遺伝子を修正する必要があるため、同一ドナー配列をもち、かつ、異なる薬剤選択マーカーをもった2種のドナープラスミドを構築することで、クローンスクリーニングの際に二重薬剤耐性選択を行うこともできます。
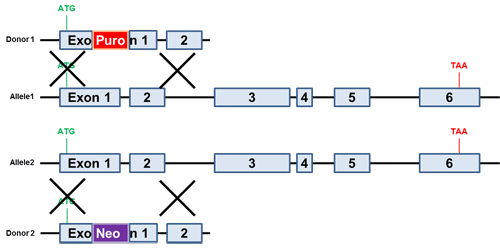
図.2 ノックアウトドナー方略
異なる選択マーカーをもつ2つのドナーを構築した。二重薬剤選択により2つの対立遺伝子ノックアウトクローン選択が亢進する。
条件的ノックアウト
希望する時間、特定組織、または特異的な生理的条件下においてのみ遺伝子ノックアウトを実施したい場合、条件的ノックアウトが必要となります。一般的な条件的ノックアウト手法として、エキソンに隣接する各イントロン内にloxPを配置する方法があります("floxed"アリル)。loxPが挿入されたアリルはCreリコンビナーゼが発現するまで活性型です。CreによりloxP部位間の組換えが生じエキソンが欠失します(図.3)。
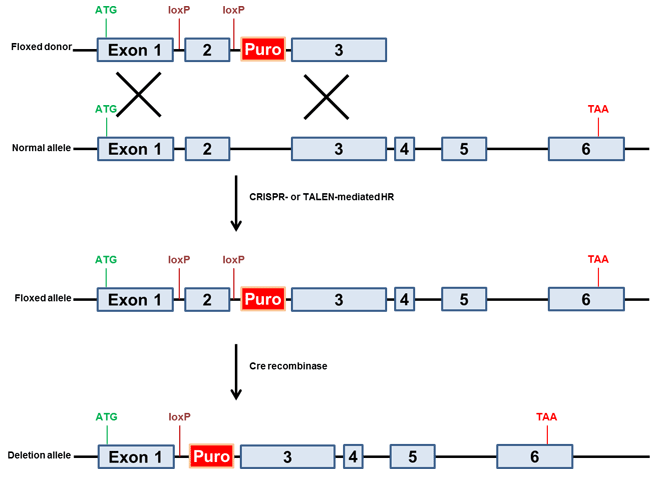
図.3 条件的ノックアウトドナー
必須エキソンに隣接したloxP部位をもつ、loxPが挿入されたアレルのドナー(最上図)をCRISPR Casシステム(gRNA)またはTALENシステムと同時にトランスフェクションした。薬剤選択によりloxPが挿入されたアレルをもつクローン抽出が可能。その後のCreリコンビナーゼ処理により必須エキソン欠失が生ずる。
塩基置換
ゲノム編集において最も有用なアプリケーションのひとつに単一塩基対置換導入があります。対象遺伝子への変異導入や疾患変異の修復など、何れにおいても有用です。染色体への塩基対置換導入には一般に2種類のタイプのドナーが利用されています。まず、選択マーカーをもつドナークローンの利用があげられますが、この種のドナーは培養細胞において使用され、上述の条件的ノックアウトで使用されるものと同様、薬剤耐性遺伝子は近隣のイントロンに配置されます(図.4)。次に、希望部位の隣接領域とおよそ40〜80塩基の相同配列をもつ1本鎖オリゴ(ssODN)を利用する方法があげられます。ssODNドナーの主な利点として、正確な相同組換えにより内在性配列の「傷跡のない」塩基対変化が生じ、他の配列の変更が生じないことがあります。本アプローチの成功例としてWu, et. al. (2013)があり、マウスにおいてssODNとCRISPR-Casを使用して白内障の原因となる変異の矯正を行っています。しかし、培養細胞実験ではssODNドナーを用いると正確に修正されたクローンを同定するためのセレクションを行うことができず、特にトランスフェクション効率の低い細胞では選択マーカーを持つドナークローンがより有用です。
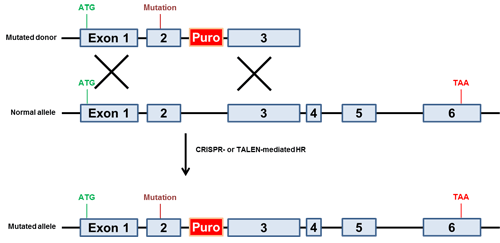
図.4 変異誘発性ドナー方略例
単一塩基対置換をもつドナー(最上図)とsgRNAs(CRISPR-Cas9)またはTALENsを同時にトランスフェクション。薬剤選択により変異したアリルを単離。
インフレームの融合標識法
次に一般的なゲノム編集ドナーのアプリケーションとしてタンパク質のインフレームでの融合標識法があげられます。例えば、対象とする内在性タンパク質のC末端にGFPタグを付加する場合を想定すると、CRISPR-CasあるいはTALENドナーを利用して、標的遺伝子の元来の染色体座位の3'末端にGFPをノックインする方法が考えられます(図.5)。条件的ノックアウトや変異誘発性用のドナーと同様、培養細胞において正しく修正されたクローンの選択を目的として、近接のイントロン内に選択マーカーを配置することが多々あります。
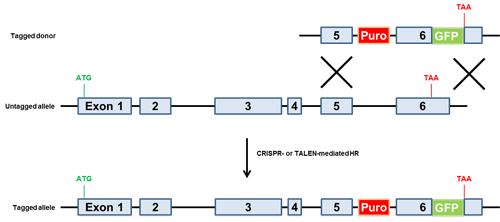
図.5 ドナー介在型融合標識法
ドナーは終止コドンの上流に3'末端タグをもつ。クローンセレクション用の薬剤選択マーカーはイントロン内に配置。
本稿で最後に議論するドナーは、ヒトAAVS1またはマウスRosa26といった「セーフ・ハーバー」部位に導入遺伝子をノックインする方法です。これらの部位は細胞や動物に対して有害な効果を示すことなく導入遺伝子挿入が可能であることが数々の研究により実証されています。さらに、セーフ・ハーバー部位に組込みを行うことで、ランダムな導入遺伝子組込みと異なり一貫性のある定常的な導入遺伝子発現が保証されます。セーフ・ハーバー部位への組込み用途として、ヒト以外の遺伝子をヒト細胞へ組込む、あるいは、染色体遺伝子欠損表現型を安定組込みORFクローンによりレスキューするなどといった、様々な可能性が考えられます。GeneCopoeia(GCP)社では、検証済みCRISPR-Cas9試薬をひとつにしたキットやお客様の対象導入遺伝子をAAVS1またはRosa26相同腕(ホモロジーアーム)間にご自身で挿入できるベクターなど、先端的なセーフ・ハーバー部位組込み用の製品をご提供しています。さらに、GeneCopoeia(GCP)社ではヒトおよびマウスのORFクローンをゲノムワイドに保有しており、36,000種以上のセーフ・ハーバーノックインORFクローンをご用意しています(図.6)。これらのクローンはヒトまたはマウスの配列検証済みORFsがセーフ・ハーバー相同腕(ホモロジーアーム)間に組込まれています。
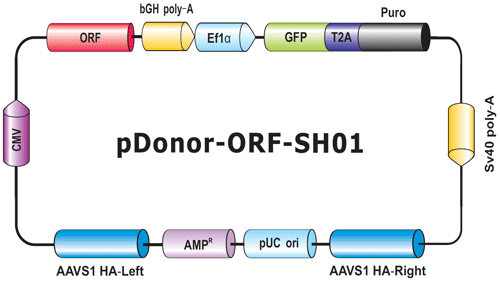
図.6 Genome-CRISPTM ヒトAAVS1セーフ・ハーバーノックインクローン骨格
ORFはAAVS1の相同性左腕と右腕の間に選択カセットとともに配置されている。
本稿では、ドナークローンを使用したゲノム編集アプリケーションについてその効果的な方略をいくつかご紹介致しました。GeneCopoeia(GCP)社では、ご自身で構築できる数種類のドナークローニングベクターなどゲノム編集用ドナーアプリケーション関連商品を種々取り揃えています。ドナーベクターをデザインする際には本稿で焦点をあてた事項以外にもさまざまな複雑な点を考慮する必要があります。GeneCopoeia(GCP)社では、ゲノム編集熟練チームによりデザインや構築を行い、配列検証済みクローンをお届けするカスタムノックインドナー構築サービスもご提供しております。また、お客様の標的遺伝子をCRISPR-Cas技術により改変した安定発現細胞株や遺伝子改変マウスの構築も承っています。