CRISPR や TALEN などゲノム編集ツールを用いた実験では、標的の細胞や動物が適切に編集されているか確認するためのスクリーニング実験や検証実験が必要になることがしばしばあります。一般的によく使用されている検証ツールとして “mismatch cleavage assay” があります。GeneCopoeia社では “mismatch cleavage assay” を利用した IndelCheck™ 変異検出キットを提供しており、簡単にゲノム編集による変異導入の確認を行っていただけます。
技術情報
なぜ CRISPR や TALEN を用いたゲノム編集には機能検証が必要なのか
まず、CRISPR や TALEN を用いてゲノム編集を行う際には、事前に sgRNA の効率を検証することをお勧めします。CRISPR や TALEN によるゲノム編集は高効率と考えられていますが、ターゲット部位の性質によって編集効率は大きく変化します。したがって、変異導入後、下流の実験に使用する前に、どの細胞クローンが最も適切かを評価することが重要です。
IndelCheck™ システムは二本鎖切断を介した非相同末端結合(NHEJ)により生じた indel(変異; 挿入または欠失)を検出するためのセルベースアッセイシステムです。(ワークフローは図1を参照)
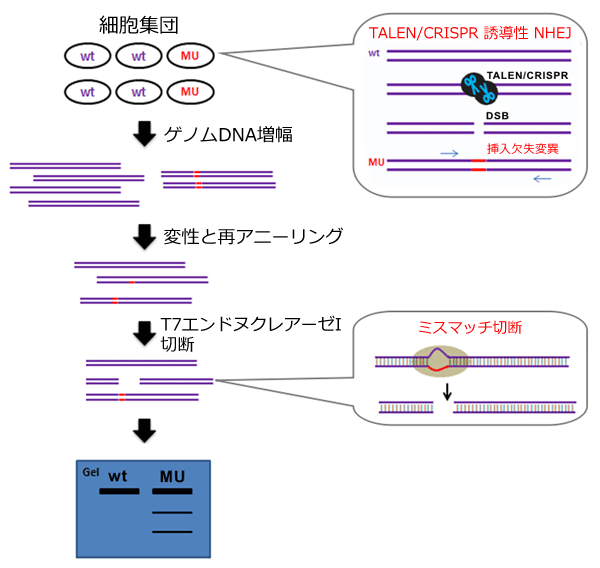
CRISPR または TALEN にてゲノム編集を行った細胞について、標的部位を挟んだ位置に設計したプライマーを用いてPCRを行う。次にPCR産物を変性後、再アニーリングすることで、ミスマッチを含む二本鎖集団を形成させる(ホモ二本鎖、ヘテロ二本鎖)。このミスマッチは T7 Endonuclease I の基質となり切断される。CRISPR または TALEN が細胞内で機能した場合、アガロースゲル上で切断産物を確認することができる。
IndelCheck™ システムの使用例
哺乳類細胞でゲノム編集を行う場合(in vitro)、同じ細胞株で検証実験を行うことをお勧めします。また、マウスやラットなど、in vivo でゲノム編集を行う場合は、マウスでは Neuro2A 細胞や NIH3T3 細胞、ラットでは PC-12 細胞などのモデル細胞株で検証実験を行うことをお勧めします。図1に示したように、IndelCheck™ システムで検証を行う場合には、CRISPR Cas9/sgRNA または TALEN ベクターのみを一過性導入(プラスミドトランスフェクション)します。実際に細胞ゲノムを編集する際に相同組換え用のドナーベクターが必要だとしても、この検証実験にはドナーベクターは不要です。IndelCheck™ システムでは、ドナーベクターの有無に関わらず、NHEJ により導入された変異を検出します。
実験手順としては、まず、トランスフェクションして1-2日後、細胞プールを回収して、ターゲット部位近傍に設計したプライマーを用いてPCRを行います。

図2. IndelCheck™ システム用PCRプライマーの設計方法
黄色のハイライトで示す配列をPCRプライマー結合部位とした。(PCR産物の推奨サイズ: 500-1,000 bp)PCRプライマーを設計する際、二本鎖切断位置はプライマー設計領域の中間点から少なくとも 100 bp 内になるよう設計するよう注意する。ターゲット部位は3か所とし(緑色、ピンク色、水色)、うまくミスマッチが検出される場合、下記のサイズのバンドが検出される。
human HDAC6遺伝子のPCR産物:989 bp(NCBI GeneID: 9636)
341 bp + 648 bp(緑色)
555 bp + 434 bp(ピンク色)
616 bp + 373 bp(水色)
NHEJ を介した変異導入は確率的で、挿入、欠失どちらも起こり得ます。また、挿入欠失配列の長さは実験毎に異なります。NHEJ を介した変異導入が成功した場合、細胞プールにはターゲット部位に異なる変異をもつ様々な集団が含まれます。また、細胞プールには編集されていない野生型の集団も含まれますので、様々な細胞集団が混在している状態は非常に複雑だと考えられます。
PCRでの増幅後、産物を 95℃ で変性し、常温になるまでゆっくりと冷ますことで再アニールします。冷却により、マッチ配列、ミスマッチ配列についてそれぞれアニーリングが起こり、ホモ二本鎖およびヘテロ二本鎖が生じます(図1)。
その後、再アニーリングしたDNAを T7 Endonuclease I で処理します。T7 Endonuclease I は二本鎖のミスマッチを認識し、ヘテロ二本鎖のミスマッチ部分を切断しますが、ホモ二本鎖は切断されません。T7 Endonuclease I で消化した断片はアガロースゲルで電気泳動します。一般的に、NHEJ を介した変異が導入されると3つのバンドが見られます。1つ目は切断されなかったDNA(ホモ二本鎖)、他2本のバンドは切断された断片(ヘテロ二本鎖)です(図3)。
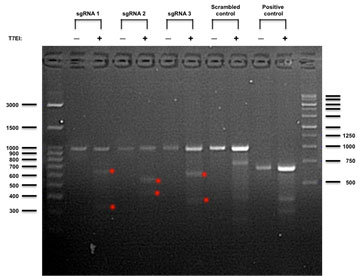
HEK293 細胞に CRISPR/sgRNA クローンをトランスフェクションし、IndelCheck™ システムにて変異検出を行った。アニーリングしたDNA断片を T7 Endonuclease I で消化したサンプル(+)、消化していないサンプル(-)について 2% アガロースゲルで分離した。
*印で示したミスマッチ切断断片が検出されたサンプルについては CRISPR が細胞内で機能したと考えられる。通常、スクランブル sgRNA コントロールでは二本鎖切断は起こらないが、本実験では、スクランブル sgRNA コントロール群で T7 Endonuclease I 消化後にミスマッチ切断断片が検出された。これは、自然に存在する異なる染色体上の遺伝子多型によるものと考えられる。スクランブルコントロール群の切断断片の大きさは sgRNA 群(1, 2, 3)の切断断片とは異なっているため、疑陽性ではないと考えられる。
IndelCheck™ システムを用いたスクリーニング実験
IndelCheck™ システムは、NHEJ を介したノックアウトのスクリーニングにも使用可能です。スクリーニングの手順は上述の検証方法と同様ですが、細胞サンプルは異なるアレルを含む細胞集団のミックスではなく単一クローンである必要があります。単一クローンが同一の変異をホモでもっている場合、IndelCheck™ システムではヘテロ二本鎖DNAは生じず、電気泳動データからは、そのクローンが変異導入細胞であるとは判断されません。このような状況を避けるため、T7 Endonuclease I 処理を行う前に、野生型のPCR産物をスパイクインすることをお勧めします。
このステップによりヘテロ二本鎖DNAを検出し、変異の有無を見分けることが可能です。変異が導入されたクローンが同定されたら、変異およびアレルを調べるため、TAサブクローニングやシークエンス解析を行います。
商品は「研究用試薬」です。人や動物の医療用・臨床診断用・食品用としては
使用しないように、十分ご注意ください。
























