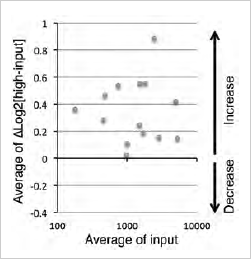
専用デコンボリューションソフトウェアを使って、投入群と選抜群それぞれについての次世代シーケンサーの配列情報から各shRNAコンストラクトを同定し、それぞれのリード数の情報を得た。その結果、得られた各2千万リードのうち、最大/最小リード数は各モジュールでそれぞれ12,314/14及び2,806/8であった(表2)。このことから、ライブラリーウイルス感染後に各コンストラクト間の存在比率の差は拡大したものの、全てのshRNAコンストラクトについて評価できていることがわかる。各データの正規化を行った後、投入群と選抜群との比率をLog2変換(ΔLog2)し、3回の実験について平均値と標準偏差を算出した(表2)。平均値によってshRNAコンストラクトのID及び標的遺伝子を並べ直してランキング化した。標準偏差のおよそ95%が0.2以下(平均はそれぞれ0.0913及び0.0880)だったので、ざっくりと分けるとすれば、平均値が0.4以上のshRNAコンストラクトは投入群と比較して高発現の選抜群で有意に濃縮されていることが期待される(図2及び表2)。この範囲には全コンストラクトのおよそ2.5%が相当していた。また、それらの10〜20%が同一の遺伝子を標的としたものだった。
| 最小リード数 (/ 2x107) | 最大リード数 (/ 2x107) | 平均標準偏差 (Δlog2) | ΔLog2 > 0.4 (1.32倍以上濃縮) | 1.5倍以上濃縮 | |
|---|---|---|---|---|---|
| Module 1 | 14 | 12,314 | 0.0913 | 632(498 genes) | 158(124 genes) |
| Module 2 | 8 | 2,806 | 0.0880 | 692(604 genes) | 170(157 genes) |
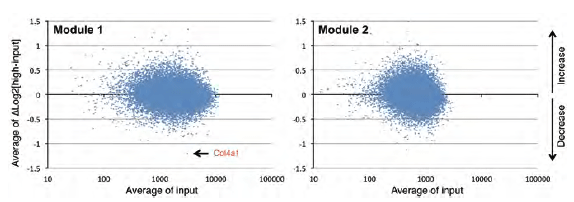
図2 全shRNAコンストラクトの濃縮率
全shRNAコンストラクトの濃縮率の分布。X軸は投入群において検出された各shRNAコンストラクトの正規化リード数の平均値(N=3)で、Y軸は選抜群と投入群の平均値の比をLog2変換したものである(ΔLog2)。Y軸値のΔLog2>0 は投入群よりも選抜群でリードが多く検出され、濃縮傾向にあることを示す(Increase)。ΔLog2>0.4を示すshRNAコンストラクトは有意に濃縮されている可能性がある。