クロマチン免疫沈降法(ChIP)によりショウジョウバエの組織からクロマチンを回収し、次世代シークエンシング用のライブラリ調製を行うためのキットです。
独自のバッファーとビーズ不要のChIP技術により、ヒストン修飾や転写因子を迅速かつ簡単に高感度で同定できます。
キットには、DNAの抽出およびライブラリ構築用のDNAを得るために必要なほぼ全ての試薬が含まれています。
ChIPからライブラリ調製までの試薬セット
Chromatrap® ショウジョウバエUniqSeqキット
- カテゴリから探す > 抗体アッセイ > クロマチン免疫沈降(ChIP)
- カテゴリから探す > シグナル伝達 > クロマチン > クロマチン免疫沈降(ChIP)アッセイ
- カテゴリから探す > 分子生物 > DNAシーケンシング > 次世代シーケンシングキット
背景
ChIPはエピジェネティック研究において非常に重要であることが知られています。本技術は特定のタンパク質同士の関連性や特定のゲノム領域内で修飾されたイソ型の研究に使用され、タンパク質-DNAの相互作用の特性の解析に使用されます。ChIPアッセイでは、特異的抗体を用いてタンパク質-DNA複合体(クロマチン)を選択的に免疫沈降し、得られた断片をDNAとタンパク質成分に分離します。標的DNA断片の同定には、PCR、リアルタイムPCR(qPCR)、マイクロアレイ上でのハイブリダイゼーション、またはダイレクトシーケンスが用いられます(図1)。
遺伝子制御の研究においてタンパク質-DNA間の相互作用をゲノムワイドでマッピングすることは非常に重要です。
遺伝子制御ネットワークの解析にはエピジェネティクマークや転写因子結合を詳細にマップすることが必要です。これらのタンパク質-DNA相互作用を検討するために、ChIPとその後に行うChIP-seqが広く使用されています。ChIPで濃縮されたDNAを次世代シーケンシング(NGS)することで、in vivoにおいての制御機構や標的DNA部位の同定を行うことができます。配列が決定したこれらのDNA断片を全ゲノム配列データベースに対してマッピングすることで、転写因子やエピジェネティック修飾における相互作用を迅速かつ効率よく分析することができます。
次世代シーケンシング(NGS)により産生された短いリードはChIP-seqに適しており、配列モチーフの同定が改善されるだけでなくタンパク質結合部位の詳細なマッピングが可能となります。ChIP-seqでは、クロマチンやヒストン変異体の翻訳後修飾やヌクレオソームの位置をプロファイリングにより空間的にとらえることができ、タンパク質-DNA相互作用のゲノムスケールでの解析において最先端技術であるといえます。
本キットには、標的とする濃縮DNAをIllumina® platforms上で配列決定するため、高品質のNGSシーケンシングライブラリへと変換するために必要なほぼ全ての試薬が含まれています。これらのライブラリは単一チューブ内で500pg〜1μgのDNAを調製でき、最適化されたプロトコールと試薬を用いて調製するため、酵素反応ごとに精製を行う必要がありません。
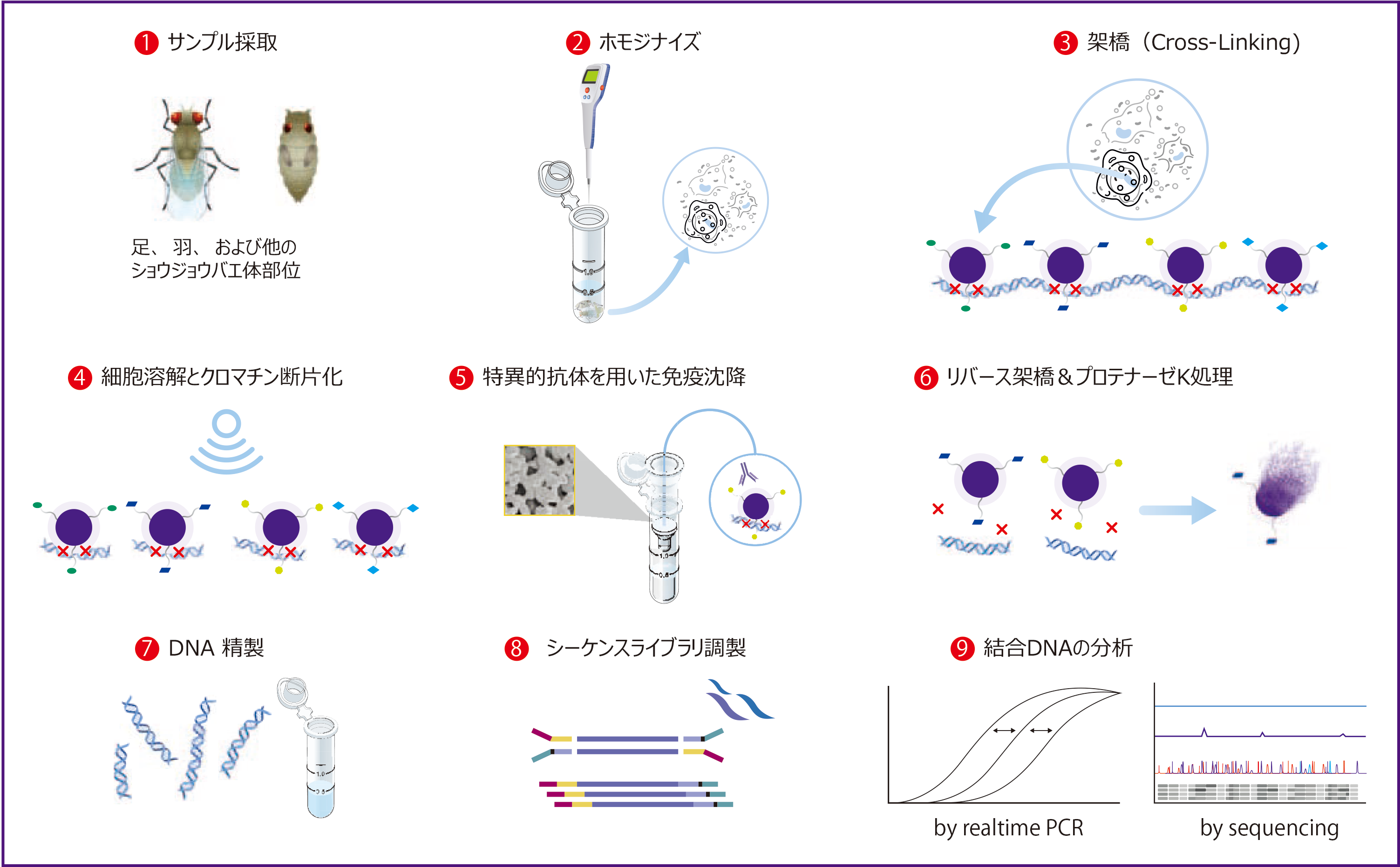
図1. Chromatrap® Drosophila ChiP-seqプロセスの概略
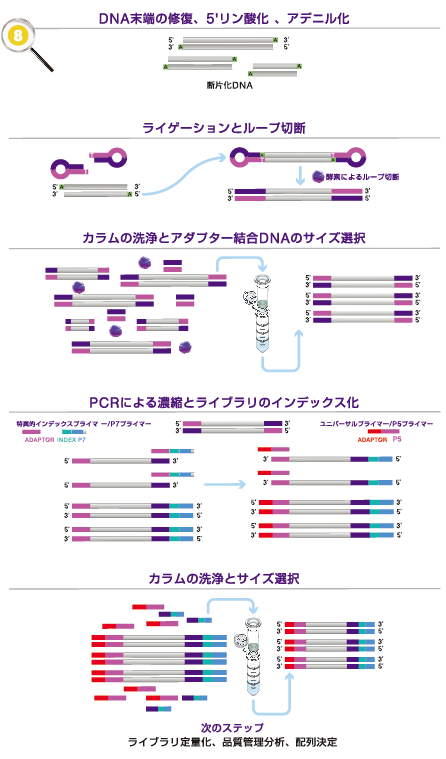
図2. ライブラリ調製の概要
使用目的
Chromatrap®独自の特許取得済み*技術を利用し、迅速、簡単、かつより効率的、高感度にChIPアッセイを行うことができます。磁気やアガロースビーズではなく、多孔性プラスチックのフィルターディスク:Vyon®を使用し、フィルター全体に均一にプロテインAまたはプロテインG付加することで標的クロマチン-抗体複合体の回収率が向上します。
本キットは、ショウジョウバエ組織のChIP-seq解析用にカラムやNGSライブラリ調製酵素及び試薬を組み合わせたキットです。
*UK Patent No. GB2482209, US Patent No. 9523681, Chinese Patent No. ZL 2011 8 0067254.X, Japan Patent No. JP 6088434, Australian Patent No. AU 2011340263.
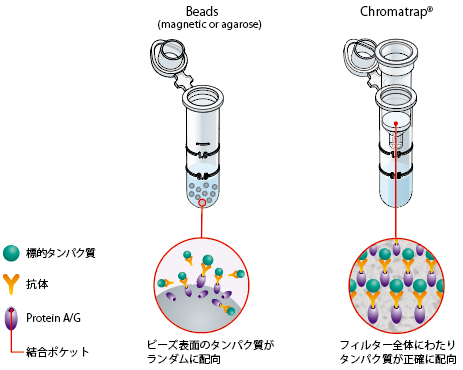
図3. Chromatrap® ChIP技術
特長
- ショウジョウバエの組織からクロマチンを高感度に回収
- 少量のサンプル(5匹分)からChIP-seqが可能
- 不活性フィルターにより低バックグラウンド
- ビーズ不使用
- ブロッキングやオーバーナイトインキュベーションが不要
- 1回のIPでサンプルをプールすることなくNGS品質のDNAを回収
- 1チューブ反応、ライブラリ反応ステップごとの精製が不要
- 5時間以内に高品質NGSライブラリが調製可能
- 10回のクロマチン調製と 24回の ChIP アッセイが可能
構成内容
本キットには、24ライブラリ調製に必要な試薬が含まれています。MiSeq、HiSeq、およびNextSeqといったIlluminaシークエンシング機器に対応しています。Ion Torrent、SOLiD® system、およびRoche 454については適切なライブラリキットをご用意ください。
- キットコンポーネント
- Chromatrap® Spin Columns(24個)
- Column Conditioning Buffer
- Wash Buffer 1
- Wash Buffer 2
- Wash Buffer 3
- 1.3 M Glycine
- Hypotonic Buffer
- Lysis Buffer
- ChIP-Seq Elution Buffer
- 5 M NaCl
- 1 M NaHCO3
- Protease Inhibitor Cocktail (PIC)
- Proteinase K Stop Solution
- Proteinase K
- 1.5 ml Collection Tube(50個)
- Chromatrap® DNA Purification Columns(24個)
- DNA Binding Buffer
- DNA Wash Buffer
- DNA Elution Buffer
- End Prep and Tail Enzyme
- End Prep and Tail Buffer
- Ligation Mix
- Adapter Ligase
- Adapter for Illumina®
- Loop Restriction Enzyme
- Library Enrich and Index Mix
- 15 primer for Illumina®
- Index primer 1〜12 for Illumina®
- 追加で必要な試薬・消耗品
- PBS
- 37% ホルムアルデヒド、分子生物学グレード
- 水(ヌクレアーゼフリー)
- 100bp ラダー
- 遠心チューブ(0.5mLと1.5mL)
- PCR プレート
- DNA LoBindチューブ(オプション)
- ピペットとチップ(フィルターチップを推奨)
- ChIP検証済み抗体
- 対象遺伝子に対するqPCRプライマーペア
- 3M 酢酸ナトリウム、pH 5.2
- 追加で必要な機器
- 組織ホモジナイザー
- 微量遠心機(4℃)
- アガロースゲル電気泳動装置
- DNA定量用吸光光度計/蛍光光度計
- 超音波処理機
- ローテーター
- Illumina® platformsでのシーケンシングに追加で必要な試薬・機器
- PCR装置
- Qubit® 2.0蛍光光度計・dsDNA高感度キット(または同等の蛍光定量手法)
- Agilent Technologies 2100 Bioanalyzer・高感度DNAキット
- ライブラリ定量キット
- サイズ選択可能なDNA精製キット(Chromatrap® size selection kit (品番:500262) を推奨)
プロトコール
内容
| ステップ | プロセス | 必要な時間 | 日数 |
|---|---|---|---|
| 1 | 細胞固定・回収 | 0.5時間 | 1 |
| 2 | 細胞溶解・クロマチン断片化 | 0.5時間 | 1 |
| 3 | 免疫沈降 | 1時間 | 1 |
| 4 | リバースクロスリンク・DNA精製 | 3.5時間 | 1 |
| 5 | 定量PCR解析 | 1時間 | 1 |
| 6 | *Chromatrap® NGSライブラリ調整 | 5時間 | 2 |
| 7 | ライブラリ数量化と定量分析 | 2時間 | 2 |
注意事項
■抗体の品質について
ChIP-seq実験は抗体の品質に左右されます。特異的な抗体を使用すると、データ解析の際に結合現象を簡単に検出することができます(バックグラウンドに比べて標的化合物の相対的な濃縮が増大するため)。抗体は、ChIP-seqグレード抗体をご使用ください。ロット間の変動がございますので、メーカーではChIPに使用する前にバリデーションすることを推奨しています。
■サンプル量について
ChIP-seqライブラリ調製に十分な濃縮DNAを得るための必要なサンプル量は、クロマチンと抗体の品質、標的タンパク質や遺伝子量、使用するライブラリ調製キットの感度や効率に依存します。本キットは、Chomatrap® Drosophila UniqSeq kit (品番:500276、500277) を用いたライブラリ調製に最適化されています。これらのキットを使用することで、最少5匹の昆虫より高品質ChIP-seqデータを得ることができます。
Chomatrap® Drosophila UniqSeq kit (品番:500276、500277)を用いたライブラリ調製には最低500 pgのDNAが必要です。他のライブラリ調製キットをご使用になる場合は、最適化してご使用ください。ただし、配列の重複が問題となる場合は、重複レベルを最小限に抑えられるようライブラリ増幅時の初期サンプル量を増やすことをお勧めします。重複問題が解決しない場合には、濃縮ステップで使用したPCRサイクル数を減らしてください。
■断片化について
100-500bpの断片を得られるよう最適化することが重要です。配列決定する場合には、200-300bpの範囲内でお願いします。大きな断片はバックグラウンドが高く、シークエンシングの際にクラスターを形成する可能性があります。
■コントロール実験について
シークエンシング解析で同定されたピークは、コントロールサンプルの同一領域と比較する必要があります。例えば、反復配列のランダムな領域はその領域のコピー数により濃縮されたように見え、偽陽性を生じます。一般に使用されるコントロールは、インプットDNA(免疫沈降していないDNA)、mock IP(同様に処理したDNAではあるが、IPの際に抗体を使用しなかったもの)、および非特異的IP(IgGなどDNA結合に関与しないタンパク質を標的とする抗体を使用したIP)の3種類があります。どのコントロールの使用が最も適切であるかはコンセンサスが得られておりませんが、インプットDNAやIgGコントロールがDNAの断片化や増幅に関連するバイアスの要因となるため、一般的に使用されています。
■反応条件について
ChIP-seqでシークエンシング解析を行うには、ショウジョウバエの組織量から断片クラスター数に至るまでさまざまな反応条件を最適化することが必要ですが、本キットを使用することで、必要な最適化ステップ数を低減でき、またIllumina® sequencing platformとともに利用できるよう調整されています。Chromatrap® Drosophila UniqSeq Kit (品番: 500276、500277) には、MiSeq、HiSeq、NextSeqといったIlluminaシークエンシング機器のみに対応したアダプターや指標が添付されています。Ion Torrent、SOLiD® システム、および Roche 454といった他のシークエンシングプラットフォームをご利用の場合は、ご自身で適切なライブラリキットをご用意ください。
Chromatrap® ショウジョウバエUniqSeqキット
| 品名 | メーカー | 品番 | 包装 | 希望販売価格 |
|---|---|---|---|---|
Chromatrap UniqSeq ProA, Drosophila |
PVS | 500276 | 1 KIT [24 assay] |
お問い合わせ |
Chromatrap UniqSeq ProG, Drosophila |
PVS | 500277 | 1 KIT [24 assay] |
お問い合わせ |
【関連商品】
| 品名 | メーカー | 品番 | 包装 | 希望販売価格 |
|---|---|---|---|---|
ChIP Validated H3K4me3 Antibody Positive Primer Set, Drosophila (Rabbit)  |
PVS | 900036 | 1 SET [50ul] |
販売終了 |
Positive primer set, Drosophila |
PVS | 900037 | 1 SET [250ul] |
販売終了 |
商品は「研究用試薬」です。人や動物の医療用・臨床診断用・食品用としては使用しないように、十分ご注意ください。
※ 表示価格について
- 「Chromatrap® ショウジョウバエUniqSeqキット」は、下記のカテゴリーに属しています。
-
- カテゴリから探す > 抗体アッセイ > クロマチン免疫沈降(ChIP)
- カテゴリから探す > シグナル伝達 > クロマチン > クロマチン免疫沈降(ChIP)アッセイ
- カテゴリから探す > 分子生物 > DNAシーケンシング > 次世代シーケンシングキット























 このページを印刷する
このページを印刷する












