遺伝子ノックアウト実験用のグルタミン酸作動性ニューロン、ミクログリア
ioCells™は、Bit Bio社独自の「opti-ox」技術を使用して作製された、ヒトiPS細胞由来の神経細胞、グリア細胞、骨格筋細胞です。ioCells™シリーズに、遺伝子ノックアウト実験用のグルタミン酸作動性ニューロン、ミクログリアが加わりました。最適化されたプロトコールで、ioCells™での遺伝子ノックアウト実験を行っていただけます。
遺伝子ノックアウト実験用のグルタミン酸作動性ニューロン、ミクログリア
ioCells™は、Bit Bio社独自の「opti-ox」技術を使用して作製された、ヒトiPS細胞由来の神経細胞、グリア細胞、骨格筋細胞です。ioCells™シリーズに、遺伝子ノックアウト実験用のグルタミン酸作動性ニューロン、ミクログリアが加わりました。最適化されたプロトコールで、ioCells™での遺伝子ノックアウト実験を行っていただけます。
CRISPR-Ready ioGlutamatergic Neurons は凍結細胞として提供され、推奨培地を用いて培養することで急速に成熟します。細胞を播種後、4日間の安定化、ニューロンを成熟させるメンテナンス培養を行います。gRNAは、day 1 からday 3の間(安定化の期間)で導入し、数日のうちに実験に使用可能です。
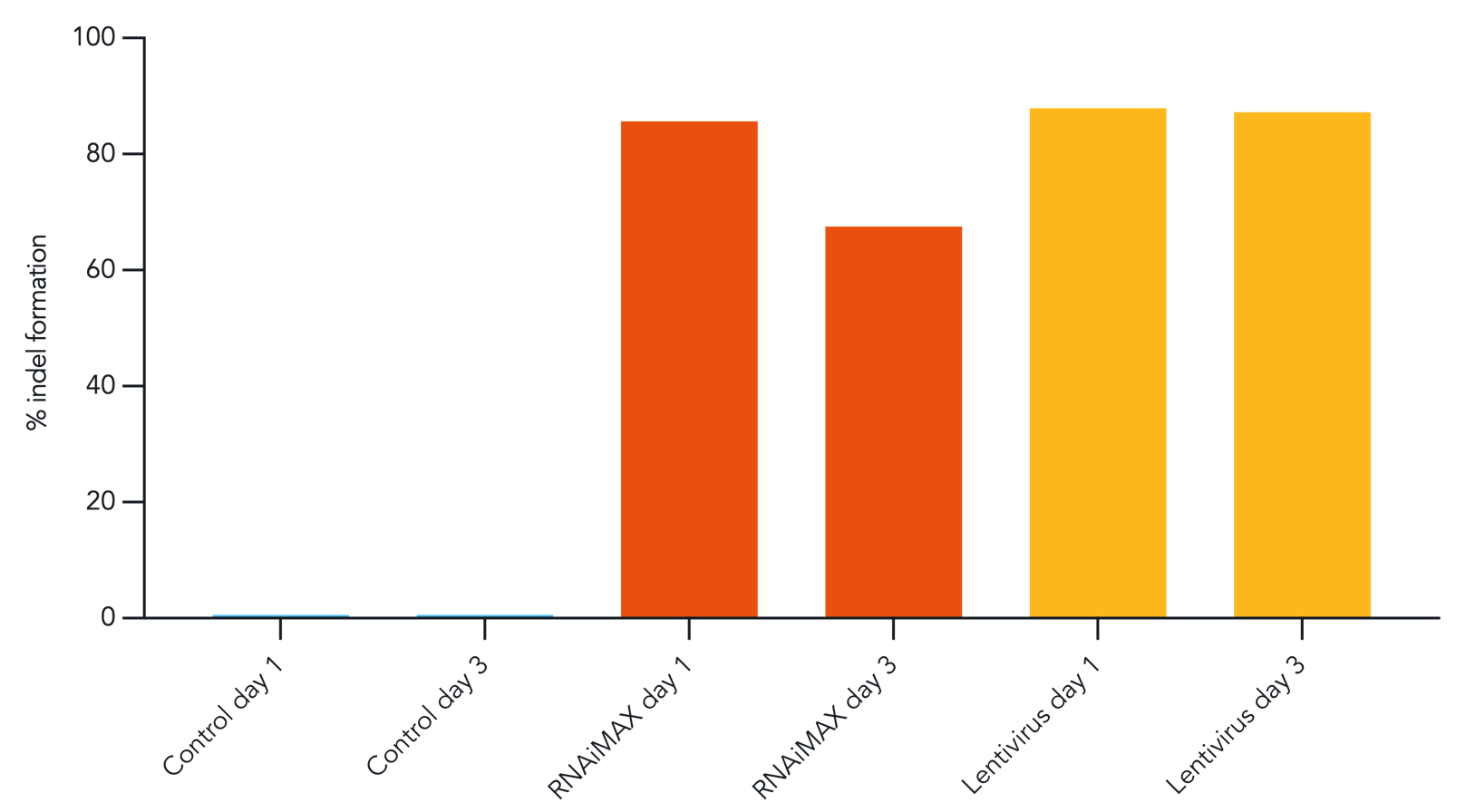
図 アンプリコンシーケンス解析によるノックアウト効率の評価
CRISPR-Ready ioGlutamatergic Neuronsに、レンチウイルスまたは脂質ベースのトランスフェクション試薬(Lipofectamine RNAiMAX)を用いてSOX11遺伝子を標的とするgRNAを導入した。gRNAは、細胞播種後1日目もしくは3日目に導入した。gRNA導入から3日後、DNAを抽出し、アンプリコンシーケンス解析を行った。どちらの導入方法でも、十分なノックアウト効率を確認した。遺伝子を標的としないgRNAをコントロールとして使用した。
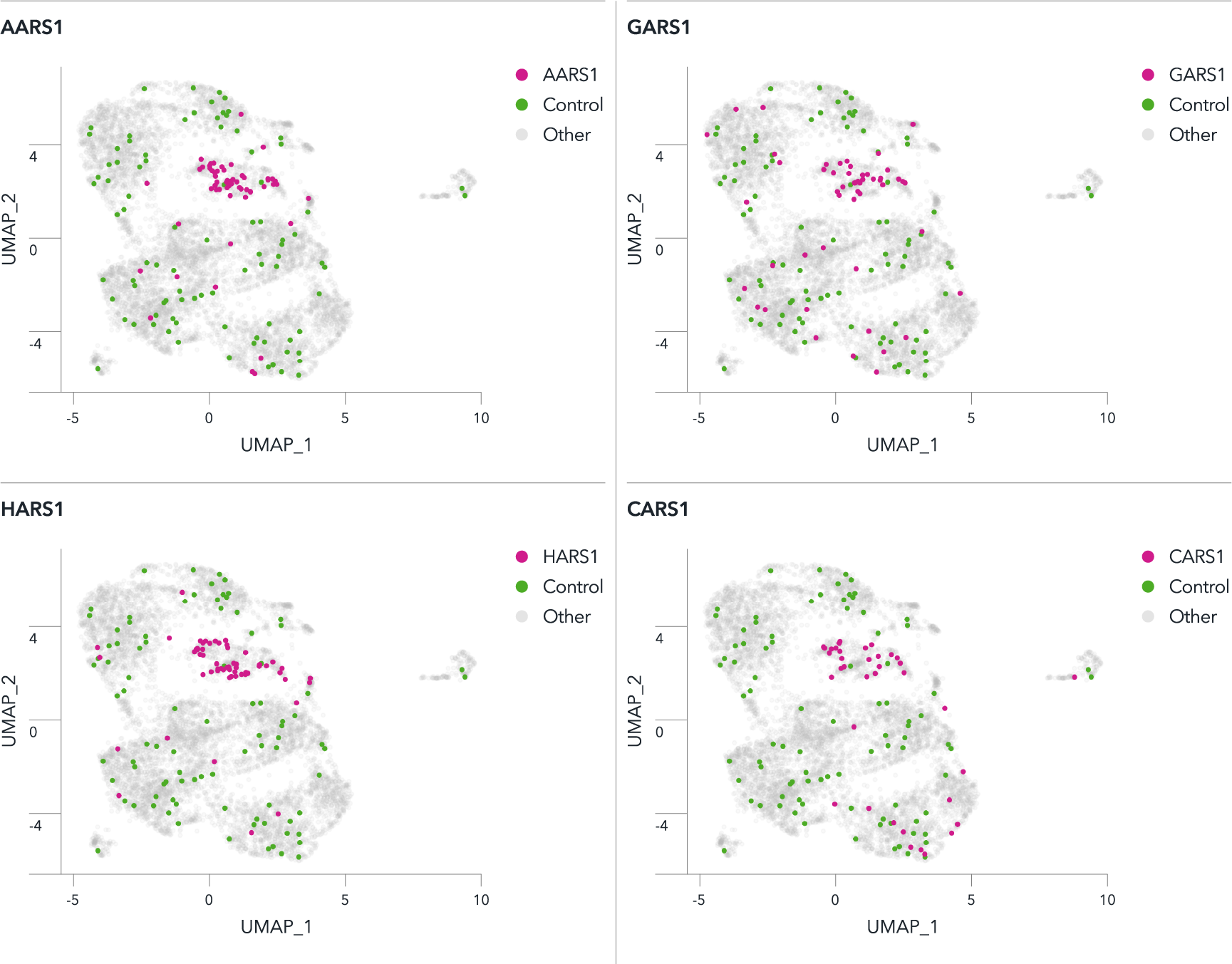
図 プール型CRISPRノックアウトスクリーニングによる神経変性疾患関連遺伝子の解析
プール型CRISPRノックアウトスクリーニング用に、神経変性疾患に関連する100種類の既知遺伝子を選択した。CRISPR-Ready ioGlutamatergic Neuronsを播種後3日目に、レンチウイルスによりgRNAを導入し、12日目にシングルセル遺伝子発現解析を行った。その後、シングルセルを、UMAPおよびshared nearest neighbor法を用いてクラスタリングした。AARS1, HARS1, CARS1, GARS1遺伝子等のaminoacyl-tRNA synthetase (aaRSs) ノックアウトのクラスターが検出された。対照的に、遺伝子を標的としないコントロールgRNAは広く分布していた。パスウェイ解析では、aaRSsを標的とするgRNAは小胞体ストレス応答 (unfolded protein response; UPR) を活性化することを示した。
| 品名 | メーカー | 品番 | 包装 | 希望販売価格 |
|---|---|---|---|---|
CRISPRko-Ready ioGlutamatergic Neurons for commercial users |
BIT | IO1090S | 1 SET [3 vials, >1 x 10^6 viable cells/vial] |
お問い合わせ |
CRISPR-Ready ioMicroglia (Male) は凍結細胞として提供され、推奨培地を用いて培養することで急速に成熟します。細胞を播種後、24時間の安定化、9日間の成熟、メンテナンス培養を行います。gRNAは、day 1 からday 18の間で導入し、導入から5日のうちで実験に使用可能です。タンパク質レベルでノックアウトを検出できるか否かは、標的とそのタンパク質の半減期の両方に影響されるため、検出のタイムポイントは、標的タンパク質に合わせて調整する必要があります。
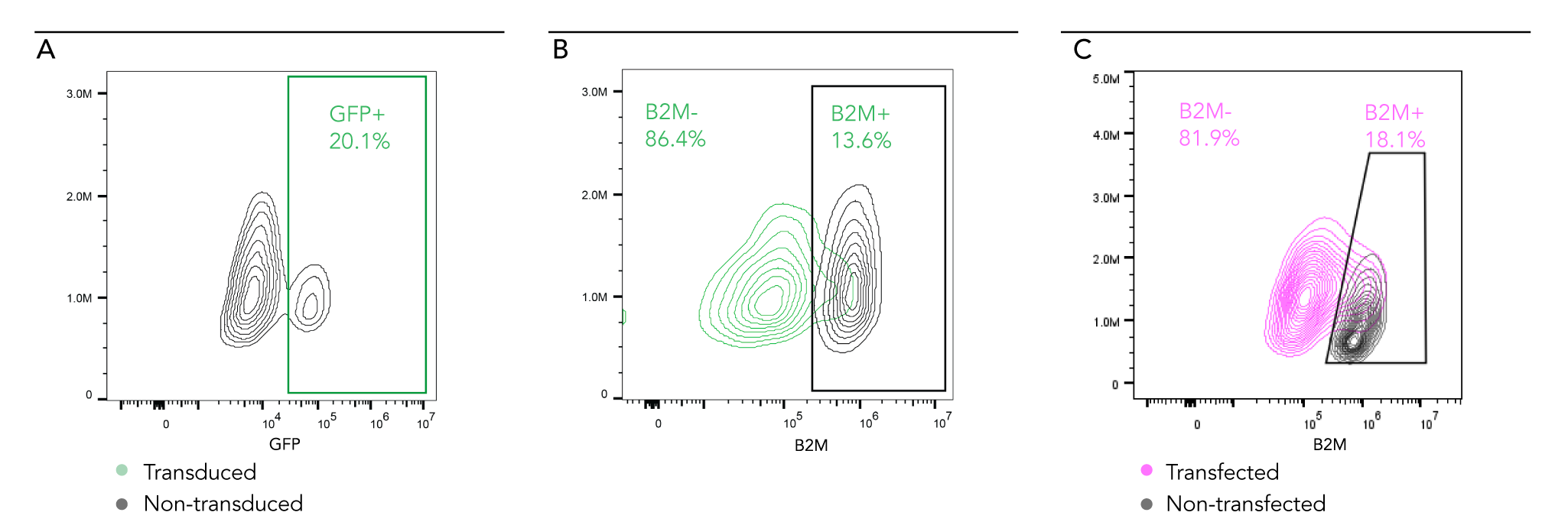
図 フローサイトメトリー解析によるノックアウト効率の評価
CRISPR-Ready ioMicroglia (Male) に、レンチウイルスまたは脂質ベースのトランスフェクション試薬(Lipofectamine RNAiMAX)を用いてbeta-2 microglobulin (B2M) 遺伝子を標的とするgRNAを導入した。gRNAは、細胞播種後10日目に導入した。gRNA導入から5日後、フローサイトメトリー解析を行った。(A) レンチウイルスによるB2M gRNA導入: 20%の細胞にgRNAが導入された (GFPの発現で評価)。(B) GFP陽性細胞のうち86%でB2Mノックアウトが確認された。(C) 脂質ベースのトランスフェクション試薬を用いた群では、約82%でB2Mノックアウトが確認された。
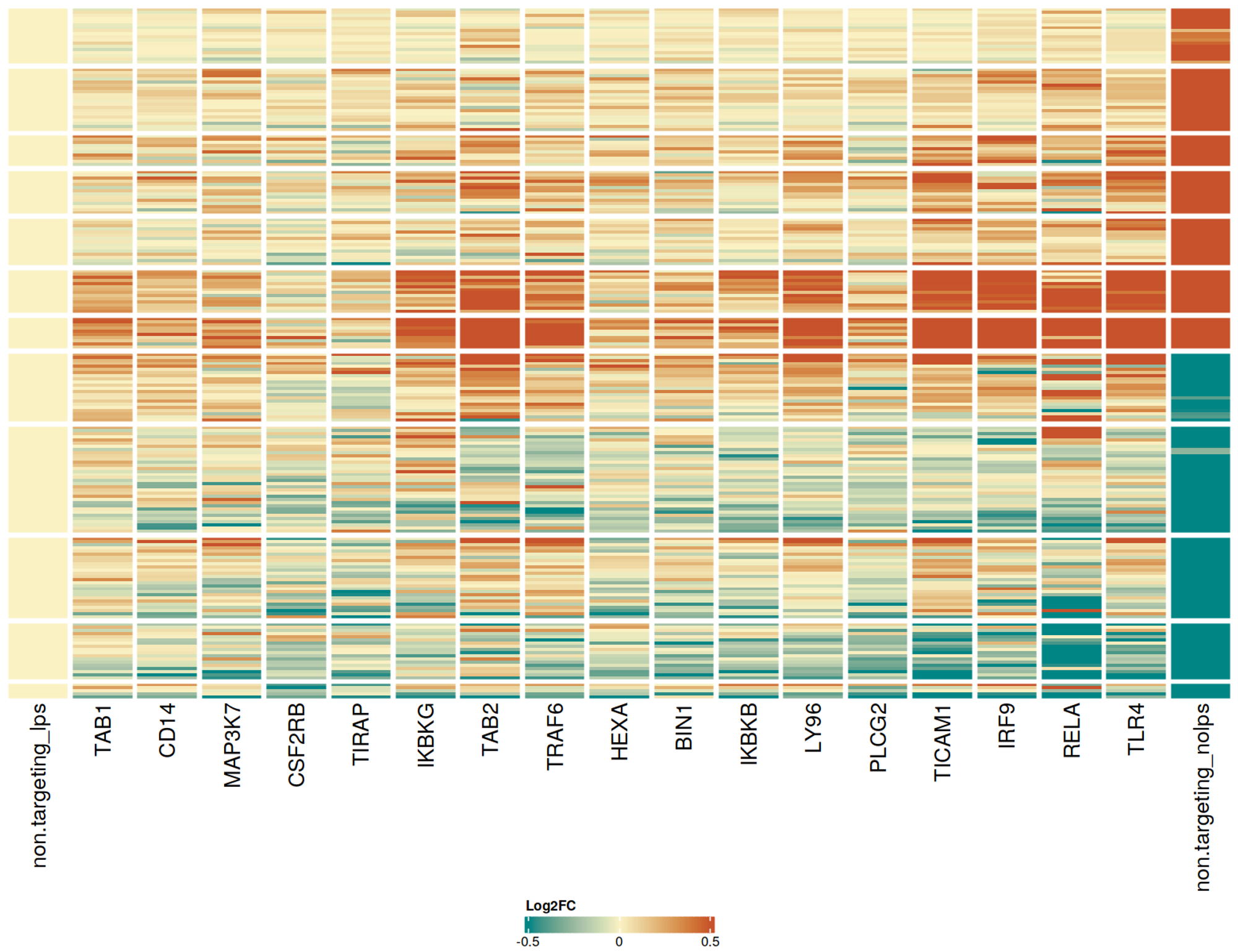
図 シングルセルCRISPRスクリーニングによるミクログリア活性化調節因子の解析
CRISPR-Ready ioMicroglia にLPSを処理し、未処理群と比較し遺伝子発現の変化があった258種類の遺伝子を同定した。別途、神経変性および神経炎症に関連する110種類の候補遺伝子を選択した。これらの遺伝子についてgRNAを用意し、播種後10日目のCRISPR-Ready ioMicrogliaにレンチウイルスを用いてgRNAを導入した。細胞を培養、LPS (+/-) を処理し、24時間後にシングルセルを回収した (day 15)。コサイン類似度の分析では、LPS 処理した CRISPR-Ready ioMicroglia のノックアウト細胞を、休止状態と活性化状態の両方と比較し、17種類の遺伝子がLPS刺激に対する応答に影響を及ぼすことを示した。コサイン類似度0.3以上(任意のしきい値)の遺伝子ノックアウトについてのLog2FCプロファイルをヒートマップ表示した。CD14, MAP3K7, TIRAP, IKBKG, TRAF6, IKBKB, LY96, TICAM1, RELA, TLR4 などのTLR4シグナル伝達経路を介したLPS刺激に関与する遺伝子が検出された。
| 品名 | メーカー | 品番 | 包装 | 希望販売価格 |
|---|---|---|---|---|
CRISPRko-Ready ioMicroglia Male for commercial users |
BIT | IO1094S | 1 SET [2 vials, >1.5 x 10^6 viable cells/vial] |
お問い合わせ |
商品は「研究用試薬」です。人や動物の医療用・臨床診断用・食品用としては使用しないように、十分ご注意ください。
※ 表示価格について
© COSMO BIO