● 非常に高品質:配列の最適化を行うことで、長鎖配列(200bp〜60kb、最長:約400kb)の配列やクローニングが難しい配列も合成します。
本サービスは、ATUM社オフィス(米国カリフォルニア州)で行います。
● 制限酵素部位、プロモーター、その他モチーフ配列の追加・除去のアレンジはフレキシブルに行えます。
● ATUM社独自の GeneGPS™ アルゴリズム(図1〜3) により、最大限のタンパク質発現が得られるよう配列の最適化が可能です。
● 様々なベクターにクローニング可能です。
ATUM社の発現ベクター(大腸菌、哺乳類、酵母用)またはお客様のお持ちのベクターからお選びいただけます。
● サブクローニングやライゲーション操作は不要です
● 遺伝子バリアントやライブラリーも迅速に合成します。
● デザイン用のソフトウェア(Gene Designer 2.0)を無償でご利用いただけます。
最大限のタンパク質発現が得られるよう配列を最適化いたします!
GeneGPS™アルゴリズムについて
GeneGPS™アルゴリズム(特許取得済)を用いて遺伝子の最適化を行うと、他社のアルゴリズムでデザインされたものと比較して、10〜100倍ものタンパク質量が得られます。
この驚くべき結果は、下記論文で報告されています。
Welch M, Govindarajan S, Ness JE, Villalobos A, Gurney A, et al. (2009) Design Parameters to Control Synthetic Gene Expression in Escherichia coli. PLoS ONE 4(9): e7002. doi:10.1371/journal.pone.0007002
膜タンパク質といった発現が難しいタンパク質でさえも、ATUM社の技術でより高レベルの発現が得られます。
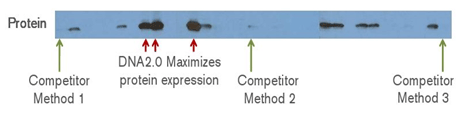
図1. S.cerevisiaeでのヒト膜タンパク質発現
・膜分画のトータルタンパク質
・WT遺伝子の発現は検出されず
・最大の発現レベル〜1mg/mL

図2. 他社アルゴリズムとの比較
大腸菌で発現させたトータルタンパク質(Triplicate)をPAGEで分離し、クマシー染色。
A:ATUM社GeneGPS™ アルゴリズム
B:他社アルゴリズム
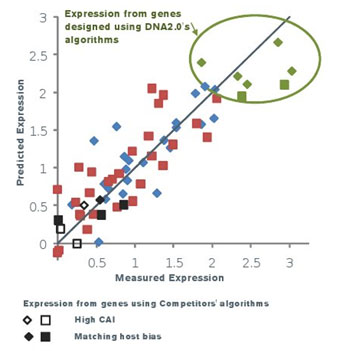
図3. ATUM社アルゴリズムと実際のタンパク質発現量の相関性
ポリメラーゼバリアント(■印)とscFv抗体バリアント(◆印)の発現を示した。各印は、異なるコドンバイアス由来のデータを示し、ATUM社のアルゴリズムで合成した遺伝子は緑色で示した。黒印は、他社が使用した2種類の主なアルゴリズムを示し、◆■印は大腸菌ゲノムバイアスにマッチングに基づくアルゴリズム、◇□印はCAI(Codon Adaptation Index)に基づいたアルゴリズムを使用している。
分子生物学研究者のためのDNAデザインツール
Gene Designer 2.0 について
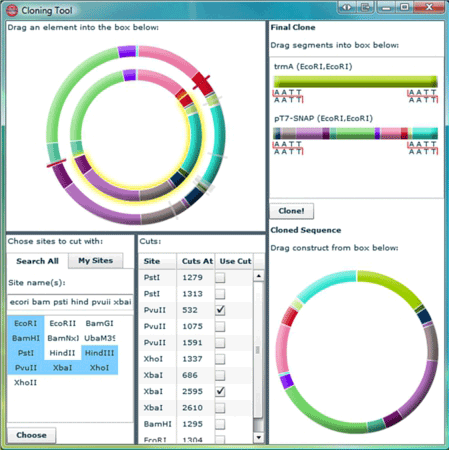
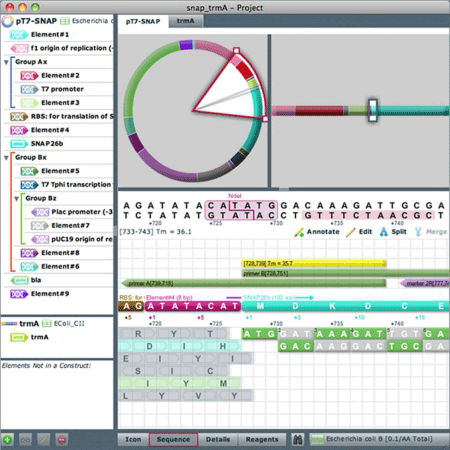
● 制限なくde novoで配列をデザインできるソフトウェアです。
● グラフィックが多くて使いやすく、目的のコンストラクトを簡単にアノテート/編集できます。
● コンストラクトや遺伝子、遺伝因子を迅速に追跡/処理/保存してカスタマイズできます。
● 画面上で、配列因子、コドンの選択、オリゴヌクレオチドの位置を簡単に操作でき、ドラッグ&ドロップで簡単に動かせます。
● オープンリーディングフレームや制限酵素サイト、モチーフ配列をサーチできます。
● オープンリーシングフレームを再コード化し、翻訳フレームや融合部位をチェックできます。
● リアルタイムな融点計算機能で、シーケンシングプライマーのオリゴヌクレオチドを一瞬でデザインできます。
ご注意:Gene Designer 2.0は、ご自身で簡単に配列のデザインや一次的な最適化をしていただく為のソフトウェアで、GeneGPS™ アルゴリズムは含まれておりません。GeneGPS™ アルゴリズムによる最適化がご必要な場合には、御見積依頼時にご指定ください。























 このページを印刷する
このページを印刷する