Spike-In RNA Variant(SIRV)Control は、トランスクリプトームの複雑性を模した人工的な転写産物のセットで、試料(組織や細胞の溶解液、精製RNA)に添加し使用します。SIRVsは最終的なシーケンスリードのうち1-2% を占めます。SIRVsを使用することで、RNA シーケンス解析パイプラインや、サンプル調製毎の手技的ノイズを評価するための正確性や精度といった値が得られます(図1、Evaluation)。実験間での整合性はこれらの値や変動係数(CoD)によって算出されます(図1、Comparison)。
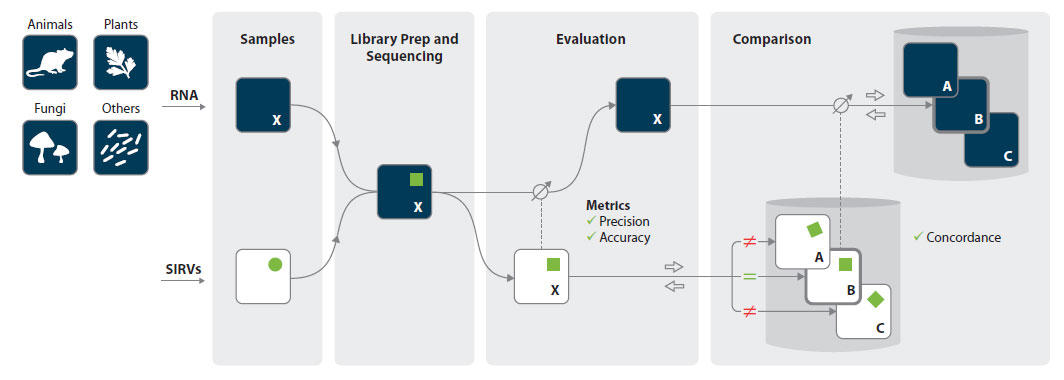
図.1 RNAシーケンス解析におけるSpike-In RNA Variant(SIRV)Control の利用
Spike-In RNA Variants(SIRVs)を試料に少量添加し、ライブラリ作製を行う。用いた試料のゲノムとSIRV にリードをマッピングした後、SIRV コントロールのデータを基準として、解析データを評価する。RNA-Seqワークフローの評価により、バイアスや盲点が明らかになります。また、このコントロールのデータは、作製
したライブラリが様々な解析に使用可能な精度か否かの判断基準となります。
モジュールコンセプト
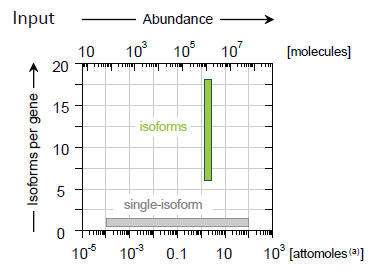
RNA spike-in control として、アイソフォームモジュール、 ERCC モジュール、long SIRV モジュールを利用可能です(図2)。
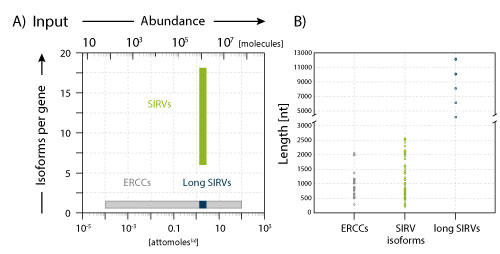
図.2 各 SIRV モジュールのアイソフォーム、転写産物量、鎖長
SIRV アイソフォーム Mix E0 は SIRV 転写産物が等モルで混合されており、アイソフォームの複雑性を表現しているが、ERCC とlong SIRVは1遺伝子について1アイソフォームのみ含まれ、転写産物量や鎖長の複雑性を表現している。
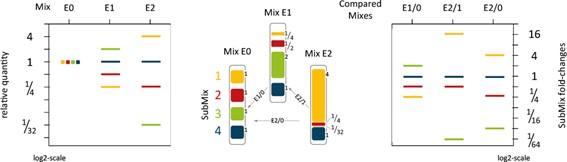
(注)SIRV-Set1には、SIRVアイソフォームが異なるモル比率で混合されている。 Mix E1(〜 8 倍)、Mix E2(〜 128 倍)
アイソフォームモジュール
SIRV アイソフォームモジュールは、ヒトの7つのモデル遺伝子に由来する69種類の転写バリアントから成り、トランスクリプトームの複雑性を模しています(図 3)。このモジ ュールは選択的スプライシング、選択的な転写開始 / 転写終結、重複遺伝子、アンチセンス転写産物等を反映しています。 6 種類または 18 種類の転写バリアントを用いることで十分なアイソフォームの複雑性を生み出し、厳密な RNA シークエンスワークフローを可能にします1。また、SIRV アイソフ ォームは、異なるシークエンサーや解析パイプラインを用いて作成したデータの整合性の評価にも利用されています2。 SIRV アイソフォームは SIRV 転写産物が等モルで混合されている Mix E0、異なるモル比率で混合されている Mix E1(〜8倍)、Mix E2(〜 128 倍)で構成されています。
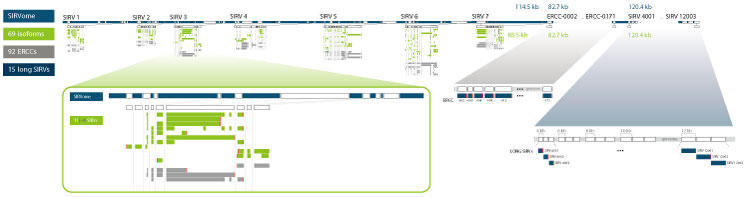
図.3 SIRVs のデザイン
SIRV1 から SIRV7 はヒトモデル遺伝子を模倣しており、主要な選択的スプライシングや転写を表している。7 種類の SIRV アイソフォーム遺伝子と 92 種類の ERCC遺伝子を人工的な染色体“SIRVome”として示す。SIRV3 を拡大し、11 種類の転写バリアントを緑色で示す。灰色で示した転写バリアントは他の評価法のための追加アノテーションである。ERCC モジュールは、1 遺伝子 1 転写産物のコンセプトで 106 のダイナミックレンジをカバーする。long SIRV には 5 つの長さのカテゴリ(4 kb, 6 kb, 8 kb, 10 kb, 12 kb)それぞれに 3 つの異なる転写産物が含まれる。
ERCC モジュール
ERCC モジュールは、外部 RNA 標準コンソーシアム(ERCC)により開発された重複のない 92 種類の人工的な転写産物です。ユニークな配列特性から、ERCC コントロールはアイソフォームに関わりなく各種パラメーターの評価に最適です。 ERCC Mix 1 は 220(106)のダイナミックレンジをカバーしており、幅広い転写産物濃度に対応しています 3, 4。濃度既知のリードを比較することでダイナミックレンジ、用量反応、検出限界、ワークフローの効率を評価することが可能です。
Long SIRV モジュール
ロングリードシークエンス解析プラットフォームの導入により、リード長は著しく上昇し、現在では転写産物の平均の長さを超えるリード長となっています。Lexogen社では、「Long SIRV」を開発しました。5 つの長さのカテゴリ(4 kb, 6 kb, 8 kb, 10 kb, 12 kb)それぞれに 3 つの異なる転写産物が含まれます。これら 15 種類の RNA の配列はユニークであり、他の spike-in control または内在性転写産物とも重複しておらず、RNA-Seq ワークフローで転写産物長を評価するための最適なツールとなっています。
図 4 に、locus SIRV6 におけるアイソフォーム Mix E0(18 転写産物)の予想リードカバレッジを例として示しました。予測リードと測定リードの差異は変動係数(CoD)として RNA-Seq のパフォーマンスとバイアスの目安となります1。
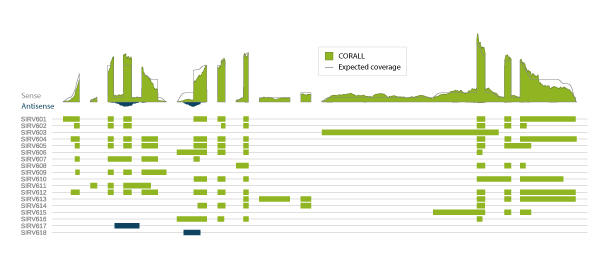
図.4 SIRV6 のカバレッジ
locus SIRV6 における 18 転写産物のエキソン - イントロン構造を下パネルに示す(アンチセンス転写産物は青色で示す)。 CORALL RNA-Seq Library Preparation Kit で作製したライブラリについてリードを SIRVome にマップし、locus レベルで可視化した。
ERCC モジュールは、input-output 濃度相関測定や、検出限界(LLoD)の算出に最適です(図 5A)。ある遺伝子について複数のアイソフォームが広い濃度範囲で分布しているより複雑な設定の input-output 相関は、アイソフォーム Mix E0、E1、および E2 を用いて評価可能です。さらに、転写産物レベルで発現変化を見る際、RNA-Seq パイプラインを評価することが可能です(図 5B)。
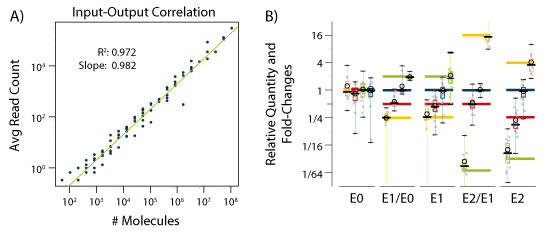
図.5 ERCC および SIRV アイソフォームモジュールを用いた input-output 相関測定
A) SIRV3 セットに含まれる ERCC Mix 1(1遺伝子 1転写産物)を QuantSeq 3' mRNA-Seq で評価した。 R2 は 0.972 で相関が高かった。
B) SIRV Mix E0,E1,E2 に含まれる転写産物濃度の概要。結果は既知のインプットを参照して示しており、色付きのバーは SIRV サブミックス 1:1(E0),1:8(E1), 1:128(E2)の濃度比率を示す。黒丸は平均値、灰色は中央値を示す。四角はデータポイントの 25 〜 75%の領域におよび、連結線のある黒い線は最小値と最大値で、グラフの外れ値も示す。
スパイクインRNA シーケンス実験のデータは、リードがゲノムDNA のリファレンス配列やSIRVome にマッピングされる段階まで一様に処理されます。バイオインフォマティックツールは、リードマッピング(いわゆる生データ)から転写産物を同定、定量するまでの各段階において、検出されたリード分布と予想リード分布を比較するものです。
データ解析ワークフローとして、無料で利用可能なSIRV Suite がおすすめです。SIRV Suite はデータ解析プラットフォームGalaxyのツールやアルゴリズムを利用しており、実験間の比較を行うだけでなく、SIRV 実験のデザインや評価が可能です。ERCC リードの評価には、NIST が提供している“ERCC dashboard”5 と呼ばれるソフトウェアパッケージやSEQC/MAQC-III コンソーシアムが紹介している評価法6 が利用可能です。
算出されたクオリティ値は、正確性や精度、測定されたカバレッジと予想カバレッジ間の変動係数(Coeffi cient of Deviation;CoD)を含みます(図2)。データ比較において、決定的なパラメーターはバイアスの程度よりもバイアスの一貫性であり、実験間の差は一貫して蓄積されたSIRVs を基に同定されます。このように、データセットが比較可能かは情報に基づき判断ができ、実験固有の変動に対してベースラインを設定することが可能です。
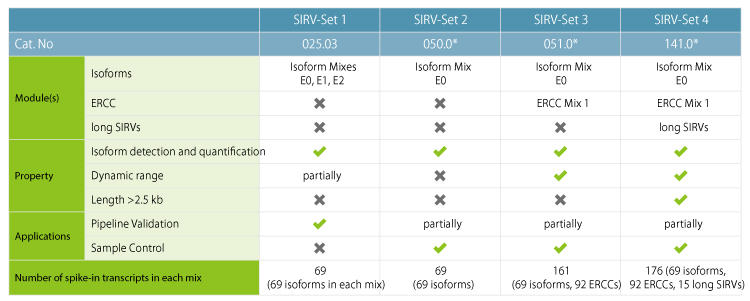
用途によって 4 種類の SIRV セットをご用意しています。
詳細なアイソフォーム解析には SIRV-Set 1、データセットの比較には SIRV-Set 2、濃度の検証には SIRV-Set 3、long spike-in RNA を利用した全体的な評価には SIRV-Set 4 を利用可能です。
表.1 SIRVセットのセレクションガイド
SIRV-Set 1(品番:025.03)はアイソフォームモジュールのアイソフォーム Mix E0、E1 および E2 を含みます。SIRV-Set 2(品番:050.0)はアイソフォーム Mix E0 のみです。SIRV-Set 3(品番:051.0)はアイソフォーム Mix E0 と ERCC との混合物です。SIRV-Set 4(品番:141.0)は long SIRV、SIRV アイソフォーム Mix E0、ERCC の混合物です。
* バイアル数、1 または 3
✓:適用あり、-:適用なしまたは部分的に適用可
























 このページを印刷する
このページを印刷する