アンチセンス技術の基礎となる概念はそれほど難しくはありません。特定の mRNAに相補的な配列を使ってハイブリダイゼーションすることでその発現を阻害し、DNAからタンパク質への遺伝情報の伝達を阻害します31。近年、治療薬として、アンチセンスオリゴヌクレオチド技術により開発されたビトラベン (サイトメガロウイルス網膜炎用32) の商業化が初めてFDAで承認され、その後、多くの治療用オリゴヌクレオチド33の臨床試験が行われています。ホスホロチオエートオリゴヌクレオチドは、アンチセンス用途 (例えば、Vitravene) で使われた最初の修飾オリゴです。それらが持つエンドヌクレアーゼ耐性やRNase H活性により、RNaseH 指向性および立体阻害アンチセンスに対しての適した候補になります。しかし、オリゴヌクレオチドが高度のホスホロチオール化を有する場合、非特異的結合が起こることが知られています34。RNAへの結合効率が DNA よりもずっと低いという問題もあります。それにもかかわらず、アンチセンスオリゴヌクレオチド治療薬の開発において、完全なホスホロチオエートや一部のホスホロチオエートオリゴが依然として使用されています。
このような問題を解決するために、塩基や糖、リン酸化修飾が開発されています。これらの「第二世代」オリゴヌクレオチドは、細胞内ヌクレアーゼによる分解に抵抗性があり、同じ配列のホスホジエステルやホスホロチオエートよりも高い親和性で、その標的mRNAに特異的にハイブリダイズします。しかし、このようなアンチセンス効果は RNase H非依存性メカニズムに起因します。最も一般的なオリゴヌクレオチド修飾には、2-O-メチル基があります。これらのオリゴヌクレオチドは、標的mRNAと高融点のヘテロ二重鎖を形成35し、RNase H非依存的なメカニズム36、すなわち立体障害を介してアンチセンス効果を誘導します。また、天然のリン酸リボース骨格を持たない安定なオリゴも作製されています。PNAは、N-(2-アミノエチル)グリシンの繰り返し単位からなる非荷電で柔軟なポリアミド骨格を持ち、これに塩基が結合しています。
これらのオリゴマーは、核酸、つまり一本鎖、二本鎖DNAまたはRNAと非常に安定な二重鎖または三重鎖を形成することができます37。高親和性核酸結合の特性は、PNAオリゴマー上に負電荷がないために静電反発がないことで説明できます。PNAは RNase H や他の RNaseの基質にはならないので、PNAのアンチセンス機構は立体障害に依存します。また、PNAはDNAに結合し、RNAポリメラーゼの開始・伸長を阻害38したり、転写因子の結合・作用を阻害することもあります39。その他にも PNAは mRNA に結合し、スプライシング40や翻訳の開始・伸長を阻害することも可能です41。
このような修飾はそれ自体もアンチセンスの応用に有効であること証明されています。ホスホロチオエート結合の使用など、これらの修飾の組み合わせることで劇的な改良がなされたのです。キメラオリゴヌクレオチドを用いると、つまり RNase Hに適合する部分 (通常はホスホロチオエート部分) の片側または両側の末端に、より親和性の高い修飾RNA領域42 (通常は2'-O-アルキルオリゴボヌクレオチド) を配置すると、特異性や効果を高めることができます。この置換は、オリゴヌクレオチドの標的への親和性を高めるだけでなく、RNase Hによる非標的mRNAの切断を減少させます43。
「第2世代」アンチセンスオリゴヌクレオチドの他の例には、ホスホロジアミデートモルフォリノオリゴマー44、およびリボースの3 '位の酸素がアミン基で置換された結果として生じるN3'-P5'PNがあります45。
商品情報
参考文献
31 Antisense Oligonucleotides: Basic Concepts and Mechanisms, N. Dias and C. A. Stein, Mol. Cancer Ther., 1, 347, 2002.
32 (a) Technology evaluation: fomivirsen. Isis Pharmaceuticals Inc/CIBA vision, R.M. Orr, Curr. Opin. Mol. Ther., 3, 288-294, 2001; (b) Fomivirsen approved for CMV retinitis, B. Roehr, J. Int. Assoc. Physicians AIDS Care, 4, 14-16, 1998.
33 Antisense therapeutics: is it as simple as complementary base recognition?, S. Agrawal and E.R. Kandimalla, Mol. Med. Today, 6, 72-81, 2000.
34 (a) Mac-1 (CD11b/CD18) is an oligodeoxynucleotide-binding protein, L. Benimetskaya, J.D. Loike, Z. Khaled,G. Loike, S.C. Silverstein, L. Cao, J. el Khoury, T.Q. Cai and C.A. Stein, Nat. Med., 3, 414-420,1997; (b) Controversies in the cellular pharmacology of oligodeoxynucleotides, C.A. Stein, Ciba Found. Symp., 209, 79-89, 1997; (c) Phosphorothioate oligodeoxynucleotides bind to basic fibroblast growth factor, inhibit its binding to cell surface receptors, and remove it from low affinity binding sites on extracellular matrix, M.A. Guvakova, L.A. Yakubov, I. Vlodavsky, J.L. Tonkinson and C.A. Stein, J. Biol. Chem., 270, 2620-2627, 1995; (d) Inhibition of high affinity basic fibroblast growth factor binding by oligonucleotides, S.M. Fennewald and R.F. Rando. J. Biol. Chem., 270, 21718-21721, 1995.
35 Evaluation of 2’-modified oligonucleotides containing 2’-deoxy gaps as antisense inhibitors of gene expression, B.P. Monia, E.A. Lesnik, C. Gonzalez, W.F. Lima, D. McGee, C.J. Guinosso, A.M. Kawasaki, P.D. Cook and S.M. Freier, J. Biol. Chem., 268, 14514-14522, 1993.
36 2’-O-(2-Methoxy)ethyl-modified anti-intercellular adhesion molecule 1 (ICAM-1) oligonucleotides selectively increase the ICAM-1 mRNA level and inhibit formation of the ICAM-1 translation initiation complex in human umbilical vein endothelial cells, B.F. Baker, S.S. Lot, T.P. Condon, S. Cheng-Flournoy, E.A. Lesnik, H.M. Sasmor and C.F. Bennett, J. Biol. Chem., 272, 11994-12000, 1997.
37 (a) Sequence-selective recognition of DNA by strand displacement with a thymine-substituted polyamide, P.E. Nielsen, M. Egholm, R.H. Berg and O. Buchardt, Science, 254, 1497-1500, 1991; (b) Kinetics for hybridization of peptide nucleic acids (PNA) with DNA and RNA studied with the BIAcore technique, K.K. Jensen, H. Orum, P.E. Nielsen and B. Norden. Biochemistry, 36, 5072-5077, 1997.
38 Effects in live cells of a c-myc anti-gene PNA linked to a nuclear localization signal, G. Cutrona, E.M. Carpaneto, M. Ulivi, S. Roncella, O. Landt, M. Ferrarini and L.C. Boffa, Nat. Biotechnol., 18, 300-303, 2000; (b) Invasion of the CAG triplet repeats by a complementary peptide nucleic acid inhibits transcription of the androgen receptor and TATA-binding protein genes and correlates with refolding of an active nucleosome containing a unique AR gene sequence, L.C. Boffa, P.L. Morris, E.M. Carpaneto, M. Louissaint and V.G. Allfrey, J. Biol. Chem., 271, 13228-13233, 1996; (c) Antisense and antigene properties of peptide nucleic acids, J.C. Hanvey, N.J. Peffer, J.E. Bisi, S.A. Thomson, R. Cadilla, J.A. Josey, D.J. Ricca, C.F. Hassman, M.A. Bonham, K.G. Au et al, Science, 258, 1481-1485, 1992.
39 inhibition of NF-K B specific transcriptional activation by PNA strand invasion, T.A Vickers, M C Griffith, K Ramasamy, L M Risen, and S M Freier, Nucleic Acids Research, 23, 3003-3008, 1995.
40 Peptide nucleic acids are potent modulators of endogenous pre-mRNA splicing of the murine interleukin-5 receptor-α chain, J.G. Karras, M.A. Maier, T. Lu, A. Watt and M. Manoharan, Biochemistry, 40, 7853-7859, 2001.
41 (a) Inhibition of promyelocytic leukemia (PML)/retinoic acid receptor-α and PML expression in acute promyelocytic leukemia cells by anti-PML peptide nucleic acid, L. Mologni, E. Marchesi, P.E. Nielsen and C. Gambacorti-Passerini, Cancer Res., 61, 5468-5473, 2001; (b) In vitro transcriptional and translational block of the bcl-2 gene operated by peptide nucleic acid, L. Mologni, P.E. Nielsen and C. Gambacorti-Passerini, Biochem. Biophys. Res. Commun., 264, 537-543,1999; (c) Antisense PNA tridecamers targeted to the coding region of Ha-ras mRNA arrest polypeptide chain elongation, N. Dias, S. Dheur, P.E. Nielsen, S. Gryaznov, A. Van Aerschot, P. Herdewijn, C. Helene and T.E. Saison-Behmoaras, J. Mol. Biol., 294, 403-416, 1999; (d) Antisense inhibition of gene expression in bacteria by PNA targeted to mRNA, L. Good and P.E. Nielsen, Nat. Biotechnol., 16, 355-358, 1998; (e) In vitro transcription and translation inhibition by anti-promyelocytic leukemia (PML)/retinoic acid receptor α and anti-PML peptide nucleic acid, C. Gambacorti-Passerini, L. Mologni, C. Bertazzoli, P. le Coutre, E. Marchesi, F. Grignani and P.E. Nielsen, Blood, 88, 1411-1417, 1996.
42 (a) Characterization of a potent and specific class of antisense oligonucleotide inhibitor of human protein kinase C-α expression, R.A McKay, L.J. Miraglia, L.L. Cummins, S.R. Owens, H. Sasmor and N.M. Dean, J. Biol. Chem., 274, 1715-1722, 1999; (b) Selecting optimal oligonucleotide composition for maximal antisense effect following streptolysin O-mediated delivery into human leukaemia cells, R.V. Giles, D.G. Spiller, J. Grzybowski, R.E. Clark, P. Nicklin and D.M. Tidd, Nucleic Acids Research, 26, 1567-1575, 1998.
43 (a) Mixed-backbone oligonucleotides as second generation antisense oligonucleotides: in vitro and in vivo studies, S. Agrawal, Z. Jiang, Q. Zhao, D. Shaw, Q. Cai, A. Roskey, L. Channavajjala, C. Saxinger and R. Zhang, Proc. Natl. Acad. Sci. USA, 94, 2620-2625, 1997; (b) Impact of mixed-backbone oligonucleotides on target binding affinity and target cleaving specificity and selectivity by Escherichia coli RNase H, L.X. Shen, E.R. Kandimalla and S. Agrawal, Bioorg. Med. Chem., 6, 1695-1705, 1998.
44 Morpholino antisense oligomers: design, preparation, and properties, J. Summerton and D. Weller,Antisense Nucleic Acid Drug Dev., 7, 187-195, 1997.
45 (a) Oligonucleotide N3’->P5’ phosphoramidates, S.M. Gryaznov, D.H. Lloyd, J.K. Chen, R.G. Schultz, L.A. DeDionisio, L. Ratmeyer and W.D. Wilson, Proc. Natl.Acad. Sci. USA, 92, 5798-5802, 1995; (b) Synthesis of oligodeoxyribonucleotide N3’->P5’ phosphoramidates, J.K. Chen, R.G. Schultz, D.H. Lloyd and S.M.Gryaznov, Nucleic Acids Research, 23, 2661-2668, 1995; (c) Oligonucleotide N3’->P5 phosphoramidates as antisense agents, S. Gryaznov, T. Skorski, C. Cucco, M.Nieborowska-Skorska, C.Y. Chiu, D. Lloyd, J.K. Chen, M. Koziolkiewicz and B. Calabretta, Nucleic Acids Research, 24, 1508-1514, 1996; (d) Antileukemia effect of c-myc N3’->P5’phosphoramidate antisense oligonucleotides in vivo, T. Skorski, D. Perrotti, M. Nieborowska-Skorska, S. Gryaznov and B. Calabretta, Proc. Natl. Acad. Sci. USA, 94, 3966-3971, 1997.
アンチセンスオリゴヌクレオチドとは異なり、siRNAはセンス (パッセンジャー) 鎖とアンチセンス (ガイド) 鎖からなる二重鎖です。この場合、遺伝子サイレンシングのメカニズムはより複雑になっています。まず、二重鎖は RNA誘導型サイレンシング複合体 (RNA Induced Silencing Complex:RISC) にロードされ、そこで鎖が分離されます。そして、アンチセンス鎖は、RISCの触媒成分であるアグロナウトというタンパク質を使い、標的mRNAの配列特異的切断を誘導します。
アンチセンスオリゴヌクレオチドと同様に、治療薬にヌクレアーゼ耐性を持たせることが重要です。また、アンチセンス鎖と標的mRNAの結合効率が高いことが望まれます。その結果、アンチセンス技術を向上させるために開発された「アンチセンス療法」で掲載されている修飾剤は、siRNAにも同様に適用できます。典型的な修飾は、2'-OMe、2'-F、LNA、ホスホロチオエートのような 2'-または糖修飾ヌクレオシドの組み合わせです。FDAが承認した最初のsiRNA医薬品であるhATTR治療薬パティシランは、アルニラム社が開発したsiRNAオリゴです。
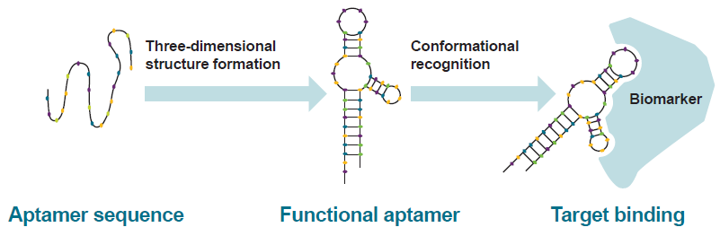
核酸である「アプタマー」は一本鎖 (ss) オリゴヌクレオチド (DNAまたはRNA) であり、明確に定義された3次元構造に折りたたまれ、目的のターゲット (小分子、タンパク質、細胞、さらには生物全体など) と相補的な相互作用を持つ形状を形成します46, 47。この「錠と鍵 ‘lock and key’ 」結合メカニズムの結果として、抗体と同等の非常に高い親和性 (ナノモルおよびピコモルの解離定数) と特異性 (> 1000倍) が備わります (図2)。重要なことは、アプタマーは抗体に比べていくつかの利点があるという点です。第一に、合成の観点から、アプタマーは費用効果の高い方法で大規模に生産でき、化学的方法によって部位特異的に修飾することが可能です48, 49。さらに、熱に対して基本的に安定であり、抗体とは違って何度も熱変性を行ってもリフォールドすることが可能です。第二に、治療への応用において、アプタマーはサイズが小さいため (抗体の約 150 kDa に対して 8-25 kDa)47、組織への浸透が早く、通常は宿主免疫系に認識されないため毒性および免疫原性が低いということが挙げられます。
図2. アプタマー-ターゲット複合体を形成するためのそのターゲットとのアプタマーコンフォメーション相互作用の概略図。
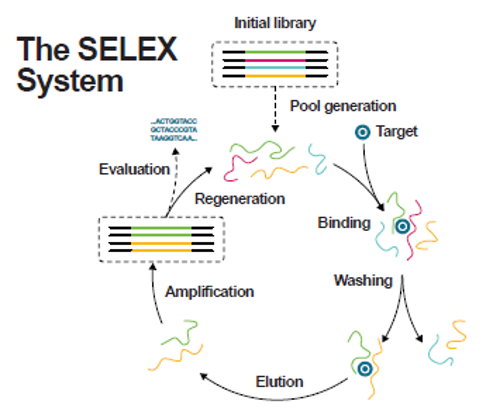
通常、アプタマーは、1990年代に開発されたSELEX(試験管内選択法)と呼ばれるin vitro選択技術51,52 により、1058〜1061 ssDNA または ssRNA分子のランダムライブラリから派生します (図2参照)。ssDNAライブラリーは、多くの場合、2本鎖のPCR産物の鎖分離によって調製されます。53 一方、ssRNAアプタマーライブラリーは、組換えT7 RNAポリメラーゼを用いた二本鎖 (ds) DNAの in vitro転写によって調製されます。51 SELEXでは、ランダムオリゴヌクレオチドライブラリーを標的分子とインキュベーションし、結合した核酸と結合していない核酸を分離し、標的から結合した核酸を溶出させて増幅した集団が次の段階の選択で「新しい」スタートライブラリとして使用されます。その後、クローニングと配列解析が行われます。従来の手作業によるSELEXでは、完了までに数週間かかることもあります。ニトロセルロース膜ろ過ベースのSELEX54,55、アフィニティークロマトグラフィー56, 57, 58 および磁気ビーズベースのSELEX59, 60, 61、キャピラリー電気泳動ベースのSELEX62, 63, 64、マイクロ流体ベースのSELEX65, 66、Cell-SELEX67, 68, 69や、あまり一般的でない技術70, 71も含め、いくつかのin vitro選択方法が研究されてきました。近年開発された技術により、SELEXに基づくアプタマーの探索は飛躍的に向上しています72。
臨床の観点から、いくつかの重要な進展がありました。抗VEGFアプタマー (ペガプタニブまたは黄斑変性症) は、血管新生 (湿性) 加齢性変性疾患 (AMD) の治療薬として承認されています。73, 74 さらに、多くのアプタマーが現在、遺伝子治療75,76,77,78、免疫療法79,80、癌治療81,82,83のために、そして分子造影剤84,85,86として、前臨床または臨床試験段階にあります。
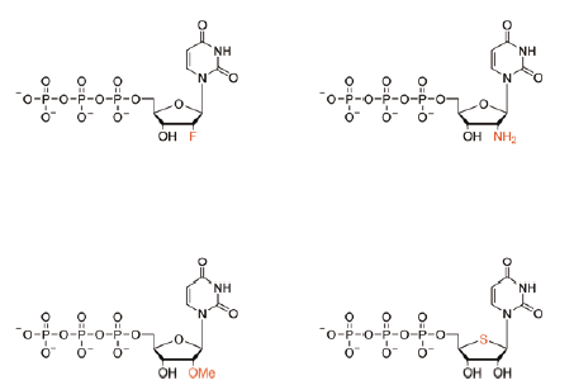
治療への応用において、天然核酸をベースとしたアプタマーは、生体内における安定性が低いことが大きな制約となっています。これらはヌクレアーゼによる分解を受けやすく、局所環境の組成 (例えば、酸性または塩基性媒体、金属イオン) に感受性があります。70,87前者の点を解決するには、2'-位置にアミノ基(-NH2)、O-アルキル基(例:-OMe)またはチオール基(-SH)を配置することです(図4)。70,88,89,90,91
図4. ヌクレアーゼ耐性の増加をもたらすアプタマー生成に使用される糖修飾ヌクレオチドの化学構造:2'-フルオロウリジン-5'-三リン酸、2'-アミノウリジン-5'-三リン酸、2'-メトキシウリジン-5'-三リン酸および4 '-チオウリジン-5'-三リン酸。
近年、クリックケミストリー (CuAAC反応) により、PCR後のステップで嵩高い修飾を導入し、アプタマーの構造多様性を大幅に高める「クリックSELEX」手法が報告されています。92これにより、SELEXのPCR増幅ステップで克服しなければならない、修飾dNTPを組み込む際のDNAポリメラーゼの限界に起因する問題を排除することができます。93
さらに、4つの天然塩基に加え、2つの疎水性人工塩基を含むDNAアプタマーを発見し、アプタマーの親和性と標的特異性も向上したという報告もあります。94
参考文献
46 Aptamers: an emerging class of therapeutics, Nimjee, S. M., Rusconi, C. P. & Sullenger, B. A, Annual Review of Medicine 56, 555-583, doi:10.1146/annurev. med.56.062904.144915 (2005).
47 Oligonucleotide aptamers: new tools for targeted cancer therapy, Sun, H. et al.,Molecular Therapy. Nucleic Acids 3, e182, doi:10.1038/mtna.2014.32 (2014).
48 Preclinical and phase 1A clinical evaluation of an anti-VEGF pegylated aptamer (EYE001) for the treatment of exudative age-related macular degeneration. Retina (Philadelphia, Pa.) 22, 143-152 (2002).
49 Anti-vascular endothelial growth factor therapy for subfoveal choroidal neovascularization secondary to age-related macular degeneration: phase II study results. Ophthalmology 110, 979-986, doi:10.1016/s0161- 6420(03)00085-x (2003).
50 A Highlight of Recent Advances in Aptamer Technology and Its Application, Sun, H. & Zu, Y, Molecules 20, 11959-11980, doi:10.3390/molecules200711959 (2015).
51 In vitro selection of RNA molecules that bind specific ligands, Ellington, A. D. & Szostak, J. W, Nature 346, 818-822, doi:10.1038/346818a0 (1990).
52 Systematic evolution of ligands by exponential enrichment: RNA ligands to bacteriophage T4 DNA polymerase, Tuerk, C. & Gold, L. Science 249, 505-510 (1990).
53 Selection of single-stranded DNA molecules that bind and inhibit human thrombin, Bock, L. C., Griffin, L. C., Latham, J. A., Vermaas, E. H. & Toole, J. J,Nature 355, 564- 566, doi:10.1038/355564a0 (1992).
54 Methods developed for SELEX, Gopinath, S. C., Analytical and Bioanalytical Chemistry 387, 171-182, doi:10.1007/s00216-006-0826-2 (2007).
55 Analytical applications of aptamers, Tombelli, S., Minunni, M. & Mascini, M., Biosensors & Bioelectronics 20, 2424-2434, doi:10.1016/j.bios.2004.11.006 (2005).
56 In vitro selection and characterization of RNA aptamers binding thyroxine hormone, Levesque, D., Beaudoin, J. D., Roy, S. & Perreault, J. P.,The Biochemical Journal
403, 129-138, doi:10.1042/BJ20061216 (2007).
57 Gold nanoparticle-based colorimetric detection of kanamycin using a DNA aptamer, Song, K. M. et al., Analytical Biochemistry 415, 175-181, doi:10.1016/j.ab.2011.04.007 (2011).
58 In vitro selection of DNA aptamers that bind L-tyrosinamide, Vianini, E., Palumbo, M. & Gatto, B., Bioorganic & Medicinal Chemistry 9, 2543-2548 (2001).
59 ssDNA aptamers that recognize diclofenac and 2-anilinophenylacetic acid, Joeng, C. B., Niazi, J. H., Lee, S. J. & Gu, M. B., Bioorganic & Medicinal Chemistry 17, 5380-5387, doi:10.1016/j.bmc.2009.06.044 (2009).
60 Single-stranded DNA aptamers specific for antibiotics tetracyclines, Niazi, J. H., Lee, S. J. & Gu, M. B.,Bioorganic & Medicinal Chemistry 16, 7245-7253, doi:10.1016/j.bmc.2008.06.033 (2008).
61 In vitro selection of high-affinity DNA aptamers for streptavidin, Wang, C., Yang, G., Luo, Z. & Ding, H., Acta biochimica et biophysica Sinica 41, 335-340 (2009).
62 In vitro selection of high-affinity DNA ligands for human IgE using capillary electrophoresis, Mendonsa, S. D. & Bowser, M. T.,Analytical Chemistry 76, 5387-5392,doi:10.1021/ac049857v (2004).
63In vitro selection of aptamers with affinity for neuropeptide Y using capillary electrophoresis, Mendonsa, S. D. & Bowser, M. T., Journal of the American Chemical Society 127, 9382-9383, doi:10.1021/ja052406n (2005).
64 Capillary electrophoresis-SELEX selection of aptamers with affinity for HIV-1 reverse transcriptase, Mosing, R. K., Mendonsa, S. D. & Bowser, M. T., Analytical Chemistry 77, 6107-6112, doi:10.1021/ac050836q (2005).
65 A microfluidic SELEX prototype, Hybarger, G., Bynum, J., Williams, R. F., Valdes, J. J. & Chambers, J. P., Analytical and Bioanalytical Chemistry 384, 191-198, doi:10.1007/ s00216-005-0089-3 (2006).
66 Micromagnetic selection of aptamers in microfluidic channels, Lou, X. et al., Proceedings of the National Academy of Sciences of the United States of America 106, 2989-2994, doi:10.1073/pnas.0813135106 (2009).
67 A tenascin-C aptamer identified by tumor cell SELEX: systematic evolution of ligands by exponential enrichment, Daniels, D. A., Chen, H., Hicke, B. J., Swiderek, K. M. & Gold, L., Proceedings of the National Academy of Sciences of the United States of America 100, 15416-15421, doi:10.1073/pnas.2136683100 (2003).
68 Selection of DNA aptamers recognizing small cell lung cancer using living cell-SELEX, Kunii, T., Ogura, S., Mie, M. & Kobatake, E., The Analyst 136, 1310-1312, doi:10.1039/c0an00962h (2011).
69 Study of the molecular recognition of aptamers selected through ovarian cancer cell-SELEX, Van Simaeys, D. et al., PloS one 5, e13770, doi:10.1371/ journal.pone.0013770 (2010).
70 SELEX with modified nucleotides, Keefe, A. D. & Cload, S. T., Current Opinion in Chemical Biology 12, 448- 456, doi:10.1016/j.cbpa.2008.06.028 (2008).
71 Selection of RNA aptamers against human influenza virus hemagglutinin using surface plasmon resonance, Misono, T. S. & Kumar, P. K., Analytical Biochemistry 342, 312-317, doi:10.1016/j.ab.2005.04.013 (2005).
72 Aptamer Selection Technology and Recent Advances, Blind, M. & Blank, M., Molecular Therapy. Nucleic Acids 4, e223, doi:10.1038/mtna.2014.74 (2015).
73 Anti-VEGF aptamer (pegaptanib) therapy for ocular vascular diseases, Ng, E. W. & Adamis, A. P., Annals of the New York Academy of Sciences 1082, 151-171, doi:10.1196/annals.1348.062 (2006).
74 Pegaptanib, a targeted anti-VEGF aptamer for ocular vascular disease, Ng, E. W. et al., Nature Reviews. Drug Discovery 5, 123-132, doi:10.1038/nrd1955 (2006).
75 Synergistic inhibition of lung cancer cell invasion, tumor growth and angiogenesis using aptamer-siRNA chimeras, Lai, W. Y. et al., Biomaterials 35, 2905-2914, doi:10.1016/j.biomaterials.2013.12.054 (2014).
76 Nucleolin-targeting liposomes guided by aptamer AS1411 for the delivery of siRNA for the treatment of malignant melanomas, Li, L. et al., Biomaterials 35, 3840-3850,doi:10.1016/j.biomaterials.2014.01.019 (2014).
77 Cell type-specific delivery of siRNAs with aptamer-siRNA chimeras, McNamara, J. O., 2nd et al., Nature Biotechnology 24, 1005-1015, doi:10.1038/nbt1223 (2006).
78 Aptamer mediated siRNA delivery, Chu, T. C., Twu, K. Y., Ellington, A. D. & Levy, M., Nucleic Acids Research 34, e73, doi:10.1093/nar/gkl388 (2006).
79 CTLA4 aptamer delivers STAT3 siRNA to tumor-associated and malignant T cells, Herrmann, A. et al., The Journal of Clinical Investigation 124, 2977-2987, doi:10.1172/JCI73174 (2014).
80 Multivalent RNA aptamers that inhibit CTLA-4 and enhance tumor immunity, Santulli-Marotto, S., Nair, S. K., Rusconi, C., Sullenger, B. & Gilboa, E., Cancer Research 63, 7483-7489 (2003).
81 Cancer immunotherapy via nucleic acid aptamers, Khedri, M., Rafatpanah, H., Abnous, K., Ramezani, P. & Ramezani, M., International Immunopharmacology 29, 926- 936, doi:10.1016/j.intimp.2015.10.013 (2015).
82 Nucleic acid aptamers in cancer research, diagnosis and therapy, Ma, H. et al., Chemical Society Reviews 44, 1240-1256, doi:10.1039/c4cs00357h (2015).
83 Aptamers: A promising chemical antibody for cancer therapy, Zhou, G. et al., Oncotarget 7, 13446-13463, doi:10.18632/oncotarget.7178 (2016).
84 Diagnostic applications of gastric carcinoma cell aptamers in vitro and in vivo, Ding, F. et al., Talanta 134, 30-36, doi:10.1016/j.talanta.2014.09.036 (2015).
85 In vivo fluorescence imaging of tumors using molecular aptamers generated by cell-SELEX, Shi, H. et al., Chemistry, an Asian Journal 5, 2209-2213, doi:10.1002/asia.201000242 (2010).
86 Applications of aptamers as sensors, Cho, E. J., Lee, J. W. & Ellington, A. D., Annual Review of Analytical Chemistry 2, 241-264, doi:10.1146/annurev. anchem.1.031207.112851 (2009).
87 Instability and decay of the primary structure of DNA, Lindahl, T., Nature 362, 709-715, doi:10.1038/362709a0 (1993).
88 Enzymatic recognition of 2’-modified ribonucleoside 5’-triphosphates: towards the evolution of versatile aptamers, Lauridsen, L. H., Rothnagel, J. A. & Veedu, R. N.,Chembiochem : a European Journal of Chemical Biology 13, 19-25, doi:10.1002/cbic.201100648 (2012).
89 Post-SELEX chemical optimization of a trypanosomespecific RNA aptamer, Adler, A., Forster, N., Homann, M. & Goringer, H. U., Combinatorial Chemistry & High Throughput Screening 11, 16-23 (2008).
90 Building oligonucleotide therapeutics using non-natural chemistries, Wilson, C. & Keefe, A. D., Current Opinion in Chemical Biology 10, 607-614, doi:10.1016/j.cbpa.2006.10.001 (2006).
91 Generation of Aptamers with an Expanded Chemical Repertoire, Diafa, S. & Hollenstein, M., Molecules 20, 16643-16671, doi:10.3390/molecules200916643 (2015).
92 Click chemistry with DNA, El-Sagheer, A. H. & Brown, T., Chemical Society Reviews 39, 1388-1405, doi:10.1039/b901971p (2010).
93 A Versatile Approach Towards Nucleobase-Modified Aptamers, Tolle, F., Brandle, G. M., Matzner, D. & Mayer, G., Angewandte Chemie 54, 10971-10974, doi:10.1002/anie.201503652 (2015).
94 Generation of high-affinity DNA aptamers using an expanded genetic alphabet, Kimoto, M., Yamashige, R., Matsunaga, K., Yokoyama, S. & Hirao, I., Nature Biotechnology 31, 453-457, doi:10.1038/nbt.2556 (2013).
近年、CRISPR/Cas技術の急速な発展により、生体内の遺伝子に狙いを定めて変化を与えることができるようになり、大きな変革が起こっています。この技術は、CRISPR(Clustered Regularly Interspaced Short Palindromic Repeats)とCRISPR-Associated (Cas) nucleasesを使用して侵入したウイルスから外来DNAを認識し破壊する細菌の免疫防御機構に由来します95。
CRISPR/Casヌクレアーゼは数種類確認されており、その中でもCas9とCas12a(Cpf1とも呼ばれる)が最もよく使われています。いずれのヌクレアーゼも、遺伝子座を特異的に標的とするために、ガイドRNAと呼ばれるRNA分子を必要とします。ガイドRNA(gRNA)とヌクレアーゼは一緒になって、二本鎖DNAの両鎖を標的にして切断します。これを生体内で行うと、エラーが起こりやすいメカニズムでDNAが修復され、遺伝子が変化してしまいます。gRNAはRNA分子であるため加水分解されやすく、ヌクレアーゼが存在する細胞内の環境ではCRISPR/Casシステムの活用が妨げになることがあります。
CRISPR/CASシステムによる遺伝子編集の改善のために、分解からgRNAを保護する化学修飾が重要視されています96,97,98,99。これらの修飾には、gRNAの最初と最後の3つのヌクレオチドの間のバックボーンにホスホロチオエート結合を付加することや、2'-O-メチル基や2'-フルオロ基への置換など2'-ヒドロキシルに対する修飾が含まれます。これらの修飾は、細胞内のRNAエキソヌクレアーゼ活性からgRNAを保護しますが、Cas活性に影響を与えない gRNA配列内の特定の位置にしか配置することはできません。
蛍光標識されたgRNAはさまざまなアプリケーションで使用されています。蛍光ガイドにより、トランスフェクション後の細胞へのCas-gRNA送達のモニタリングが可能になります。さらに、蛍光gRNAを受け取った細胞は、蛍光活性化セルソーティング(FACS)でソーティングできます。これにより、CasgRNA複合体を含む細胞を濃縮できるため、編集された細胞を採取する可能性が高まります。gRNAと複合体を形成した不活性Casヌクレアーゼは、遺伝子座を切断せずにタグ付けするために使用できます。このアプローチの例では、gRNAは、分子ビーコンのアニーリング部位を含むように設計されています。これは、フルオロフォアとクエンチャーが付加された構造化オリゴヌクレオチドです100。CRISPR / Casは合成生物学にも応用されており、蛍光ガイドを用いてCRISPR/Casによる合成DNA回路を研究するアプローチもあります101。
参考文献
95 Biology and Applications of CRISPR Systems: Harnessing Nature’s Toolbox for Genome Engineering, Wright AV, Nuñez JK, Doudna JA. (2016). Cell 164(1-2): 29-44.
96 Heavily and fully modified RNAs guide efficient SpyCas9-mediated genome editing, Mir A, Alterman JF, Hassler MR, Debacker AJ, Hudgens E, Echeverria D, Brodsky
MH, Khvorova A, Watts JK, Sontheimer EJ. (2018). Nat Commun 9(1): 2641.
97 Extensive CRISPR RNA modification reveals chemical compatibility and structure-activity relationships for Cas9 biochemical activity, O’Reilly D, Kartje ZJ, Ageely EA, Malek-Adamian E, Habibian M, Schofield A, Barkau CL, Rohilla KJ, DeRossett LB, Weigle AT, Damha MJ, Gagnon KT. (2019). Nucleic Acids Res 47(2): 546-558.
98 Synthetic CRISPR RNA-Cas9-guided genome editing in human cells, Rahdar M, McMahon MA, Prakasha TP, Swayze EE, Bennett CF, and Cleveland DW. (2015). PNAS 112(51): E7110-7.
99 Chemically Modified Cpf1-CRISPR RNAs Mediate Efficient Genome Editing in Mammalian Cells, McMahon MA, Prakash TP, Cleveland DW, Bennett CF, Rahdar M. (2018).Mol Ther 26(5): 1228-1240.
100 A CRISPR/molecular beacon hybrid system for live-cell genomic imaging, Wu X, Mao S, Yang Y, Rushdi MN, Krueger CJ, and Chen AK. (2018). Nucleic Acids Res 46(13): e80.
101 Fluorescent Guide RNAs Facilitate Development of Layered Pol II-Driven CRISPR Circuits, Menn DJ, Pradhan S, Kiani S, and Wang X. (2018). ACS Synth Biol 7(8): 1929-1936.
代謝過程と環境要因に起因するDNA損傷の研究は、それらに関連する修復メカニズムとともに、遺伝的変異、神経変性疾患、癌、および老化過程の発生についてのより深い理解につながっています。
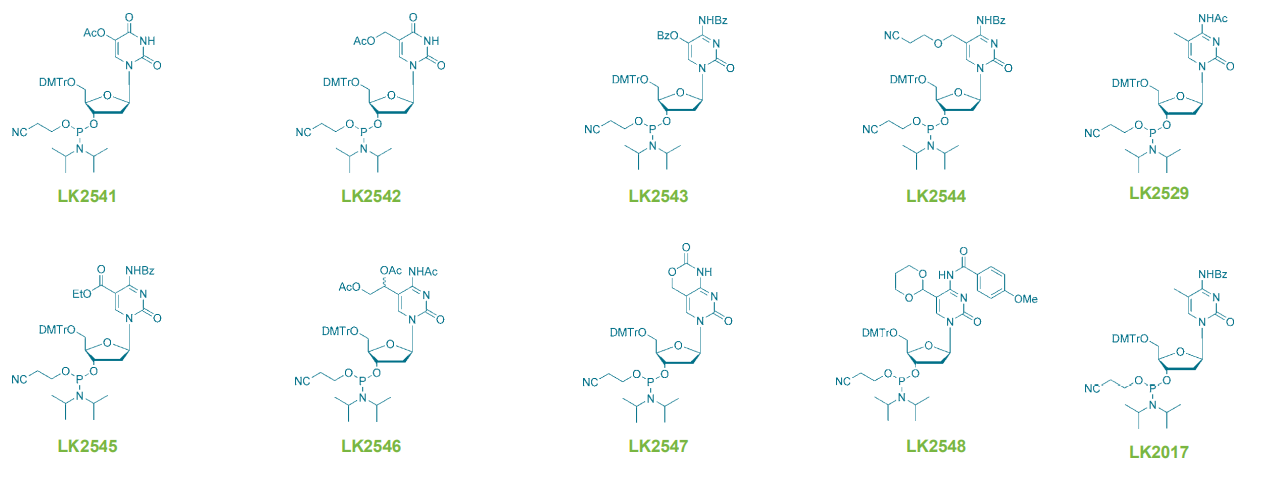
LNK社では、この研究用にさまざまな修飾子をご提供しています。以下は酸化的損傷および修復、メチル化、エピジェネティクスの研究に使用されます。5-hydroxy-dC (品番:LK2543), 5-hydroxy-dU (品番:LK2541),
5-hydroxymethyl-dU (品番:LK2542), 5-hydroxymethyl-dC (品番:LK2544), 5-carboxy-dC (品番:LK2545), 5-formyl-dC (品番:LK2546/ BA0367), 5-hydroxymethyl-dC II (品番:LK2547)、5-formyl-dC III (品番:LK2548)。
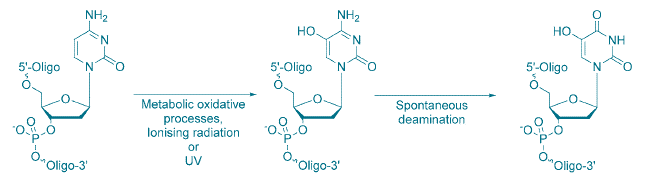
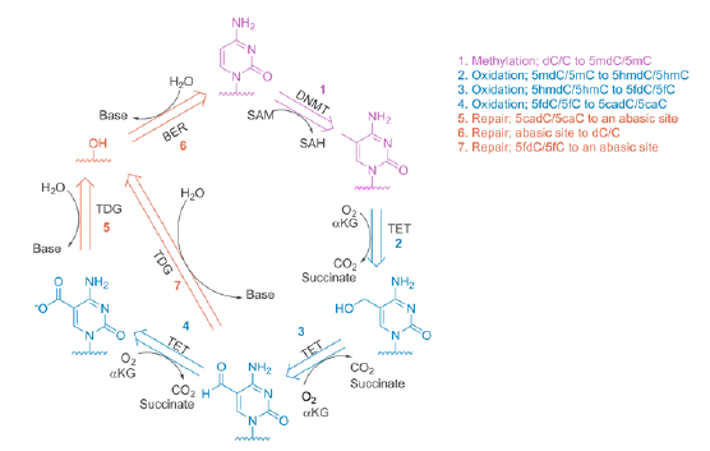
5-hydroxy-dU や 5-hydroxy-dC などの酸化ピリミジンは、酸化的代謝プロセス、UVまたは電離放射線を介してdCから誘導され、5-HO-dCを形成します。これは、自発的に脱アミノ化されて5-HO-dUを形成します(図5を参照)。
図5. dCからの5-HO-dCと5-HO-dUの生成
5-HOピリミジンをdCに戻す修復機構は存在しますが102、細胞内のDNAに一定レベルで観察されているということは、少なくともある種の細胞では、これらの修復機構が非効率的であることを示唆しています103。LK2541 または LK2543 で修飾したオリゴヌクレオチドは、このようなプロセスを理解するのに有用です。
5-HO-dU と 5-HO-dC は、それぞれ Aおよび A/C とミスマッチすることにより、突然変異を引き起こす可能性があり、修復機構が働かない場合、その突然変異は結果として遺伝子に永久的に取り込まれる可能性があります。
5-Hydroxymethyl-dU (5-hmdU、品番:LK2542)も酸化過程や電離放射線の結果生じたものですが、この場合、dTは修飾されています104。また、5-hmdU は 5-hmdC の脱アミノ化によって生成する可能性もありますが、近年、これがマウス胚性幹細胞における hmdU の定常レベルに寄与せず、dTが ten eleven translocation enzymes (Tet) の基質となって5-hmdUの生成に至ることが明らかになっています105。したがって、品番:LK2542は酸化過程とエピジェネティクスの両方を研究するための重要な試薬です。
エピジェネティクスは、コード配列に変化がないにもかかわらず、遺伝的に遺伝子を抑制することを研究する学問です。この分野への関心は、特にノンコーディングRNAによる遺伝子サイレンシング、ヒストン修飾、CpGアイランドのシチジンのDNAメチル化106によって誘発・維持される変化について、過去数年間で著しく高まっています。
品番:LK2544 ~ 品番:LK2548は、後者に適用可能なリン酸アミダイトです。オリゴヌクレオチドに組み込まれると、これらの修飾因子は、dCの修飾の生化学的経路におけるさまざまな生成物を表します(図6を参照)。
図6. Cの動的修飾の完全な経路(参考文献100から引用)。
DNAでは、シチジンはS-アデノシルメチオニンとのDNAメチルトランスフェラーゼの触媒反応によりメチル化され、5-mdCが形成されます。これがTet酵素によって5-hydroxymethyl-dCに酸化され、さらに5-formyl-dCに酸化、さらに5-carboxy-dCに酸化されます。どちらも5-carboxy-dCと5-formyl-dCはチミジンDNAグリコシラーゼを介した塩基除去修復によりdCに戻ることが可能です107。
これまで、この研究分野の製品範囲は5-methyl-dC(品番:LK2017 [N-Bz] および品番:LK2529 [N-Ac])に限定されていましたが、現在はこれら修飾を追加していますので、この分野の研究者の方に、この重要な経路に関する理解を進めるための必要なツールになります。
商品情報
参考文献
104 Oxidative damage to DNA: formation, measurement, and biological significance, J. Cadet, M. Berger, T. Douki and J.-L. Ravanat, Rev. Physiol. Biochem. Pharmacol., 131, 1-87, 1997.
105 Tet oxidizes thymine to 5-hydroxymethyluracil in mouse embryonic stem cell DNA, T. Pfaffeneder, F. Spada, M. Wagner, C. Brandmayr, S.K. Laube, D. Eisen, M. Truss, J. Steinbacher, B. Hackner, O. Kotljarova, D. Schuermann, S. Michalakis, O. Kosmatchev, S. Schiesser, B. Steigenberger, N. Raddaoui, G. Kashiwazaki,
U. Müller, C.G. Spruijt, M. Vermeulen, H. Leonhardt, P. Schär, M. Müller and T. Carell, Nat. Chem. Biol., 10 (7), 574-81, 2014.
106 Epigenetics in human disease and prospects for epigenetic therapy, G. Egger, G. Liang, A. Aparicio and P.A. Jones. Nature, 429, 457-463, 2004.
107 Tet enzymes, TDG and the dynamics of DNA methylation, R.M. Kholi and Y. Zhang, Nature, 502, 472-479, 2013.