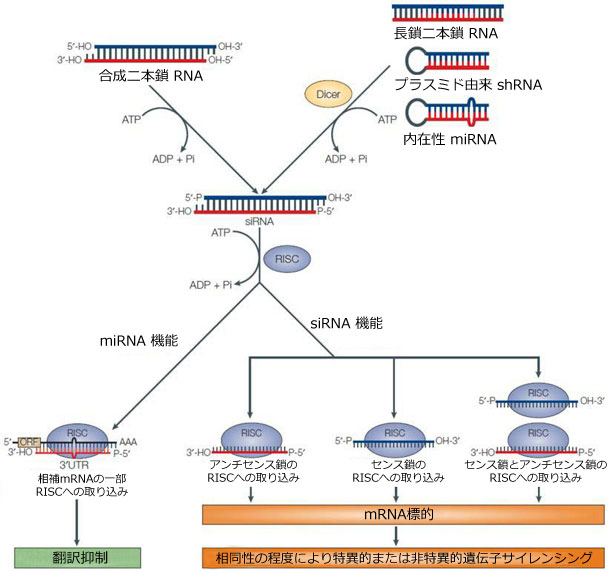
一方、ゲノム編集では遺伝子コードを変更し、通常「ノックアウト」を誘導するか、または遺伝子機能を完全除去します。この過程では、まず染色体上にDNA二本鎖切断(DSB)を生じます(Bogdanove & Voytas, 2011)。近年、高効率でDSB(DNA二本鎖切断)を作製する、TALENおよびCRISPR-Cas9の2種類のツールが開発されています(Bogdanove & Voytas, 2011; Jinek, et al., 2012; Shalem, et al., 2014; Wang, et al., 2014)。これらのいずれのツールも、植物病変形成(TALEN)、あるいは挿入変異原性からのゲノム防御(CRISPR-Cas9)といった細菌システムを利用したものです。TALENは制限エンドヌクレアーゼ Fokl に融合した部位特異的DNA結合タンパク質からなるキメラタンパク質です。一方、CRISPR-Cas9は、部位特異的な20塩基単鎖ガイドRNA(sgRNA,gRNA)を利用してCas9ヌクレアーゼをその標的座位に誘導します。TALENとCRISPR-Cas9のいずれにおいても、ヌクレアーゼが標的DNAの両鎖を切断します。この切断を修復しなくては細胞死となる可能性があるため、真核細胞では主に2種類の機序で修復応答します(図.2)。ひとつは非相同性末端結合(NHEJ)であり、染色体上の2つの遊離末端を再度結合させます。しかし、NHEJは誤りをおかしがちであり、しばしば遺伝子の破壊またはノックアウトにつながる短い挿入や欠失を生じます。ふたつめは相同組換え(HR、HDR)によるDSB(DNA二本鎖切断)修復であり、遺伝子ノックアウトを行う上でさらなる選択肢となり得ます。限定欠損の導入、挿入変異の導入、または単一塩基の置換などのアプリケーションが考えられます。
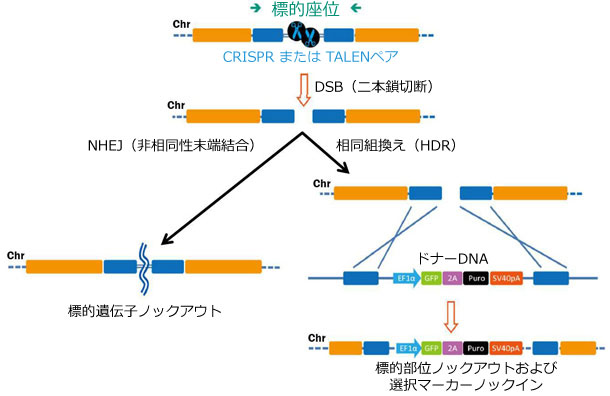
図.2 ゲノム編集ツールにより誘導されたDSB(DNA二本鎖切断)の修復経路
左:非相同性末端結合
右:ドナー鋳型存在下における相同組換え(HDR)