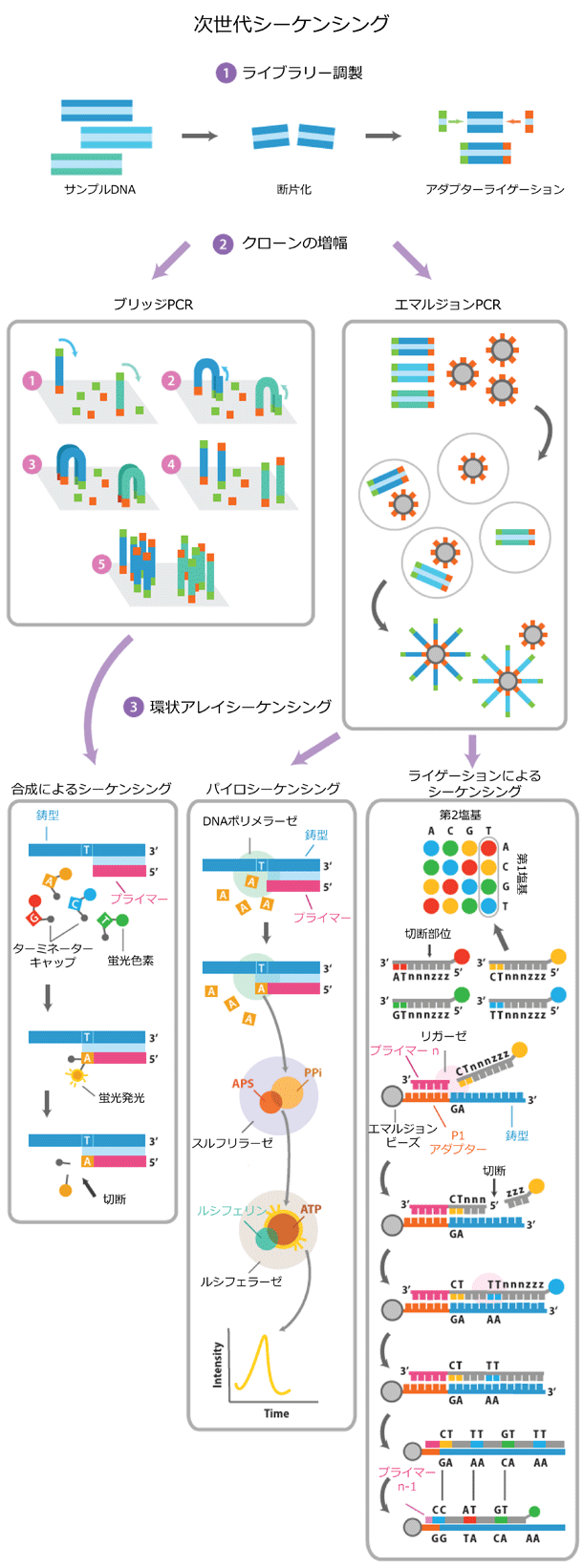
各次世代シーケンシングプラットフォームの主な相違点は、シーケンシング反応にあります。以下に、各技術の違いを簡単にまとめていますが、より詳細な情報については各セクションに記載のリンクより、各社ウェブサイトの解説をご確認ください。
• パイロシーケンシング
パイロシーケンシングでは、核酸取り込み時のピロリン酸放出を介してシーケンシング反応をモニターします。一つのヌクレオチドがシーケンシングチップに付加され、これが鋳型依存的に取り込まれます。この取り込みにより一連の化学反応で使用されたピロリン酸が放出され、光を産生、この光の放出をカメラで検出し、クラスターの該当配列を記録します。次のヌクレオチドが添加される前に、組込まれなかった塩基は全てアピラーゼにより分解されます。シーケンシング反応が完了するまでこのサイクルを継続します(表.1)。
欠点 : 試薬が高価であることおよび6つ以上連なる単一塩基ヌクレオチドにおける誤り率が高いこと。
表.1 入手可能なパイロシーケンシングを利用したNGS機器の技術概要
NGSシステム GS Junior GS Junior Plus GS FLX+ システム
GS FLX Titanium XL+ GS FLX Titanium XLR70
リード長
400bp
700bp
700bp
450bp
処理量
35Mb
70Mb
700Mb
450Mb
実行あたりのリード数
100,000 ショットガン,
100,000 ショットガン,
1,000,000 ショットガン
1,000,000 ショットガン,
精度
99% at 400bp
99% at 700bp
99.997%
99.995%
実行時間
10 時間
18時間
23時間
10時間
* 詳細は、Roche社ウェブサイト をご参照ください。
• 合成によるシーケンシング
合成によるシーケンシングでは、可逆的な蛍光と終止核酸の段階的な取り込みをDNAシーケンシングに利用します。これは、Illumina社の次世代シーケンシングプラットフォームに利用されている手法です。本手法で使用されるヌクレオチドは以下の2つの点が変更されています。
各ヌクレオチドは特有の発光波長をもつ単一の蛍光分子に可逆的に結合されている。
各ヌクレオチドはまた可逆的に終結され、1回のサイクルごとに一つのヌクレオチドのみが組込まれるようになっている。
4種全てのヌクレオチドがシーケンシングチップに添加され、ヌクレオチド取り込みの後、残りのDNA塩基は洗い流されます。各クラスターにおいて蛍光シグナルが読み取られ、その後、蛍光分子と終止基の両方が切断されて洗い流され、シーケンシング反応が完了するまでこの工程が繰返されます。このシステムでは、一度に1つのヌクレオチドを取り込ませることでパイロシーケンシングシステムの欠点を克服しています(表.2)。
欠点 : シーケンシング反応が進行するに従い、機器によるエラー率が上昇する。蛍光シグナルの取り込みや除去により、バックグラウンドノイズのレベルがより高くなるためである。
表.2 入手可能な合成によるシーケンシングを利用する次世代シーケンシング機器の技術詳細
NGSシステム MiSeq NextSeq 500 HiSeq 2500 HiSeq 3000 HiSeq 4000
実行モード
N/A
中出力
高出力
迅速な実行
高出力
N/A
N/A
実行ごとのフローセル数
1
1
1
1 or 2
1 or 2
1
1 or 2
出力範囲
0.3-15 Gb
20-39 Gb
30-120 Gb
10-300 Gb
50-1000 Gb
125-750 Gb
125-1500 Gb
実行時間
5-55 hrs
15-26 hrs
12-30 hrs
7-60 hrs
<1-6 days
<1-3.5 days
<1-3.5 days
フローセルごとのリード数
25,000,000
130,000,000
400,000,000
300,000,000
2,000,000,000
2,500,000,000
2,500,000,000
最大リード長
2 x 300bp
2 x 150bp
2 x 150bp
2 x 250bp
2 x 125bp
2 x 150bp
2 x 150bp
* 詳細は、Illumina社ウェブサイト をご参照ください。
• ライゲーションによるシーケンシング
ライゲーションによるシーケンシングはヌクレオチド取り込みにDNAポリメラーゼを利用しないことから、前述の2つの手法とは異なります。この手法では、DNAポリメラーゼの代わりに、お互いに連結する短鎖オリゴヌクレオチドプローブを利用します。これらのヌクレオチドは8塩基(3'から5')からなり、2つのプローブ特異的塩基(この2塩基位は全て異なる全16種の8-merプローブ)と6つの縮重塩基をもち、4種の蛍光染色のうち1つがプローブの5'末端に付加されています。シーケンシング反応はプライマーがアダプター配列に結合し、適切なプローブへハイブリダイゼーションすることで始まります。このプローブのハイブリダイゼーションは2つのプローブ特異的塩基に誘導され、アニール後はDNAライゲースによりプライマー配列へと連結されます。結合していないオリゴヌクレオチドは洗い流されてシグナル検出後に記録され、蛍光シグナルは切断され(最後の3塩基)、その後、次のサイクルが始まります。およそ7回のライゲーションサイクルの後、DNA鎖が変性され、先に使用したプライマーより1塩基ずれた他のシーケンシングプライマーを用いて上記のステップが繰返されます。全部で5つのシーケンシングプライマーが使用されます(表.3)。
欠点 : 本手法ではシーケンシングリードが非常に短鎖になる。
表.3 入手可能なライゲーションによるシーケンシングを利用したNGS機器の技術詳細
NGSシステム Genetic Analyzer V2.0
機器処理量 5500W System 5500xl W System
・1 x 50
80 Gb
160 Gb
・1 x 75
120 Gb
240 Gb
・2 x 50 MP
160 Gb
320 Gb
・50 x 50 PE
160 Gb
320 Gb
精度
99.99%
99.99%
実行時間
7 days
7 days
* 詳細はThermo Fisher SCENTIFIC社の ウェブサイト をご参照ください。
• イオン半導体シーケンシング
イオン半導体シーケンシングでは、シーケンシング反応中の水素イオン放出を利用してクラスター配列を検出します。各クラスターは溶液のpH変化を検出できる半導体トランジスタの直上に配置されます。ヌクレオチド取り込みの際、1つのH+ が溶液中に放出され、半導体に検出されます。シーケンシング反応自体はパイロシーケンシングと同様に進行しますが、費用はその何分の1か節約することができます(表.4)。
欠点 : ヌクレオチドの同種重合体伸展に比べ、エラー率が高い。
表.4 利用可能なイオン半導体シーケンシングを利用するNGS機器の技術詳細
NGSシステム Ion Proton System
出力
最大 10 Gb
リード
60-80,000,000 リード
リード長
最大200bp
実行時間
2-4 hrs
詳細は、Thermo Fisher SCENTIFIC社 ウェブサイトをご参照ください。
次世代シーケンシング(NGS)- 各次世代シーケンシング(NGS)プラットフォームの比較
前述のデータから異なるNGS機器間の相違を理解するのは非常に困難であることがわかります。本章では、ヒト(3,300,000,000塩基)、マウス(2,800,000,000塩基)、シロイヌナズナ(135,000,000塩基)、またはE.coli (4,639,221塩基)ゲノムの配列決定を行うと仮定し、どのように各システムが遂行するか確認することで機器間比較の単純化を試みています(表.5)。シーケンシングデータを利用するには少なくとも30回カバーすることが要求されます。被覆数がこれに満たないものは赤色 で示し、これ以上のものは青色 で示しました。
表.5 実行ごとに被覆できるゲノム
Roche社 GS Junior GS Junior Plus GS FLX+ System
GS FLX Titanium XL+ GS FLX Titanium XLR70
ヒト 0 0 0 0
マウス 0 0 0 0
シロイヌナズナ 0 1 5 3
大腸菌 8 15 151 97
Illumina社 MiSeq NextSeq 500 HiSeq 2500 HiSeq 3000 HiSeq 4000
ヒト 5 12 36 91 303 227 455
マウス 5 14 43 107 357 268 536
シロイヌナズナ 111 289 889 2,222 7,407 5,556 11,111
大腸菌 3,233 8,407 25,866 64,666 215,553 161,665 323,330
Thermo Fisher Genetic Analyzer V2.0
5500W System 5500xl W System
ヒト 48 97
マウス 57 114
シロイヌナズナ 1,185 2,370
大腸菌 34,489 68,977
Thermo Fisher SCENTIFIC社 Ion Proton System
ヒト 3
マウス 4
シロイヌナズナ 74
大腸菌 2,156
次世代シーケンシング :リード(読み取り断片) :リード長 :被覆度 :ディープシーケンシング :ペアードエンドシーケンシング :メイトペアードリード :アダプター :
固定表面へのハイブリダイゼーションを行う
増幅産物とシーケンシングプライマーの双方にプライミングする場所を提供
異なるサンプル群を1回の実行でマルチプレックスする際のバーコードとして機能
ライブラリー :アライメント :参照配列/ゲノム :新規(De Novo )アセンブリ :特異性 :均一性 :同種重合体 :
VIDEO