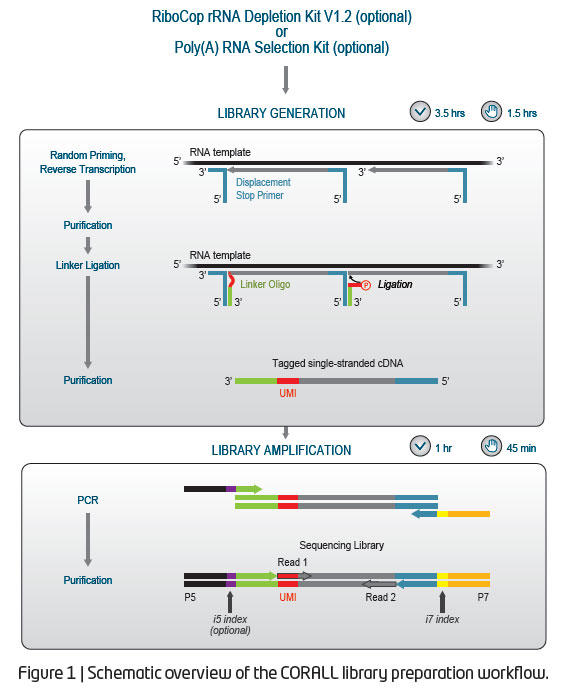
CORALL Total RNA-Seq V2 Library Prep Kitは、Lexogen社が新たに開発したTotal RNA ライブラリ調製キットで、whole transcriptomeをカバーします。CORALLはイルミナ社機器に対応するライブラリを4.5時間以内に調製可能で、Unique Molecular Identifiers (UMIs)が併用可能な点、高いストランド特異性(>99%)が特長です。Lexogen社独自のDisplacement Stop / Ligation技術を採用しており、ライブラリ調製の過程で断片化が不要で、転写開始点、転写終結点を含む完全なライブラリが得られます。
わずか1ngのtotal RNAインプットからライブラリを調製可能 CORALL RNA-Seq V2 Library Prep Kit
- カテゴリから探す > 分子生物 > DNAシーケンシング > 次世代シーケンシングキット
特長
- 転写開始点から終結点までの全長RNAを対象としたライブラリ調製
- 断片化処理が無く、ライブラリ長が安定
- 幅広いRNAインプット量に対応(0.1 ng 〜 1 µg total RNA)
- 4.5 時間以内で完了
- ストランド特異的な解析(> 99%)
- 分解サンプルやFFPE サンプルも解析可能
- 12 nt Unique Dual Indices(UDIs)とUnique Molecular Identifiers (UMIs)が付属
ワークフロー
CORALLライブラリは、わずか1ngのtotal RNAインプットから調製可能です。様々な生物種の解析に使用でき、rRNA除去RNA、poly(A)濃縮RNA、total RNA、FFPE組織由来RNA等の様々なRNAサンプルに対応しています。ライブラリ調製では、RNAの断片化は不要で、まず、イルミナ社機器対応のP7配列の一部を含むDisplacement Stop Primers (DSP) が試料RNAにランダムプライミングします。次に、逆転写反応によりDSPを次のDSP結合位置まで伸長させます。次のDSP結合位置で伸長が止まることで、誤った第二鎖合成を防ぎ、ストランド特異性を向上させます。その後、リンカーオリゴがcDNAの3’末端に結合し隙間を埋めます。
PCRステップでは、第二鎖合成が行われ、二本鎖cDNAが得られます。この過程で、イルミナ社機器でシークエンス解析する際に必要なi7インデックスやi5インデックス(オプション)を含むアダプター配列がライブラリに付加されます。精製ステップでは磁気ビーズを用いており、自動化にもおすすめです。
CORALL は最大 384 種類の pre-mixed i5 / i7 Unique Dual Indices(UDIs)と一緒に利用可能で、UDIs はインデックスエラーの修正に有用です。オプションとして、i7 インデックスを利用したシングルインデックスにてマルチプレックス化可能です。UMIs の情報は Read 1 に含まれるため、シングルリードモードで解析可能です。RiboCop rRNA 除去キ ットとのセット品もご用意しています。
性能
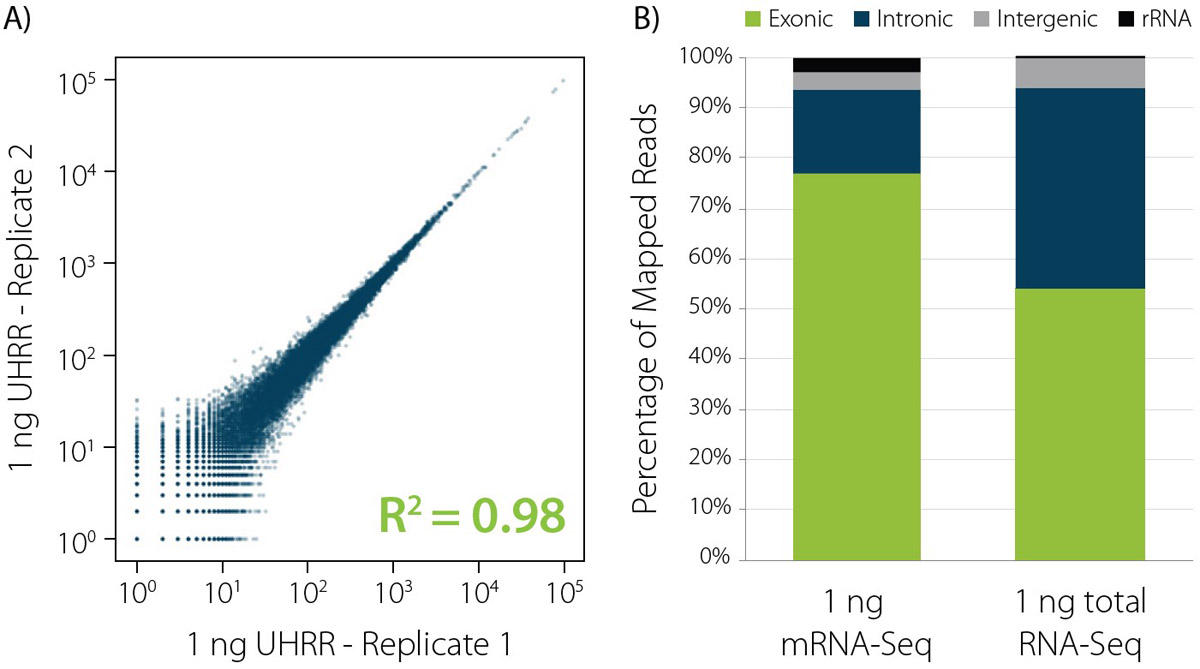
図2: 少量RNAでの優れた性能
A) 1 ngのUniversal Human Reference RNA(UHRR)サンプルをCORALL mRNA-Seq V2で2回処理した結果、非常に高い再現性が確認された。
B) CORALL mRNA-Seq V2およびCORALL Total RNA-Seq V2における1 ng input RNAのRead分布。
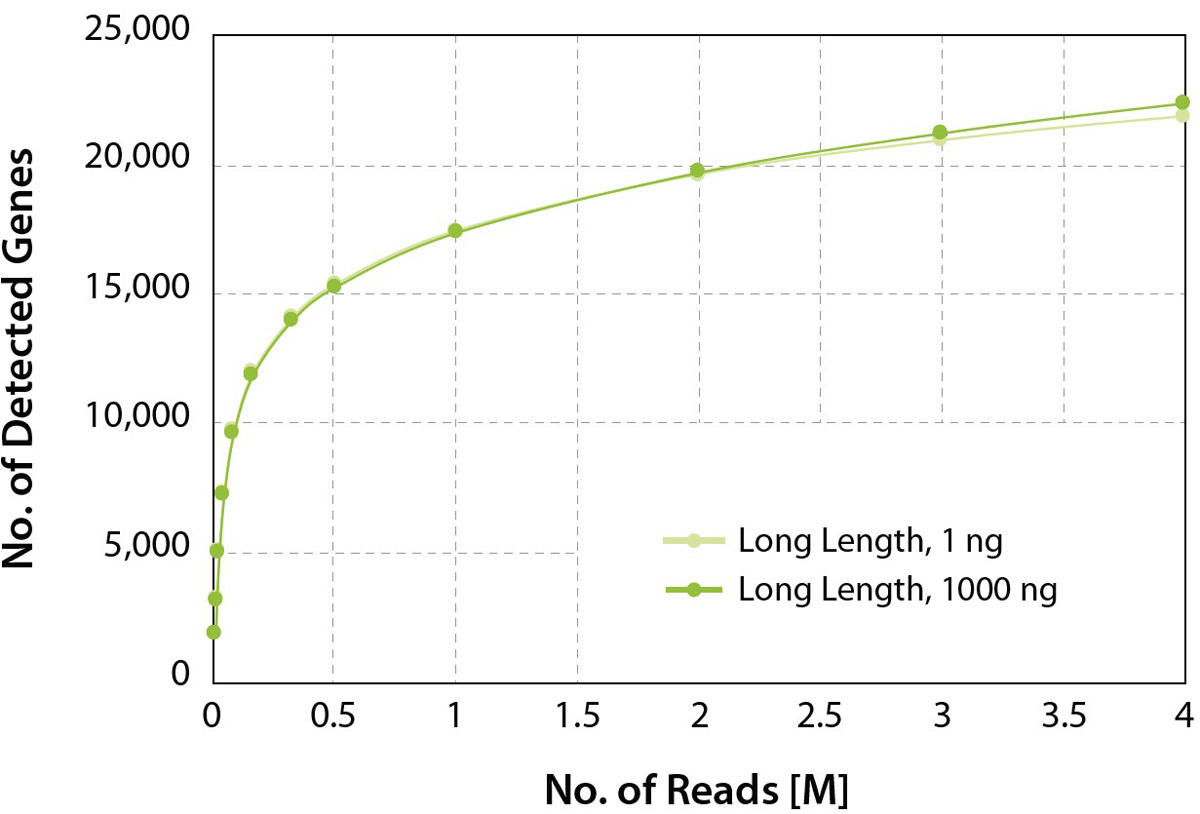
図3: 幅広いinput量に対応可能
CORALL mRNA-Seqで1 ngと1000 ngのUHRRから長いインサートを持つlibraryを作製した。検出された遺伝子の数を、ユニークにマッピングされたエクソンリードの総数に対してプロットしました。
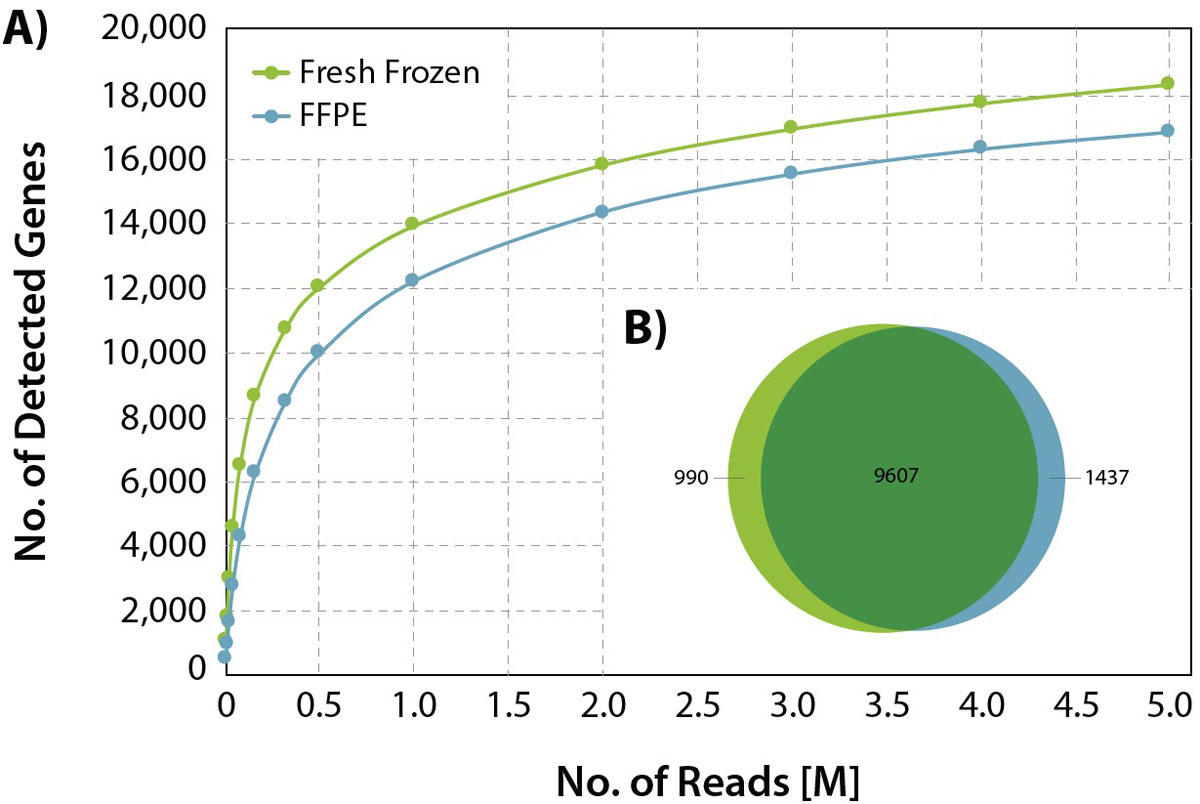
図4:FFPEサンプルに対する高いパフォーマンス
新鮮凍結(Fresh Frozen)およびFFPE由来のヒト肝臓RNAの遺伝子発見率。
A) 検出された遺伝子の数を、エクソンにユニークにマッピングされたリードの総数に対してプロットした。
B) 検出された遺伝子の重複をベン図で表した(normalized expression levels >5 CPM)
キット選択ガイド
| 特長 | CORALL | ||
| RNA-Seq | mRNA-Seq | Total RNA-Seq | |
| Input RNA量 | 0.1 ng − 100 ng | 1 ng − 1000 ng | 1 ng − 1000 ng |
| 検出対象 | Total RNA | mRNA | Total RNA (rRNA以外) |
| 品質要件 | 低品質でも使用可 (RIN値5以下の分解RNA、 FFPEサンプル) |
高品質のみ対応 (RIN値8以上) |
低品質でも使用可 (RIN値5以下の分解RNA、 FFPEサンプル) |
| 作業時間 | 4.5時間 | 5.5時間 | 6時間 |
| キットのサイズ | 24, 96, 384 | 96, 384 | 24, 96 |
| 付属品 | ー | Poly(A) RNA Selection Kit | RiboCop rRNA Depletion Kit Human/Mouse/Rat用 |
rRNAは全RNAの80〜90%を占めるため、rRNAを解析に含めない場合はmRNA-seqまたはTotal RNA-seqの使用を推奨します。
| サンプル数 | Illumina機器 | |
| Forward Strand Workflow (A) | Reverse Complement Workflow (B) | |
| HiSeq 2000/2500 HiSeq 3000/4000 (Single-Read Flow Cell) MiSeq MiniSeq (Rapid Reagent Kits) NovaSeq 6000 (v1.0 Reagent Kits) |
iSeq 100 HiSeq 3000/4000 (Paired-End Flow Cell) NextSeq 500 - 2000 MiniSeq (Standard Reagent Kit) NovaSeq 6000 (v1.5 Reagent Kits) NovaSeq X / X Plus HiSeq X |
|
| 4 - 96 | Set A1 | Set B1 |
| 97 - 384 | Set A1-A4 | Set A1-A4 |
使用する機器とサンプル数に応じて、UDIセットを選択してください。
- Dobin A., et al. (2013) STAR: ultrafast universal RNA-seq aligner.Bioinformatics, DOI:10.1093/bioinformatics/bts635
- Tuerk A., et al. (2017) Mixture models reveal multiple positional bias types in RNA-Seqdata and lead to accurate transcript concentration estimates. PLoS Computational Biology,DOI: 10.1371/journal.pcbi.1005515
- Paul, L. et al. (2016) SIRVs: Spike-In RNA Variants as External Isoform Controls in RNASequencing.bioRxiv. DOI: 10.1101/080747.
CORALL RNA-Seq V2 Library Prep Kit with UDI 12 nt
| 品名 | メーカー | 品番 | 包装 | 希望販売価格 |
|---|---|---|---|---|
CORALL RNA-Seq V2 Library Prep Kit with UDI 12 nt Set A1, (UDI12A_0001-0024) |
LEX | 171.24 | 24 PREP. |
¥286,000 |
CORALL RNA-Seq V2 Library Prep Kit with UDI 12 nt Set A1, (UDI12A_0001-0096) |
LEX | 171.96 | 96 PREP. |
お問い合わせ |
CORALL RNA-Seq V2 Library Prep Kit with UDI 12 nt Set A2, (UDI12A_0097-0192) |
LEX | 172.96 | 96 PREP. |
お問い合わせ |
CORALL RNA-Seq V2 Library Prep Kit with UDI 12 nt Set A3, (UDI12A_0193-0288) |
LEX | 173.96 | 96 PREP. |
お問い合わせ |
CORALL RNA-Seq V2 Library Prep Kit with UDI 12 nt Set A4, (UDI12A_0289-0384) |
LEX | 174.96 | 96 PREP. |
お問い合わせ |
CORALL RNA-Seq V2 Library Prep Kit with UDI 12 nt Set B1, (UDI12B_0001-0024) |
LEX | 175.24 | 24 PREP. |
¥286,000 |
CORALL RNA-Seq V2 Library Prep Kit with UDI 12 nt Set B1, (UDI12B_0001-0096) |
LEX | 175.96 | 96 PREP. |
お問い合わせ |
CORALL RNA-Seq V2 Library Prep Kit with UDI 12 nt Sets A1-A4 (UDI12A_0001-0384) |
LEX | 176.384 | 384 PREP. |
お問い合わせ |
CORALL mRNA-Seq V2 Library Prep Kit with UDI 12 nt
| 品名 | メーカー | 品番 | 包装 | 希望販売価格 |
|---|---|---|---|---|
CORALL mRNA-Seq V2 Library Prep Kit with UDI 12 nt Set A1, (UDI12A_0001-0096) |
LEX | 177.96 | 96 PREP. |
お問い合わせ |
CORALL mRNA-Seq V2 Library Prep Kit with UDI 12 nt Set A2, (UDI12A_0097-0192) |
LEX | 178.96 | 96 PREP. |
お問い合わせ |
CORALL mRNA-Seq V2 Library Prep Kit with UDI 12 nt Set A3, (UDI12A_0193-0288) |
LEX | 179.96 | 96 PREP. |
お問い合わせ |
CORALL mRNA-Seq V2 Library Prep Kit with UDI 12 nt Set A4, (UDI12A_0289-0384) |
LEX | 180.96 | 96 PREP. |
お問い合わせ |
CORALL mRNA-Seq V2 Library Prep Kit with UDI 12 nt Set B1, (UDI12B_0001-0096) |
LEX | 181.96 | 96 PREP. |
お問い合わせ |
CORALL mRNA-Seq V2 Library Prep Kit with UDI 12 nt Sets A1-A4 (UDI12A_0001-0384) |
LEX | 182.384 | 384 PREP. |
お問い合わせ |
RiboCop (HMR) and CORALL Total RNA-Seq V2 Library Prep Kit with UDI 12 nt
| 品名 | メーカー | 品番 | 包装 | 希望販売価格 |
|---|---|---|---|---|
RiboCop (HMR) and CORALL Total RNA-Seq V2 Library Prep Kit with UDI 12 nt Set A1, (UDI12A_0001-0024) |
LEX | 183.24 | 24 PREP. |
お問い合わせ |
RiboCop (HMR) and CORALL Total RNA-Seq V2 Library Prep Kit with UDI 12 nt Set A1, (UDI12A_0001-0096) |
LEX | 183.96 | 96 PREP. |
お問い合わせ |
RiboCop (HMR) and CORALL Total RNA-Seq V2 Library Prep Kit with UDI 12 nt Set B1, (UDI12B_0001-0024) |
LEX | 184.24 | 24 PREP. |
お問い合わせ |
RiboCop (HMR) and CORALL Total RNA-Seq V2 Library Prep Kit with UDI 12 nt Set B1, (UDI12B_0001-0096) |
LEX | 184.96 | 96 PREP. |
お問い合わせ |
RiboCop (HMR+Globin) and CORALL Total RNA-Seq V2 Library Prep Kit with UDI 12 nt
Total RNA-seqにグロビンmRNA除去試薬が付属したキットです。Human/Mouse/Ratの全血からグロビンmRNAを90%以上除去し、rRNA除去のみの場合と比較してシーケンス深度を約 2 〜 4 倍節約できます。
商品は「研究用試薬」です。人や動物の医療用・臨床診断用・食品用としては使用しないように、十分ご注意ください。
※ 表示価格について
- 「CORALL RNA-Seq V2 Library Prep Kit」は、下記のカテゴリーに属しています。
-
- カテゴリから探す > 分子生物 > DNAシーケンシング > 次世代シーケンシングキット























 このページを印刷する
このページを印刷する













