Lexogen社 QuantSeq 3' mRNA-Seq Library Prep Kit (FWD) のパフォーマンス
- カテゴリから探す > 分子生物 > DNAシーケンシング > DNAシーケンシングキット
- カテゴリから探す > 分子生物 > miRNA > 次世代シーケンシング
- カテゴリから探す > 分子生物 > DNAシーケンシング > 次世代シーケンシングキット
パフォーマンス
■ ストランド特異的な3' 末端マッピング
FDA Sequencing Quality Control(SEQC)standard sample A およびB はERCC external RNA control ExFold Mixes 1 および2 をそれぞれ添加したリファレンスRNA です。1, 2 これらのサンプルからQuantSeq ライブラリを作製し、Association of Biomolecular Resource Facilities(ABRF)より報告されているmRNA-Seq データセット3 と比較しました。従来のmRNA-Seq では転写産物の全長に渡りシーケンス解析を行いますが、QuantSeq では主に3'末端付近を解析します(図1)。そのため、QuantSeq では遺伝子発現解析の高い正確性を保ちながら、シーケンス深度を90% 以上節約することが可能です。スパイクインRNAにはセンス鎖のみ含まれますので、ゲノムアノテーションとは独立してストランド特異性を評価することが可能です。2つのmRNA-Seq SEQC データセットではストランド特異性がそれぞれ93.4%、97.8% でしたが3、QuantSeq のストランド特異性は> 99.9% を示しました。このようにノイズを下げることにより、アンチセンス転写産物を正確に検出、定量することが可能です。
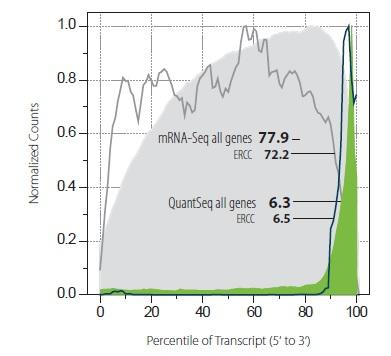
図.1 カバレッジ vs. 標準化した転写物長
RSeQC より取得したカバレッジをプロットした。転写産物は面で、ERCC mix は線で、QuantSeq は色付きで、mRNA-Seq は灰色で表した。数字は曲線下面積を示し、シーケンスカバレッジの目安である。
■ QuantSeq の定量性
遺伝子カウントの正確性を評価するために、ERCC spike-in 転写産物の平均リードカウントをインプット分子数に対してプロットしました(図2)。線形モデルならびにSpearmanの相関係数で評価されるように、遺伝子発現解析において、非常に高いinput-output 相関と正確性が見られました。
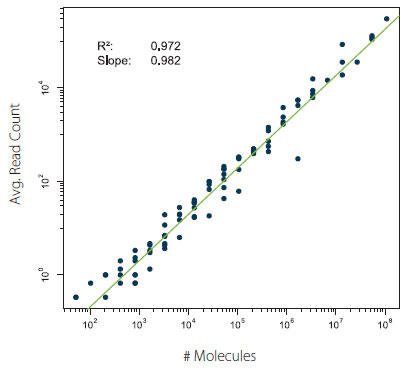
図.2 QuantSeq を用いた遺伝子発現解析において、ERCC のリードカウントはインプットと非常に高い相関を示した。
■ 差次的遺伝子発現
QuantSeq と従来のmRNA-Seq を、“erccdashboard”ソフトウェアを使用した差次的遺伝子発現の検出能の点で比較しました4。リード数を10 M から0.625 M へダウンサンプルした場合、従来のmRNA-Seq では曲線下面積(AUR)の値は0.736 〜 0.776 と低い一方、QuantSeq では0.860〜 0.897 と非常に高い値を維持しました(図3)
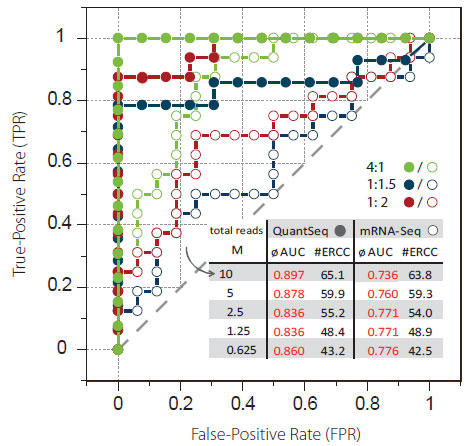
図.3 QuantSeq と従来のmRNA-Seq の差次的遺伝子発現検出能の比較
ERCC ExFold Spike-In Mix 1 および2 の比率を事前に検討し、陽性率(TPRs)と偽陽性率(FPRs)を評価した。曲線下面積(AUC)1 を最適な検出とする。曲線下面積(AUR)は検出されたERCC RNA 数(#ERCC)と併せて評価される。
FFPE 組織から抽出したRNA を使用する場合のパフォーマンス
■ QuantSeq では、高クオリティRNA と低クオリティRNA を使用した場合で収量に高い相関が見られる。
RNA 分解を受けている可能性があるサンプル(例:ホルマリン固定パラフィン包埋切片; FFPE)について、同じ由来かつRNA クオリティの異なる2 種類のサンプルを使用し、QuantSeq の有用性を評価しました。
MOLP-8 ヒト骨髄腫細胞株の異種移植片を2 片に分割し、新鮮凍結組織ブロックおよびFFPEブロックを作製し、これらをRNA クオリティの異なる試料としました。RNA クオリティの評価には、Agilent 社のRIN(RNA Integrity Number)がよく用いられており、RIN 8 以上で高いクオリティのRNAであることを示します。分解が進んでいるサンプルではRINの精度は低く、RIN よりもDV200(200 ヌクレオチド以上のRNA 断片の割合)の値が利用されます。DV200 の値が低いと、RNA の完全性が低いことを表します。
抽出したRNA について、RNA のクオリティを評価したとこ ろ、FFPE サンプルのDV200 は87%(RIN:2.8)、凍結組織のRIN は8.3 でした。その後、QuantSeq 3' mRNA-Seq FWD キットを使用して、50 ng RNA インプットにてライブラリを作製しました。QuantSeq Kit では、DV200 が52%のFFPE 組織からもライブラリを作製することができました(data not shown)。FFPE 組織については低クオリティRNA 用の推奨プロトコルに準じて、凍結組織についてはスタンダードなプロトコルに準じて操作を行いました。作製したライブラリについて、HiSeq 2500 を用いて、1 × 50 bpリード長でシーケンス解析を行いました(図4)
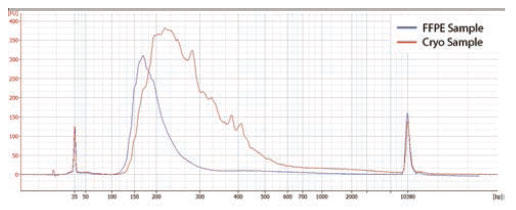
図.4 QuantSeq 3' mRNA-Seq FWD Kit を用いて作製したライブラリについて、バイオアナライザー2100 HS でDNA を定量した。FFPE 組織を青色、凍結組織を赤色で示す。
分解が進んだ試料RNA から作製したライブラリでは、滑らかな分布となり、リンカー配列により生じる副産物のピークが見られなかった。また、DNA 断片長のピークは短鎖側にシフトした。平均ライブラリサイズは204 bp(FFPE 組織)、286 bp(凍結組織)であった。
標準化した転写産物長に対してカバレッジをプロットしたところ、使用したRNA のクオリティに関わらず、カバレッジは転写物の3'末端側に集中することが示されました(図5)。しかしながら、QuantSeq FWD ライブラリはpoly(A) 鎖に向かって得られますので、カバレッジはライブラリサイズやシーケンシングの長さに依存します。FFPE 組織から作製したライブラリは、凍結組織から調製したライブラリと比較し非常に短いため、シーケンス解析の際に転写産物末端に到達する頻度が高くなります。このことはカバレッジのプロットに影響すると考えられます。
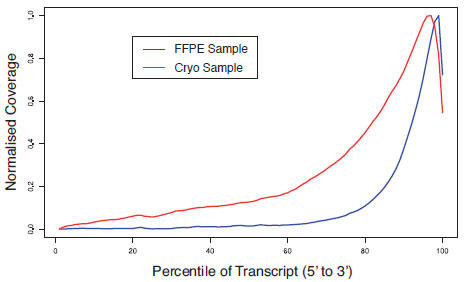
図.5 QuantSeq のリードカバレッジと転写産物長の相関性
FFPE 組織(青色)、凍結組織(赤色) より抽出した試料RNA を用いてQuantSeq ライブラリを作製し比較した。
FFPE 組織から抽出したRNA と凍結組織から抽出したRNAを用いた場合では遺伝子発現解析の相関性は高い結果となり(R2 0.86)、QuantSeq は様々なクオリティのRNA で一貫した結果が得られることが示されました(図6)

図.6 FFPE 組織、凍結組織での遺伝子カウントの相関
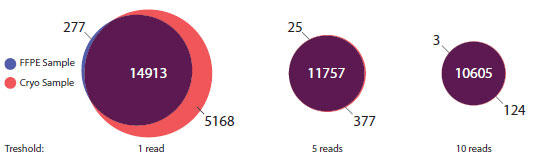
図.7 FFPE 組織、凍結組織を用いてQuantSeq 解析し(リード数2.5 M)、遺伝子発現をベン図に表した。
QuantSeq は、凍結した試料RNA の場合、リード数26.5M で25,842 遺伝子検出可能です(data not shown)。リード数を一律2.5 M にした場合、凍結組織では少なくとも1リードで20,081 遺伝子、FFPE 組織では15,190 遺伝子が検出され、検出遺伝子数に24% の差が見られました(図7)。しかしながら、検出レベルを5 リードまたは10 リードに増やすと、サンプル間の差は3% または1% にまで減少しました。これは、FFPE 組織では、低発現量の遺伝子はFFPE サンプルの調製、保存、RNA 抽出等の過程で分解を受けやすいこと、QuantSeq では凍結組織、FFPE 組織、両者において正確に遺伝子を検出していることを示しています。
QuantSeq は、逆転写の過程でoligo(dT) プライミングを用いており、1 転写産物あたり1 分子の断片を生成するため、RNA のクオリティに依存することなく、正確な遺伝子発現解析が可能です。スタンダードなmRNA-Seq のプロトコルでは転写産物全体をカバーしますが、分解が進んだRNA をサンプルとして用いると3' 末端側にバイアスのかかった結果となる可能性があります。従って、QuantSeq 3' mRNASeqは、他のmRNA-Seq プロトコルと比較し、低いクオリティのRNA サンプルから効率よく次世代シーケンス解析用ライブラリを作製する非常に効果的なツールと言えます。
血液RNA 用Globin Block モジュール
■ QuantSeq ライブラリとの併用
疾患関連の研究において、血液は情報量が多く、利用しやすい検体です。Globin mRNA(HBA1、HBA2、HBB) は血液中のtotal RNA のうち50-80% を占めるため、シーケンスリードの大部分をマスクしてしまい、遺伝子の検出感度や定量感度を下げてしまいます。従来のGlobin 除去法では、試料RNA の前処理が必要なため、多量のRNA が必要で、追加コストも必要です。Lexogen 社のGlobin Block Module はQuantSeq ライブラリを作製する際に使用することでGlobin 除去が可能です。少ないインプット量(血液由来total RNA 50 ng 程)に対応しており、その他の前処理は不要です。
Globin Block Module はQuantSeq 3' mRNA-Seq ライブラリ作製キット(イルミナ社シーケンサー用)と併用できるようデザインされています。QuantSeq ライブラリを作製する際、スタンダードなRNA Removal Solution(RS)の代わりにmodifi ed RNA Removal Solution(RS-Globin Block)を用いてGlobin Blocker を導入します。Globin Blocker はGlobin の第一鎖cDNA に結合し、第二鎖合成時に二本鎖DNA 生成を阻害します。Globin Block は自動化システムにも対応しており、ヒトおよびブタサンプル用を用意しています。
■ Globin Block はGlobin にマップされるリードを0.7% まで減少させる
Globin Block を使用してライブラリを作製した場合、Globin Block を使用しなかった場合と比較し、Globin にマ ップされたリードの割合が顕著に減少しました(図8)。Globin にマップされたリードの割合は、白血球リッチの血液の場合0.7% まで、全血+Globin Block の場合9.7% まで減少しました。
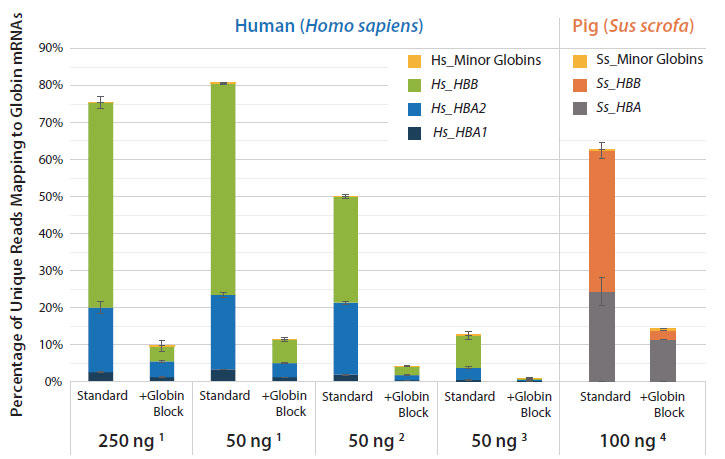
図.8 ヒトおよびブタのGlobin mRNA にマップされたリードの割合
全血より抽出したRNA を用いて、QuantSeq FWD プロトコルでライブラリを作製した群と、QuantSeq +Globin Block でライブラリを作製した群で比較した。
1.Lexogen 社SPLIT RNA Extraction Kit(赤血球の溶解なし)
2.他社Qキット(赤血球の溶解あり)
3.Lexogen 社SPLIT RNA Extraction Kit(赤血球の溶解あり)
4.他社Nキット(赤血球の溶解なし)
■ 遺伝子検出率の向上
図9 では、スタンダードなQuantSeq ライブラリとGlobin Block を併用して作製したライブラリ間でユニークに検出された遺伝子と重複して検出された遺伝子を表しています。しきい値0.5 CPM(counts per million)では、大部分の遺伝子が同じように検出されましたが、Globin Blockを併用して作製したライブラリのみで3,690 種類の遺伝子がユニークに検出されました。対照的に、スタンダードなQuantSeq ライブラリのみで検出された転写産物はわずか14、17、136 種類でした。
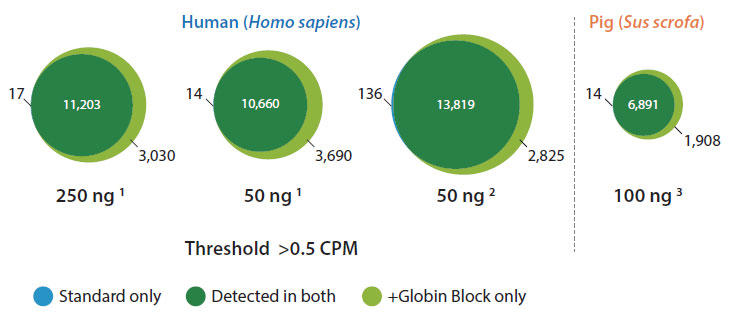
図.9 ヒトおよびブタの血液サンプルにおいて、Globin Block の使用で遺伝子検出率が向上する
全血より抽出したRNA を用いて、QuantSeq FWD プロトコルでライブラリを作製した群と、QuantSeq +Globin Block でライブラリを作製した群で比較した。検出された遺伝子数はCounts Per Million(CPM)で標準化したリードカウントから算出した(しきい値> 0.5)。重複して検出された遺伝子は深緑色で示し、ユニークに検出された遺伝子は青色(スタンダードなQuantSeq ライブラリ)、黄緑色(QuantSeq +Globin Block ライブラリ)で示した。
1.Lexogen 社SPLIT RNA Extraction Kit(赤血球の溶解なし)
2.他社Qキット(赤血球の溶解あり)
3.Lexogen 社SPLIT RNA Extraction Kit(赤血球の溶解あり)
4.他社Nキット(赤血球の溶解なし)
■BlueBee® データ解析
Lexogen 社の BlueBee® データ解析パイプラインでは、 QuantSeq ライブラリについて、アラインメント、遺伝子カウント、発現差異解析が可能です。様々な生物種の解析に対応しています。QuantSeq 3' mRNA-Seq Library Prep Kitの箱に、キットの reaction 数に応じた Bluebee® 用のアクチベーションコードを表示しています。追加解析用のコードが必要な場合は、コードのみお求めいただけます。また、 QuantSeq のデータ解析は Partek® Flow® や ROSALIND™ でも実施可能です。
- The External RNA Controls Consortium. (2005). The External RNA Controls Consortium: a progress report. Nature Methods 2:731-734
- Ambion. ERCC RNA Spike-In Control Mixes. Cat. No. 4456740, 4456739
- Li, S. et al. (2014). Multi-platform assessment of transcriptome pro ling using RNA-seq in the ABRF next-generation sequencing study. Nat. Biotechnol. 32, 915?925
- Munro, S.A. et al. (2014). Assessing technical performance in differential gene expression experiments with external spike-in RNA control ratio mixtures. Nat. Commun. 5, 5125
商品は「研究用試薬」です。人や動物の医療用・臨床診断用・食品用としては使用しないように、十分ご注意ください。
※ 表示価格について
- 「Lexogen社 QuantSeq 3' mRNA-Seq Library Prep Kit (FWD) のパフォーマンス」は、下記のカテゴリーに属しています。
-
- カテゴリから探す > 分子生物 > DNAシーケンシング > DNAシーケンシングキット
- カテゴリから探す > 分子生物 > miRNA > 次世代シーケンシング
- カテゴリから探す > 分子生物 > DNAシーケンシング > 次世代シーケンシングキット























 このページを印刷する
このページを印刷する













