【商品詳細】
- 【01】 Version 2で新しくなった点について教えてください。
- 【02】 TeloPrime Full-Length cDNA Amplification Kit V2のインプットRNAの必要量はどれくらいですか。
- 【03】 TeloPrime Full-Length cDNA Amplification Kit V2に低品質のRNAをインプットすることは可能ですか?
- 【04】 5'末端および3'末端はどのようにタグ付けされるのでしょうか。
- 【05】 完全長の二本鎖DNA合成にはどれくらいの時間がかかりますか?
- 【06】 逆転写やPCRでカスタムプライマーを使用することは可能ですか?
- 【07】 なぜqPCRを行う必要があるのですか?qPCRはスキップしてもいいですか?
- 【08】 エンドポイントPCRのサイクル数はどのように決定すればよいですか?
- 【09】 qPCRアッセイができない場合はどのようにして最適なPCRサイクル数を決定すればよいですか?
- 【10】 PCR増幅ではどのような配列が使われていますか?
- 【11】 TeloPrime Full-Length cDNA Amplification Kit V2は異なるタイプのRNAに対してどのように機能しますか?
- 【12】 NGSへのアプリケーションにマルチプレックスTeloPrime cDNAのサンプルバーコードは使用可能ですか?
- 【13】 完全長のcDNAはどのようなアプリケーションに使用可能ですか?
- 【14】 TeloPromeキットで調製したcDNAはどのNGSライブラリ調製キットと互換性がありますか?
- 【15】 TeloPrime Full-Length cDNA Amplification Kit V2はQuantSeqやCORALL Total RNA/mRNA-Seq Library Prep Kitと互換性はありますか?
- 【16】 ロングリードシークエンス用の完全長cDNAを調製するためにはTeloPrimeキットをどのように使用すればいいですか?
- 【17】 Iso-Seq™で使用するにはTeloPrime で調製したcDNAのサイズセレクションを行う必要がありますか?
- 【18】 TeloPrime で調製したcDNAライブラリのIso-Seq?のデータはどのように解析したらよいですか?
- 【19】 TeloPrimeのVersion 1は未だ購入できますか?
第二鎖合成用およびPCR用のバッファーと酵素、プロトコールを変更しています。この変更では増幅した完全長cDNAの長さと収率を改善することを目的としています。具体的には、下記のコンポーネントが改良されています。
- Second Strand Mix (SS)
- Enzyme Mix 3 (E3)
- TeloPrime PCR Mix (Telo PCR)
- DNA Buffer
Lexogen社ではTeloPrimeのプロトコールに関して、Universal Human Reference RNA(Agilent Technologies社)はもちろん、マウスの肝臓および腎臓、脳、肺、脾臓、胸腺、心臓から抽出したRNAや、Human Brain Reference RNAを用いて、1 ng~2 µgのtotal RNA インプット量で検証しています。また、増幅したcDNAの収率と長さを最大限にするためには、品質の高いtotal RNAをご使用いただくことを推奨しております。
ロングリードシークエンスを行う際は、1~2 µgのtotal RNAインプットを推奨しております。詳細についてはUser Guide![]() の「7. Appendix A: RNA Requirements」をご参照ください。
の「7. Appendix A: RNA Requirements」をご参照ください。
可能ですが、お勧めはできません。当該キットでのcDNA調製には、できる限り品質の良いRNAをインプットRNAとしてご使用いただくことを推奨しております。低品質RNA(RIN値<8)もインプットRNAとして使用可能ですが、RNAの完全性が低いと、インタクトな5'キャップを持つ転写物の割合が減少してしまいます。
TeloPrimeは低品質のtotal RNAサンプル中に存在するインタクトな5'キャップおよびポリアデニル化されたmRNAから完全長のcDNAを合成することが可能ですが、増幅したcDNAの長さは、高品質のインプットRNAから調製されたcDNAに比べて短くなる可能性がある点にご注意ください。
それでもなお、インプットRNAに低品質のRNAをご使用される場合、qPCRアッセイを使用して最適なエンドポイントPCRのサイクル数を決定してください(詳細についてはQ【08】およびUser Guide![]() の「8. Appendix B: Calculation of Optimal Endpoint PCR Cycle Number」をご参照ください)。
の「8. Appendix B: Calculation of Optimal Endpoint PCR Cycle Number」をご参照ください)。
5'末端へのタグ付けは、Lexogen社独自のcap 依存的リンカーライゲーション(Cap Dependent Linker Ligation ;CDLL)技術に基づいています。逆転写後およびRNAとcDNAの二本鎖合成後に5' C オーバーハングを有する二本鎖アダプターを、RNA/cDNA ハイブリッド(二本鎖)のcap 構造のG と結合させます。二本鎖特異的なリガーゼを用いることで、cap 構造を持ち(インタクトなRNA)、第一鎖合成がmRNA の5' 側に到達した場合のみライゲーションが起こります。また、mRNAの3'末端のタグ付けは、oligo(dT)プライマーでの逆転写中に行われます。
完全長のcDNA合成には、全体で約5時間、そのうち実際の操作時間はたったの60分です。V2キットを使用したTeloProme cDNAの増幅におけるエンドポイントPCRでは、全体で5~8時間(PCRのサイクル数によります)かかりますが、実際の操作時間はわずか25分です。また、エンドポイントPCRをする前にqPCRを行っていただくことを推奨しておりますが、qPCRはおよそ14時間かかるため、overnightで実行いただくことを推奨しております。
はい、カスタムプライマーをご使用いただけます。逆転写プライマー、およびForwardプライマーとReverseプライマーは個別に追加されるため、置き換えることも可能です。カスタムプライマーのご使用についての詳細はUser Guide![]() の「10. Appendix D: Downstream Applications」をご参照ください。
の「10. Appendix D: Downstream Applications」をご参照ください。
Lexogen社は、最適なエンドポイントPCRのサイクル数を決定するために、qPCRを行っていただくことを推奨しております。qPCRは下流のアプリケーションに影響を及ぼし得るアンダーサイクルやオーバーサイクルを防ぎます。アンダーサイクルは低収率を引き起こし、下流のアプリケーションで不十分なサンプルとなってしまいます。一方でオーバーサイクルはより長い完全長cDNAが得られにくくなります(図1参照)。一度、特定のRNAサンプルタイプやインプット量でのエンドポイントPCRのサイクル数を決定すれば、毎回qPCRを行う必要は無く、同じ値を以降の実験におけるqPCRに適用することができます。詳細についてはUser Guide![]() の「8. Appendix B: Calculation of Optimal Endpoint PCR Cycle Number」をご参照ください。
の「8. Appendix B: Calculation of Optimal Endpoint PCR Cycle Number」をご参照ください。
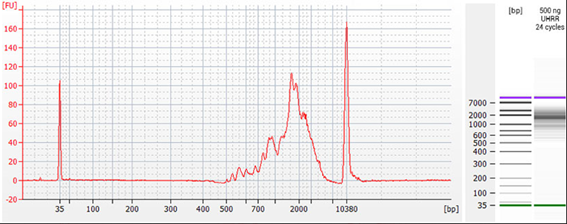
Universal Human Reference RNA(UHRR)500 ngを用いてTeloPrime cDNAを調製し、その後、1 µLのcDNAを用いてPCRサイクル数24で増幅した。1 µL得られたcDNAを10倍希釈し、Bioanalyzer Sensitivity DNA Chip(Agilent Technologies)で分析した。最適なエンドポイントPCRのサイクル数はUser Guide
qPCRアッセイでは、反応液にSYBR Green I色素を終濃度10%で添加し、リアルタイムPCR装置を用いて増幅させます。バンドの蛍光が最大値である80%の値に達するサイクル数を決定してください。エンドポイントPCRに同様のcDNAを使用する場合、エンドポイントPCRには同様のサイクル数を適用できます。エンドポイントPCRでqPCRよりも多くのcDNAを使用したい場合、サイクル数は調整する必要があります。例えば、qPCRアッセイに2 µLのcDNAを使用し、9 µLがエンドポイントPCRに使用される場合、最終的にcDNA増幅の為にテンプレートとして約4倍のcDNAを使用することを考慮して、2サイクル少なくする必要があります。詳細についてはUser Guide![]() の「8. Appendix B: Calculation of Optimal Endpoint PCR Cycle Number」をご参照ください。
の「8. Appendix B: Calculation of Optimal Endpoint PCR Cycle Number」をご参照ください。
qPCRアッセイは、PCRによってエンドポイントcDNAの増幅に最適なサイクル数を決定するための最速の方法です。最終的なライブラリの複雑さを軽減しないようにライブラリをオーバーサイクルさせないことが重要です。もしqPCRが実行できない場合(qPCRの為の装置が無い、またはSYBR Green I 色素が入手できない等)は、代わりにエンドポイントテストを実行してください。その場合、追加のPCRと精製が必要なため、TeloPrime PCR Add-on Kit V2(品番:018.16)と追加の精製試薬(Purification Module with Magnetic Beads 品番:022.96、またはPacBio社の1x Ampure PB® Beads)が必要です。ステップ29で得られた精製されたcDNAを各反応で1~2 µL用いて、ある範囲のサイクル数(例:14~20サイクル)のエンドポイントPCRを複数並行して行います。さらに詳細な評価をするためには、PCR産物を精製し、バイオアナライザーでcDNAの一部を用いて、サイズ分布プロファイルおよび収量をご確認ください。
TeloPrime Kitに含まれているPCRプライマーの配列はUser Guide![]() の「10. Appendix D: Downstream Applications」に下記のように記載されています。
の「10. Appendix D: Downstream Applications」に下記のように記載されています。
FP: 5'−TGGATTGATATGTAATACGACTCACTATAG−3'
RP: 5'−TCTCAGGCGTTTTTTTTTTTTTTTTTT−3'
TeloPrime Full-Length cDNA Amplification Kit V2のプロトコールは、植物(図2)やヒツジなどの様々な脊椎動物(図3)、Universal Human Reference RNA(UHRR)(図4)などの様々な種由来のRNAで検証されています。cDNAの長さ分布はインプットRNA の種類によって異なります。下記の図2~4の各サンプルは異なる種類のBioanalyzer 2100 DNA Chip Assaysで実行されたことにご注意ください。
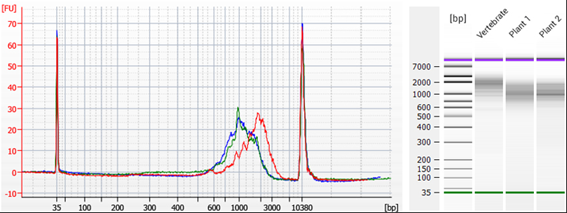
図2: 脊椎動物由来RNAおよび植物由来RNAからTeloPrime Full-Length cDNA Amplification Kit V2を用いて調製されたTeloPime cDNAのバイオアナライザートレース
TeloPime cDNAはカスタム逆転写プライマーを用いて1 µLのtotal RNAから調製した。cDNAはカスタムReverseプライマーと標準のForwardプライマーが使用され、エンドポイントPCRのサイクル数は17サイクル(脊椎動物サンプル:赤)または21サイクル(植物サンプル1:青、植物サンプル2:緑)で増幅した。PCR産物はPacBio社のAMPure® PB beadsで精製され、Agilent Technologies社のバイオアナライザー(High Sensitivity DNA Chip)での分析のために濃度300 pg/µLに揃えた。
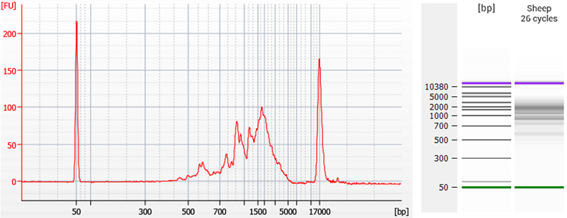
図3: ヒツジ由来total RNAからTeloPrime Full-Length cDNA Amplification Kit V2のPCRで調製されたTeloPrime cDNAのバイオアナライザートレース
TeloPrime cDNAは標準のTeloPrimeプライマーを用いて1 µLのtotal RNAから調製し、標準のTeloPrime Full-Length cDNA Amplification Kit V2のPCRプライマーで増幅した。1 µLのcDNAをエンドポイントPCRで26サイクルで増幅した。PCR産物をTeloPrime purification solutionおよびカラムで精製し、Agilent Technologies社のバイオアナライザー(DNA 12000 Chip)で分析した。
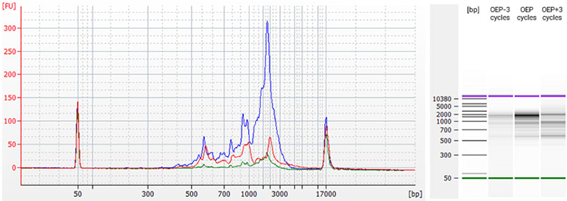
図4: 異なるPCRサイクル数で生成されたTeloPrime cDNAのバイオアナライザートレース
エンドポイントPCRは最適なエンドポイントPCRのサイクル数(最大蛍光が80%となったqPCRから算出された)または、最適なサイクル数±3で2 µLのTeloPrime cDNAを用いて行った。増幅したcDNAはAgilent Technologies社のバイオアナライザー(DNA 12000 Chip)で分析した。アンダーサイクル(最適サイクル数−3:緑)は低い収率を示し、一方でオーバーサイクル(最適サイクル数+3:赤)は比較的短い生成物が多くなり、収率も低下する結果となった。最適なサイクル数(青)はより長いサイズのcDNAに対して最も高収率だった。
サンプルへのバーコードの付加は二通りあり、カスタム逆転写プライマー(User Guide![]() の「10. Appendix D: Downstream Applications」を参照)を使った逆転写中に行うものと、完全長のcDNA増幅が完了した後にバーコードを添加する方法があります。サンプルマルチプレックスはロングリードシークエンスプラットフォームに対応しています。
の「10. Appendix D: Downstream Applications」を参照)を使った逆転写中に行うものと、完全長のcDNA増幅が完了した後にバーコードを添加する方法があります。サンプルマルチプレックスはロングリードシークエンスプラットフォームに対応しています。
PacBio IsoSeq
サンプルバーコードはalternative oligodT-primerに含めて、第一鎖cDNA合成(step1~3)用の逆転写プライマー(Reverse Transcription Primer;RTP)の代わりにご使用ください。IsoSeqライブラリにはPacBio社が提供しているsample barcoded RT primer デザインをご使用いただくことを推奨しております。(PacBio社のIsoSeqのプロトコールはこちら![]() 。)
。)
これらのプライマーは脱塩化オリゴとして注文できます。逆転写反応におけるこれらのオリゴの濃度は、12.5 mM~1.5 µMの間で最適化する必要があり得ます。さらには、エンドポイントPCRに標準のFPプライマーと一緒に使用するためにカスタムRPプライマーが必要です。このカスタムRPプライマーはバーコード付加された逆転写プライマーの5'末端に結合する必要があり、melting temperature(Tm)を一致させるか、またはTeloPrime Full-Length cDNA Amplification Kit V2に含まれる標準のプライマーのTmにできるだけ近づける必要があります。
Oxford Nanopore
Oxford Nanopore Technologies社のNanopore PCR Barcoding Kit(品番:SQK-PBK 004)を用いてエンドポイントPCRを行った後、サンプルバーコードをTeloPrimeで合成したcDNAに付加することができます。Oxford Nanopore Technologies(ONT)社のバーコード付加に対応するONTアダプターを使用するには、TeloPrime Full-Length cDNA Amplification Kit V2のエンドポイントPCRに使用するRPプライマーとFPプライマーをONTプライマーへの結合配列を含むように改変する必要があります。
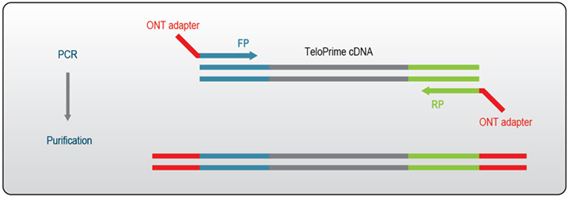
図5: ONTシークエンスにおけるTeloPrime cDNAへのONTアダプター付加の模式図
alternative FPプライマーおよびalternative RPプライマーの配列は下記の通りです。これらは脱塩化オリゴとしてオーダー出来、2 µMの最終濃度でエンドポイントPCRに使用してください。
FP-ONT:5'-TTTCTGTTGGTGCTGATATTGCTGGATTGATATGTAATACGACTCACTATAG -3
RP-ONT: 5'- ACTTGCCTGTCGCTCTATCTTCTCTCAGGCGTTTTTTTTTTTTTTTTTT -3
エンドポイントPCRのプログラムにはアニーリング温度50℃で1サイクル、残りのサイクルには62℃のアニーリング温度を使用してください。
完全長のcDNA生成物はshort read(Illumina機器対応)やlong read(PacBio社やOxfordNanopore Technologies社機器対応)のNGSライブラリ調製や、RACE法、クローニング、マイクロアレイプローブへの応用が可能です。
市販されている一般的なDNA-Seqライブラリ調製キットを使用してTeloPrime Full-Length cDNA Amplification Kit V2で調製し、増幅させたcDNAからシークエンスライブラリを調製可能です。
【15】TeloPrime Full-Length cDNA Amplification Kit V2はQuantSeqやCORALL Total RNA/mRNA-Seq Library Prep Kitと互換性はありますか?
いいえ、TeloPrime Full-Length cDNA Amplification Kit V2は最終生成物として完全長の二本鎖cDNAを生成するため、インプット材料としてRNAのみを使用するQuantSeqおよびCORALLキットとの互換性はありません。
TeloPrimeキットはPacBio社およびOxford Nanopore Technologies社のlong-read シークエンス解析プラットフォームと互換性があります。
TeloPrime のcDNAライブラリはPacBio Sequel®機器でシークエンスに成功しています。詳細についてはこちらからTeloPrimeの引用文献をご確認ください。
Iso-Seq™
TeloPrime で調製したcDNAを用いて、PacBio Sequel®でのシークエンス用にIso-Seq™ライブラリを作製する場合、追加のアダプターをcDNAの末端に添加する必要があります。このアプリケーションには下記を推奨しております。
- TeloPrime でcDNAを調製する際は1~2 µLのtotal RNAをインプットしてください。
- 可能な限り品質の高いRNAを使用してください。
- PacBio社が推奨しているプロトコールにのっとって推奨されている精製ビーズを用いて、エンドポイントPCRで増幅したcDNAを精製してください。
また、TeloPrimeライブラリはMinIONでもシークエンス可能で、最近の文献で紹介されています。
Oxford Nanopore Technologies
TeloPrime で調製したcDNAをOxford Nanopore Technologies 社の機器でシークエンスを行いたい場合は、まずcDNAの末端にアダプターシークエンスを追加する必要があります。Oxford Nanopore Technologies社のDNAライブラリ調製キット、PCRバーコード付加キットを使用してください。お客様のアプリケーションに適したキットに関する詳細についてはOxford Nanopore Technologies社にお問い合わせいただけますようお願いいたします。
サイズセレクションはIso-Seq™ライブラリ調製においてより長いcDNAの割合を高めるのに有効です。Iso-Seq™のためのサイズセレクションは任意で行ってください。サイズセレクションを行いたい場合はご使用されるライブラリ調製キットに従って、PCR後のすべての精製ステップにPacBio社のAmpure® PB Beadsを使用してください。TeloPrimeにおけるエンドポイントPCRにおいては、サイズセレクションおよび下流のライブラリ調製用の十分なインプット量を確保するために下記のように一部プロトコールを修正してください。
PacBio社のIso-Seq™ライブラリ調製ではサイズセレクションは任意のオプションです。サイズセレクションはシークエンスでより深い深度を得るためにより長い転写物の割合を高めることができますが、必須ではありません。TeloPrime Full-Length cDNA Amplification Kit V2で調製されたcDNAを用いたライブラリはサイズセレクションの有無に関わらず、問題なくシークエンス可能です。サイズセレクションを行う場合は、PacBio社のIso-Seq™のプロトコール![]() をご確認ください。
をご確認ください。
また、以下の場合にはサイズセレクションは必要ございません。
- 植物RNA
転写物の平均長が短いことがあります。お客様からは植物サンプルではサイズセレクションは必要ないとのフィードバックをいただいております。 - インプットRNAの品質がRIN 8以下
RIN 8以下のサンプルではインプットRNAは既に分解されてしまっているため、長い完全長cDNAは生成されません。 - もしノーマルな転写産物の存在量が保たれている場合
サイズセレクションは長いcDNAと短いcDNAの相対的な存在量を変えるので存在量が保たれているのであれば、必要ありません。
サイズセレクションを行う場合は以下の注意事項をご確認ください。
- TeloPrime cDNAを増幅する際に、ある量の(例として2~4 µL)のcDNAを鋳型として、エンドポイントPCRを実施ししサイクル数を調整してください。同じモル濃度で各サイズのcDNAをプールするため、サイズセレクションにはより高いcDNA収率が必要です。
PCRサイクルがqPCRアッセイを行うことで最適化される場合、それぞれのPCRは600 ng以上のcDNAを生成します。 - PCR産物をプールして全容量をmolecular biology gradeの水で160 µLに調整してください。
- プールから100 µL分取し、PacBio社の Iso-Seq™のプロトコールに従って0.4 xまたは0.5×Ampure® PBビーズで精製してください。
- プールから60 µL分取し、PacBio社の Iso-Seq™プロトコールに従って1×Ampure® PBビーズで精製してください。
- 各分画でQCを行ってPacBio社のプロトコールに従って同じモル濃度でcDNAを再プールして、SMRT bell ligationを続行してください。
PacBio社のシークエンス装置によって得たシークエンスデータはPac Bio社のIso-Seqパイプライン![]() と、Modularアルゴリズムのトランスクリプトームアノテーション(Iso-Seq™のデータ用)、またはTeloPrime でのロングリード解析を実行するために特別に改良されたTAMAソフトウェアパッケージ (https://github.com/GenomeRIK/tama) 組み合わせてご使用いただくと最適な分析が可能です。TAMAはPython 2.7で実行でき、インストールする必要はありません。TAMAは定期的に更新されるため最適のワークフローと追加事項についてはGitHubリポジトリを定期的にご確認いただけますようお願いいたします。
と、Modularアルゴリズムのトランスクリプトームアノテーション(Iso-Seq™のデータ用)、またはTeloPrime でのロングリード解析を実行するために特別に改良されたTAMAソフトウェアパッケージ (https://github.com/GenomeRIK/tama) 組み合わせてご使用いただくと最適な分析が可能です。TAMAはPython 2.7で実行でき、インストールする必要はありません。TAMAは定期的に更新されるため最適のワークフローと追加事項についてはGitHubリポジトリを定期的にご確認いただけますようお願いいたします。
下記のパイプラインに従ってTAMAでロングリード解析していただくことをお勧めしております
- CCS
- LIMA 注意:「isoseq 3 refine-require-polya」は実行しないようにしてください。
- bamtools convert-format fasta-in unpolished.flnc.bam>flnc.fasta
- ポリAクリーンナップ(実行形式:python tama_flnc_polya_cleanup.py{fasta}{outfile})
注意:現在のバージョンではA以外のランダム挿入は処理されません) - Minimap2 to the genome
- Sort bam file and convert to sam
- Run TAMA collapse






















