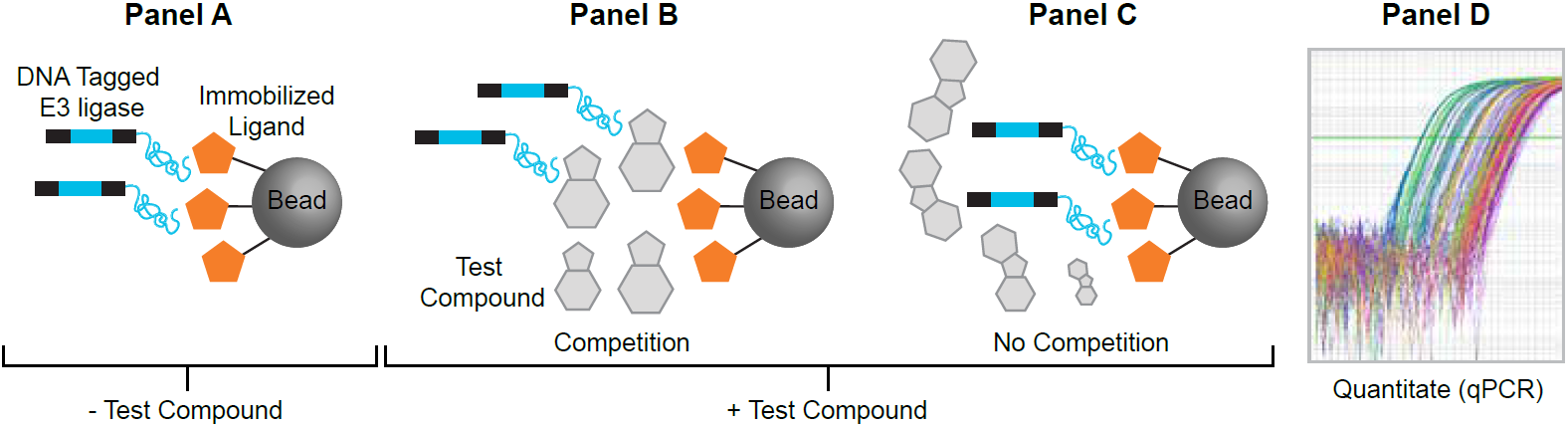
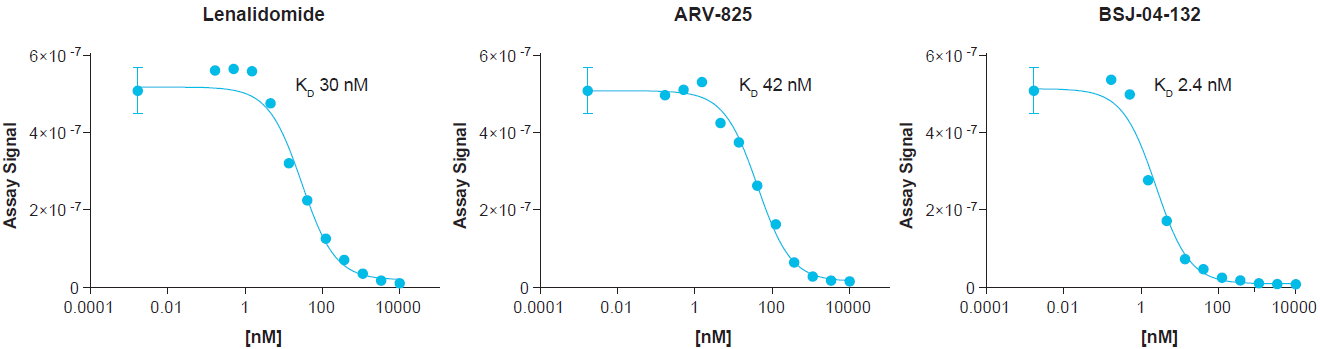
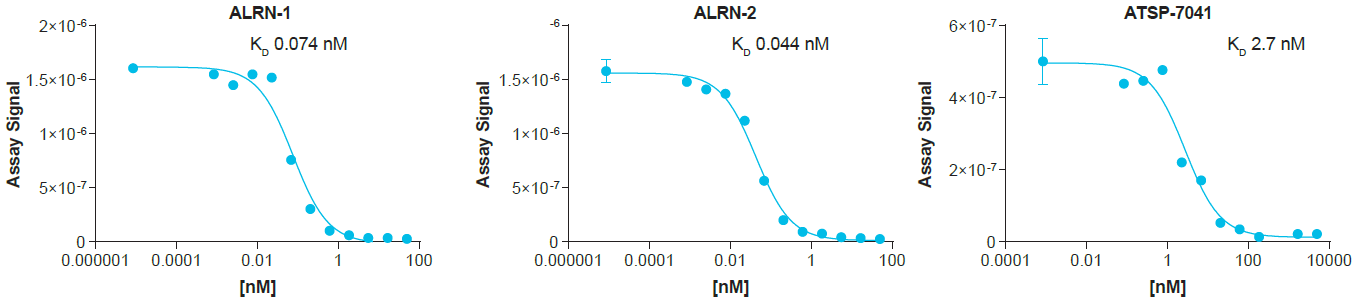
【特長】
- ご希望のタンパク質の分解検出用細胞株を構築
- 標的タンパク質は過剰発現せずアッセイが可能(Fig.4, Fig.5)
- HTSにも対応可能な簡便なアッセイ系
- 既存のPROTACの標的タンパク質でのアプリケーション実績(Fig.6)
【原理】
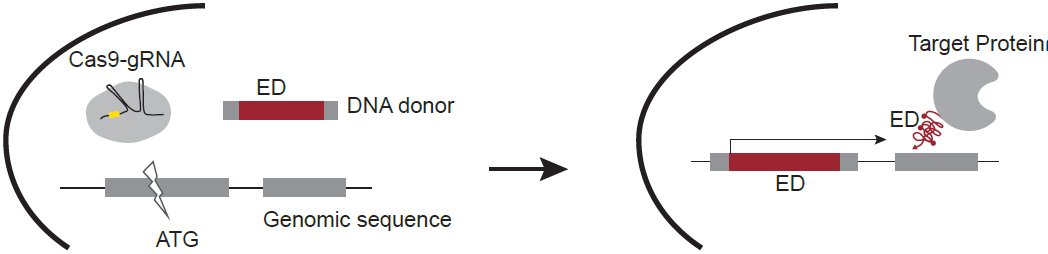
Fig.4 アッセイ用細胞構築の流れ
本サービスでは、CRISPR-Cas9を使用して、標的タンパク質にメーカー独自のEDタグを付加するようゲノム編集を行い、アッセイ用細胞株を構築する。
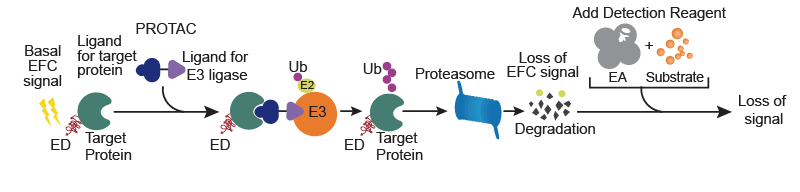
Fig.5 標的タンパク質の分解の検出原理
標的タンパク質が分解されていない定常条件下では、EDタグを介した標的タンパク質の検出系(EFC)によりシグナルが得られる。PROTACを加えることで標的タンパク質がE3 ligaseにリクルートされ、ユビキチン-プロテアソーム系を介して分解されるため、EDタグを介したシグナルが低下する。
【データ例】
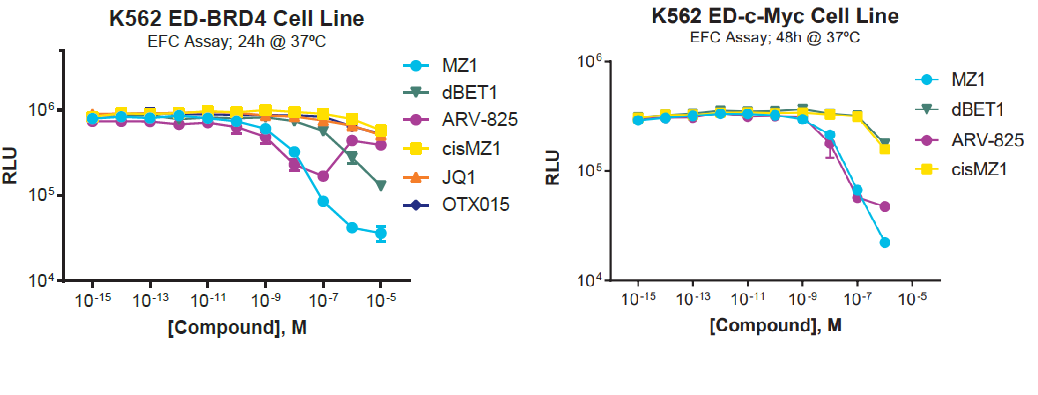
Fig.6 BRD4と下流のc-Mycの分解アッセイデータ
(左図)BRD4に対するPROTAC(MZ1, dBET1, ARV-825)はBRD4の分解を誘導したが、BRD4阻害剤(JQ1, OTX015)は分解誘導しなかった。
(右図)BRD4に対するPROTACの一部(MZ1, ARV-825)は、長時間incubationすることでBRD4の下流のc-Mycの量も減少させた。























 このページを印刷する
このページを印刷する