RNA誘導型のゲノム編集最新ツールであるCRISPR-Cas9システムでは、特異性の高いゲノム編集が可能です。この画期的な技術は、使用方法が簡単で、かつ如何なるゲノム座位も20塩基対のRNAオリゴをデザインすることで標的できる、という点で遺伝子工学を改革したといえます。gRNA(ガイドRNA、sgRNA)は、CRISPR-Cas9システムにおいて重要な構成要素であり、その配列のデザインには数々の点を考慮する必要があります。
技術情報
特異性の高いゲノム編集の鍵、ガイドRNA設計方法を解説!CRISPR-Cas9 - gRNAデザイン
CRISPR-Cas9 - crRNA、tracrRNA、およびgRNA
細菌や古細菌では、CRISPR RNA(crRNA)とトランス活性化型CRISPR RNA(tracrRNA)が複合体を形成し、侵入してきた外来の遺伝物質にCas9ヌクレアーゼを配向させる帰巣装置として機能します(詳細はCRISPR-Cas9 基本の「き」をご参照ください)。tracrRNAとcrRNAを組み合わせて単一の合成gRNAとすることで、実験デザインを簡易化できるだけでなく、同等またはより高いガイド効果が得られます(図.1)(1)。3'末端に異なるtracrRNA配列長をもつ様々な構造のgRNAがデザインされ、試行されました。全体のgRNAデザイン内に占める本来のtracrRNA配列部分が増大するごとに編集効率の上昇がみられました(2)。最も一般的に使用されるgRNAは100塩基長です。gRNAの5'末端側の20塩基対を変更することで、CRISPR-Cas9システムによりgRNAの相補配列をもつ如何なるゲノム領域をも標的することができます。
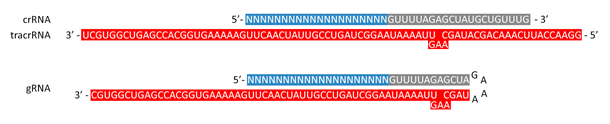
図.1 crRNA-tracrRNA複合体とgRNAの例
*Applied Biological Materials社のCRISPR sgRNAベクターは、レンチウイルスとアデノウイルス型式のいずれにおいても最大限の編集効率が得られるようデザインされています。
CRISPR-Cas9 - 一般的なgRNAデザインガイド
CRISPR-Cas9システムの標的特異性は、gRNAの5'末端に位置する20塩基配列により決定します。S. pyogenes CRISPR-Cas9システムでは、期待する標的配列は5'-NGGプロトスペーサー隣接モチーフ(PAM、詳細はCRISPR-Cas9 基本の「き」をご参照ください)の直前に配置される必要があります。gRNAは、Cas9ヌクレアーゼを ”相補的な塩基対を形成できる標的配列” に先導し、Cas9ヌクレアーゼはPAM配列から〜3塩基程度上流において二本鎖切断を媒介します(図.2)。PAM配列はgRNA配列の20塩基対に含まれないものの、CRISPR-Cas9ゲノム編集においてゲノムDNA上にこの配列が存在することは必須条件です。
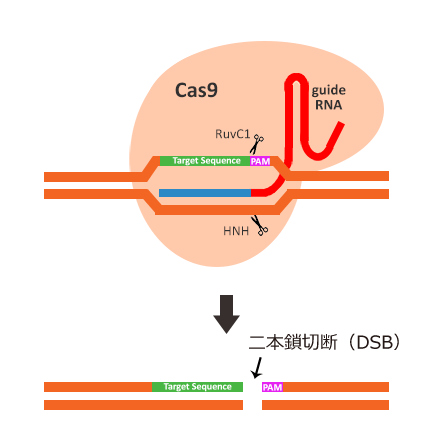
図.2 S. pyogenes システムでは、標的配列は5'-NGG PAM配列の直前に隣接している必要がある。gRNAは標的配列の反対鎖と相補的塩基対を形成し、Cas9ヌクレアーゼを介してPAM配列の〜3塩基上流に二本鎖切断を生ずる反応を媒介する。
いくつかのプロモーターは標的部位選択とgRNAデザインを制限するものの、gRNA(ガイドRNA、sgRNA)発現の駆動に適切なプロモーターを選択することは非常に重要です。例えば、RNAポリメラーゼIII依存型U6プロモーター、またはT7プロモーターは、転写開始のためにRNA配列の5'末端に「G」または「GG」がそれぞれ必要です(2)。したがって、U6またはT7プロモーターをS. pyogenes CRISPRシステムに使用する場合、標的配列はそれぞれ「GN16-19NGG」または「GGN15-18NGG」に限定されます。あるいは、20塩基対の先導配列の5'末端に余分なGまたはGGを付加しておくことでこの制限を回避することもできます(図.3)。
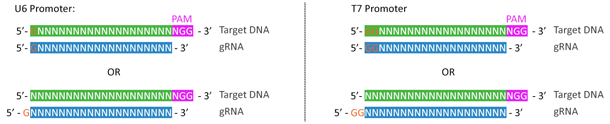
図.3 U6やT7 RNAポリメラーゼIIIプロモータをgRNA発現に使用することで生ずる配列制限は、回避することが可能
gRNA(ガイドRNA、sgRNA)の20塩基対先導配列をデザインする際は、以下の3点を考慮する必要があります。
- GC含量:一般的なGC含量は40%〜80%。GC含量が高いほどRNA:DNA二量体を安定化させて、オフターゲットハイブリダイゼーションを不安定化する。
- 鎖長:鎖長は調整することができ、その範囲は17 - 24塩基対である。より短鎖の配列はオフターゲット効果を最小化する。
- デザインしたgRNA(ガイドRNA、sgRNA)によりオフターゲットとなりうる部位。
gRNA(ガイドRNA、sgRNA)と標的部位間のミスマッチ許容性は、CRISPR-Cas9システムのオフターゲット作用を誘発することになり、一般的にその頻度は以下の項目に依存します。
- ガイド配列の5'末端におけるミスマッチは、最初の8〜4塩基対と比較して、より許容性を示す。
- gRNA(ガイドRNA、sgRNA)と標的配列間に、3塩基以上のミスマッチがある場合、オフターゲット作用を阻害する。
- オフターゲット作用はgRNA配列にも依存し、gRNA(ガイドRNA、sgRNA)によってはミスマッチに対して低い許容性を示す。
- 細胞内に導入されたCas9とgRNAの量や比率もオフターゲット作用に影響を及ぼすため、Cas9とgRNAを慎重に滴定することでこれらの効果を低減できる(3)。
非相同性末端結合(NHEJ)を利用して小さな挿入欠損を導入する場合、如何なるタンパク質にも数々の標的可能部位が存在します。タンパク質コード領域のN末端側を標的する方がC末端側を標的するより望ましく、これは多くのタンパク質が翻訳されていない段階でフレームシフトが生ずる方が、より有害となり得るためです。また、挿入欠損変異体を構築する場合は、ゲノムDNAのコード鎖と非コード鎖のいずれも標的とすることができ、いずれに対しても同等に効果的です。もし、ゲノム編集に相同性配向型修復(HDR)を使用する場合、標的配列選択は挿入したい領域に限定されることになります。gRNA(ガイドRNA、sgRNA)をデザインする際には、必要性に応じて挙げられる様々な要求事項を注意深く考慮する必要があります。gRNA(ガイドRNA、sgRNA)を慎重に設計することで、より高い編集効率とオフターゲット変異の低減が可能となります。
* Applied Biological Materials社では、実験にすぐにご利用頂けるsgRNAベクターあるいはウイルス粒子をヒト、マウス、ラット遺伝子を網羅してご提供しています。
CRISPR-Cas9 - ダブルニッキング用の配列設計において特に考慮すること
1対のgRNAペアとニッカーゼ改変型Cas9による二本鎖切断(ダブルニッキング法)を利用する場合には、上述の内容に加えて、さらに以下の内容を考慮する必要があります(図.4)。
- 理論的にはニッキングで5'末端オーバーハングが産生される必要がある。
- 2種のgRNAの間隔は種々異なり、お互いが直接隣接する場合もあれば最大20塩基対離れる場合もある。
- 2種のgRNAは標的配列に対してそれぞれが反対鎖側になるようデザインする必要がある。PAM配列が標的配列の直前に来ることに注意(4)。
2種のgRNAをデザイン後、これらを1:1の比率で混合し、ニッカーゼ改変型Cas9(D10A変異型Cas9)と共に標的細胞へと送達します。送達手法の詳細は、CRISPR-Cas9 - 実験計画を立てる!をご参照ください。
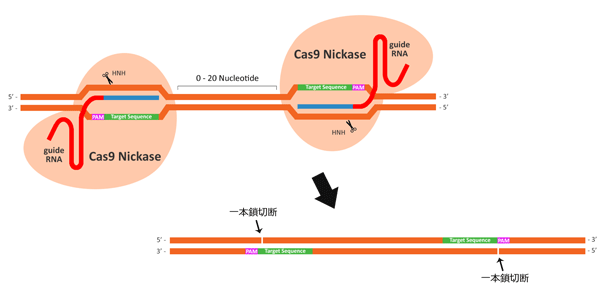
図.4 ダブルニッキング法を利用する場合、gRNAデザインについてさらに考慮する必要がある。例えば、5'オーバーハングを産生すること、2種のgRNAの間隔、および2種のgRNA標的部位の相対的な位置などである。
*Applied Biological Materials社では、ニッカーゼ改変型Cas9と組み合わせて使用するsgRNAペアをゲノムワイドにご提供しています。
CRISPR-Cas9 - gRNAデザインツール
現在、gRNAデザインに役立つツールが種々公開されています。本稿では、Chop Chop Harvard と CRISPR Designの2種類のデザインツールをご紹介します。
- Chop Chop Harvard
特定の核酸配列または対象遺伝子のアクセッション番号をChop Chop Harvard(図.5)に入力すると、ソフトウェアが配列を分析し、PAM配列を直後にもった(5'-NGG)20塩基配列候補を全て確認します。その後、GC含量やオフターゲット部位といった予め設定した基準に応じてgRNA(ガイドRNA、sgRNA)に評価(点数)をつけ、点数の高いものから低いものへと並び替えを行います。特定のgRNA配列について詳細情報を確認したい場合は、そのgRNA配列をクリックします。新規ページが開き、特定gRNAによりオフターゲット作用の可能性がある部位、これらのオフターゲット配列により生ずる変異をSURVEYORアッセイによりスクリーニングするためのプライマー配列が示されます。SURVAYORアッセイの詳細は、CRISPR-Cas9 ノックアウト実験と機能検証の手順をご参照ください。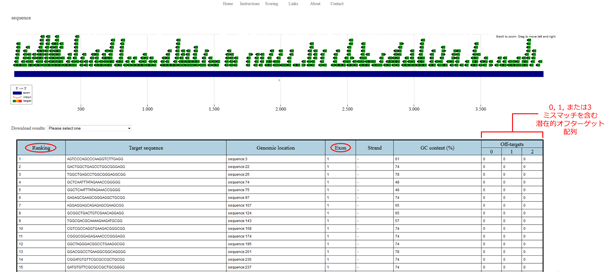
図.5 順位、エキソン位置、潜在的オフターゲット部位数に焦点をあてたChop Chop Harvard 結果ページ - CRISPRデザイン
CRISPRデザインを使用する主な利点は、全てのgRNA(ガイドRNA、sgRNA)候補におけるオフターゲット状況の詳細情報が得られることです。全てのgRNA配列をBLASTにかけ、そのオフターゲット位置、およびミスマッチ数をgRNA配列とともに詳細にレポートします。このソフトウェアは、ダブルニッキング法に使用する2種のgRNAペアをデザインする場合にも有用で、お互いに近接位置に配置された2種のgRNAを自動的にデザインします。しかし、本ソフトウェアには一度に500塩基ずつしか解析できないといった欠点もあります。したがって、gRNA配列候補の選択にはChop Chop Harvard を使用し、さらなる分析にCRISPRデザインツールを使用することがよいかもしれません。
- RNA-Guided Human Genome Engineering via Cas9. Mali, Prashant, et al. 6121, January 3, 2013, Science, Vol. 339, pp. 823-826.
- CRISPR-Cas system for editing, regulating and targeting genomes. Sander, Jeffry D and Joung, J Keith. 4, April 2014, Nature Biotechnology, Vol. 32, pp. 347-355.
- DNA targeting specificity of RNA-guided Cas9 nucleases. Hsu, Patrick D, et al. July 21, 2013, Nature Biotechnology, Vol. 31, pp. 827-832.
- Genome engineering using the CRISPR-Cas9 system. Ran, F Ann, et al. 11, October 24, 2013, Nature Protocols, Vol. 8, pp. 2281-2308.
商品は「研究用試薬」です。人や動物の医療用・臨床診断用・食品用としては
使用しないように、十分ご注意ください。


























