多機能性の細胞外サイトカインであるTGF-βは、細胞周期進行を停止して細胞増殖を抑制しアポトーシスを誘導する。一方、いくつかのがん細胞においては転位反応や血管新生を促進する。これには生長抑制機能耐性が必要であることから、このシグナル伝達経路に関与する遺伝子の変異による不活化と、TGF-βのアポトーシス機能崩壊が示唆される。TGF-βシグナル伝達は比較的よく研究されていることからCellecta社のshRNAスクリーニングライブラリーの機能評価に適したモデルシステムといえる。
技術情報
Cellecta社 ライブラリー 技術情報
背景
概要
Cellecta社では、転写因子、細胞シグナル伝達および薬物標的遺伝子を始めとする8,000種の遺伝子を標的とした25,000種のshRNAを含むレンチウイルスライブラリーを構築した。このライブラリーをヒト肝がん細胞Hep3Bに導入してスクリーニング実験を行った。さらに、2種類の他の研究所で構築されたライブラリー(例:50,000種のヒト遺伝子を標的とした200,000種のshRNAを含むライブラリーなど)を使用して同様の実験を行った。本研究では、モデル細胞にライブラリーをトランスダクションした後、多くの細胞において細胞死を誘導するTGF-βを添加して培養を行った。生存細胞に発現するshRNAはTGF-β誘導性アポトーシスを阻害する可能性が考えられ、TGF-βシグナル伝達を標的し下方制御するのに必要な遺伝子を標的していると推測される。
実験手法
・プール型25K shRNAライブラリー構築
各8,000遺伝子群に対して3〜4種のバリデート済みshRNAを選出し、25,000種のバーコード付きshRNAオリゴを合成した。プールしたオリゴをCellecta社pRSI-U6Tet-CMV-TetRep-2A-Puroベクターにクローニングし、Tet誘導型のプール型25K shRNAレンチウイルスベクターライブラリーを構築した。本ライブラリーにおけるクローニング効率および実際の構築数はバーコードを使用したHTシークエンシングにより評価し、95%以上のshRNAが少なくとも各10個以上ライブラリーに含まれることを確認した。任意に抽出したクローンをシークエンシングした結果、変異度は0.2%未満であった。
・スクリーニング
2セットの250,000 Hep3B細胞(ライブラリーに含まれるコンストラクト数の約100倍の細胞を使用)を用意し、25K shRNAライブラリーをMOI=0.5にてそれぞれトランスダクションした。この細胞群を低密度で播種した後、TGF-β (1 ng/ml)処理を3週間行った。生存細胞群よりゲノムDNAを抽出して各shRNAに対応するバーコードを増幅した後、Illumina-Solexa Genome Analyzerを用いて次世代シークエンシングを行った。スクリーニングとは別件で細胞に導入できたshRNA数を評価するため、ライブラリートランスダクション直後のTGF-β処理していない細胞よりゲノムDNAを抽出し、shRNAの同定を行った。
他の研究所で構築された2種類のライブラリー(一方は、25,000種のshRNAコンストラクトを含むと謳ったライブラリーではあるがシークエンシングによるshRNA確認が行われていないもの。他方は、50,000ヒト遺伝子を標的とした200,000種のshRNAコンストラクトを含むと謳ったもの)を使用して同様のスクリーニングを2セットずつ行った。25,000 shRNAライブラリーに対しては、従来法のシークエンシングを行って生存細胞群に含まれるshRNAを同定した。200,000 shRNAライブラリーに対してはAffymetrix U133+2 arrayにハイブリダイズしてshRNA同定を行った。
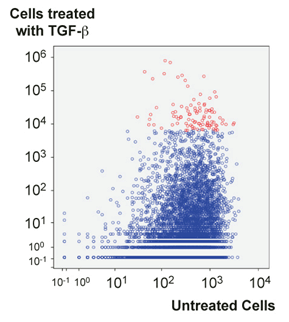
Hep3B細胞を使用したTGF-βスクリーニングにおけるshRNA分布
トランスダクション直後またはTGF-β処理後の細胞群よりゲノムDNAを抽出し、shRNAバーコードを増幅して次世代シークエンシングにより配列決定した。各処理群における各shRNAの存在比を図示した(x軸:トランスダクション直後、y軸:TGF-β処理後)。特定shRNAが導入された細胞においてもTGF-β処理が細胞増殖率に変化を与えない場合、非処理群と処理群間におけるshRNA存在比は基本的に同等となり、これらのshRNAプロットは対角線上に出現するはずである。もしTGF-β処理により増殖速度の減少や細胞死滅がみられる場合、生存細胞群中における特定shRNA分子数は処理群において低下し、これらのshRNAプロットは対角線より下方またはベースラインに出現すると考えられる。したがって、対角線より上方に出現するshRNAは、TGF-β処理においても生存し生長し続けた細胞より回収されたものであり、これらの細胞はTGF-β阻害を受けないか生長刺激を受けたかの何れかである。そのため、これらshRNAの標的遺伝子はTGF-β応答に重要な役割を担うと考えられる。
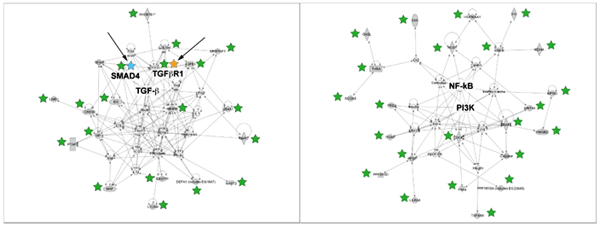
RNAiスクリーニング実験により同定されたTGF-β誘導型アポトーシス耐性遺伝子
TGF-βおよびPI3K/NFkBネットワークにおいて既知のタンパク質相互作用を模式図に示した(Ingenuity Pathway Analysis softwareを使用して作成)。緑色の星で示した通り、Cellecta社が構築した8,000の転写因子、細胞シグナル伝達および薬物標的遺伝子を標的とした25K shRNAレンチウイルスライブラリーを用いたスクリーニング実験により、この情報伝達経路に関与する41遺伝子が同定できた。
結果
TGF-βシグナル情報伝達は比較的よく研究されているため、本スクリーニングにより同定されることが期待できる遺伝子群が存在するため、スクリーニングが良好に行われたかの判断基準とすることが可能である。実際、本スクリーニングではTGF-βシグナル伝達経路に関与する既知遺伝子(SMAD4、I型およびII型TGF-βレセプター、WNT5Aなど)が高頻度で同定できただけでなく、PI3K/NFkB経路に関与する多くの遺伝子が同定できた(下のテーブル参照)。この他にも、caspase 1といった一般的に知られるアポトーシス遺伝子が複数同定できた。
他の研究所で構築された2種類のライブラリーにおいては3遺伝子が同定されたのみであった。25,000 shRNAライブラリーからはTGF-βレセプターI型のみが同定され、200,000 shRNAライブラリーではTGF-βシグナル経路に関与する転写因子SMAD4とTGF-βとの関与が報告されていないPLCB4が同定されたのみであった。興味深いことに、200,000 shRNAライブラリーを用いた場合では、TGF-βレセプターが挙がってこなかった。
本検討により、Cellecta社25K shRNAライブラリーを用いたスクリーニングでは100種以上のTGF-β関連遺伝子または新規遺伝子が同定できた。このうち、80%以上の遺伝子は2セット行った繰返し実験の何れからも同定された。2セットともから同定された19遺伝子では、shRNA発現コンストラクトを再構築して再度実験を行った。このうち18遺伝子においては、これら遺伝子のノックダウンによりTGF-β誘導性アポプトーシス耐性が獲得できた(下のテーブル参照)。
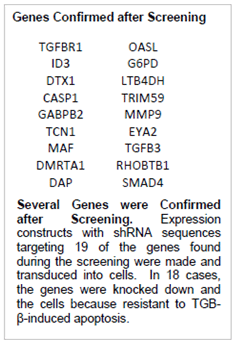
スクリーニングで同定できた遺伝子の一例
本スクリーニングにより複数の遺伝子が同定できた。スクリーニングで同定された19種の遺伝子を標的するshRNA配列をもつ発現ベクターを構築し、細胞にトランスダクションした。18遺伝子において遺伝子ノックダウンが再現でき、細胞はTGF-β誘導性アポトーシス耐性を示した。
結論
本検討より、良好にデザインされたプール型ライブラリーを用い、熟考されたRNAiスクリーニング実験を行うことで、本稿で示したTGF-β誘導型アポトーシスを始めとした特定の生物学的応答に関与する機能遺伝学的要求の網羅的解析が可能となることが示唆された。Cellecta社25K shRNAライブラリーと他の研究所により構築されたライブラリーでは結果に顕著な差がみられたことから、プール型ライブラリーを構築する際のデザインが非常に重要であると考えられる。Cellecta社ライブラリースクリーニングにより数多くの遺伝子が同定できた理由として、最適化されたshRNAデザイン、次世代シークエンシングを利用したライブラリーに含まれるshRNA存在量の確認、shRNA同定にバーコード配列および次世代シークエンシングの利用、さらにセレクションアッセイ手法の熟考などが挙げられる。


























