概要
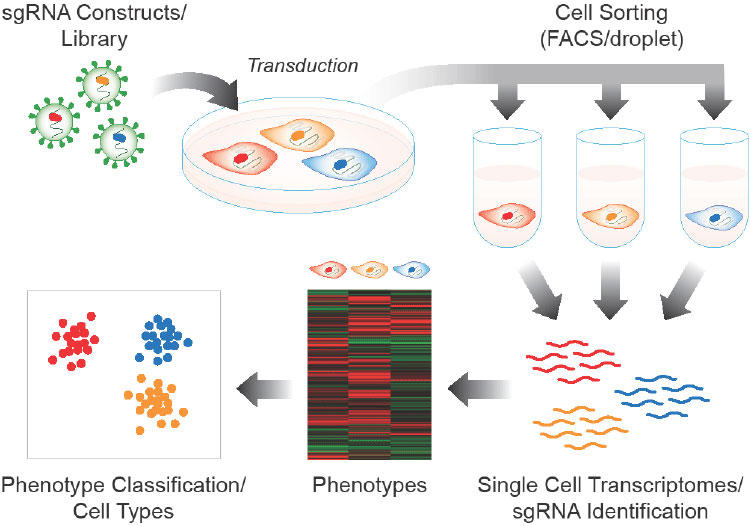
シングルセルRNA-seq分析は、個々の細胞応答を分析するための重要なツールです。しかし、全ての細胞集団は表現型的に不均一であるため、重要な遺伝子およびパスウェイであっても全ての細胞において活性化していない可能性があります。したがって、細胞集団のサブポピュレーションにのみ存在する実験条件に対する有意な応答は、容易に見逃されます。この課題に対処する1つの方法は、個々の細胞に識別可能なバーコードを標識することです。この最初のバーコード標識細胞集団からの子孫クローンにおける効果および応答は容易に評価することができ、どのような実験条件が共通の前駆細胞由来の異なる細胞群に異なる影響を及ぼし得るかというデータを抽出することができます。
- RNA-Seq分析で容易に検出できるRNAバーコードで細胞を容易に標識可能
- 5M、10Mまたは50Mレンチウイルスライブラリーの中から選択可能
- 複数の細胞集団に、識別可能で重複しないバーコードを標識可能
遺伝子発現の促進やパスウェイ解析と組み合わせて細胞特異的バーコードを検出するためには、細胞特異的なバーコードをmRNA上に発現させる必要があります。これはバーコードをRNA転写産物の3'または5'-UTRに組み込むことによって実現できます。 Cellectaのバーコードライブラリコレクションは、ゲノムDNAおよびRNA画分の両方で検出できるユニークなバーコードで100万個の細胞を容易に標識できます。シングルセルRNA-Seq分析と組み合わせて使用する場合、発現したバーコードを用いて異なる細胞における発現プロファイルおよび活性化遺伝子を同定することができるので、単一の前駆細胞由来の細胞の異なるクローン集団ならびに 異なるパスウェイの活性化や発現プロファイルを容易に同定することが可能です。
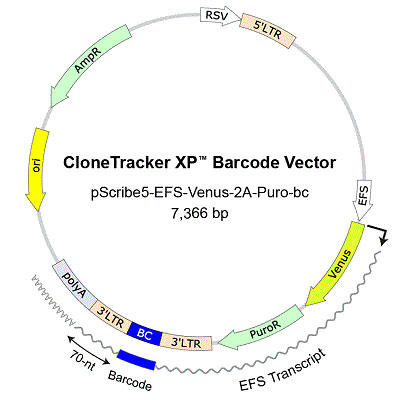
図.2 CloneTracker XP™ レンチウイルスバーコードライブラリーのベクターマップ























 このページを印刷する
このページを印刷する