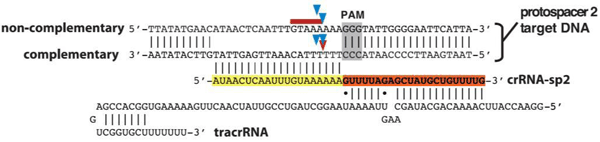
Fuら(2013)を始めとするCRISPR-Casシステムの特異性に関する初期の研究では、本技術を遺伝子治療のような高い特異性を要するアプリケーションに利用できるのか懐疑的でした。近年の研究では、CRISPR-Casの特異性は高い、ということが報告されています。例えば、MITのEric Landerの研究室では、レンチウイルスに組込んだsgRNAのプールを用いてヒト細胞における大規模な変異誘発性スクリーニングを行っています。DNA損傷薬剤への耐性スクリーニングにおいて、間違った標的をしたsgRNA(ガイドRNA, gRNA)に比べ、正確に標的したsgRNA(ガイドRNA, gRNA)のほうが、遥かに高い頻度でDSB導入によるミスマッチ修復経路における遺伝子変異を引き起こすことが示されています(Wang, et al., 2014)。
マウスの白内障治療に成功したsgRNA(ガイドRNA, gRNA)は、12検体中10検体の形質転換動物において10種の予測されたオフターゲット部位に対して何ら影響を及ぼさず、残り2検体においてわずか1ヶ所のオフターゲット部位に変異を生じたのみでした(Wu, et al., 2013)。これらの報告やその他の研究報告 (Li, et al., 2013; Yang, et al., 2013)、および高い特異性を示すようより改善されたsgRNAs予測ソフトウェアツール(Ran, et al., 2013a; Xiao, et al., 2014)の利用を併せて考慮すると、CRISPR-Cas9によるオフターゲット変異誘発性は高い傾向にあると長い間心配されてきたことが過剰反応である可能性も大いにあります。
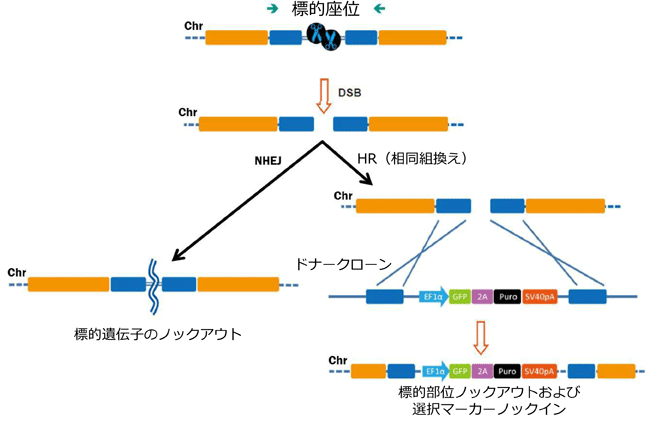
sgRNA(ガイドRNA, gRNA)デザインの改善だけではなく、Cas9変異体の使用によりCRISPR-Casによるオフターゲット(作用)変異誘発をさらに低減できることが示されています。野生型Cas9は2つのヌクレアーゼドメインをもち、それぞれが片方のDNA鎖を切断します。何れかのドメインを変異させることでCas9を「ニッカーゼ」へと転換することができ、一本鎖のみを切断するようにできます。ニッカーゼ変異体によって、sgRNA(ガイドRNA, gRNA)の結合特異性に変わりはないので、オフターゲット部位に送達される可能性はあるものの、単一のニックでは変異原性のNHEJを生ずることはありません。染色体上の各鎖を標的とする2種のsgRNA(ガイドRNA, gRNA)をデザインすることでねじれ型の二本鎖切断(DSBs)を作製することができ、NHEJまたはHRによる修復を受けることができます(図.3)。HarvardのGeorge Churchらの研究グループやMITのFeng Zhangらの研究グループはこの「一対のニッカーゼ」を利用する方策によりNHEJやHRの何れのDNA修復も効率よく誘導できることを報告しています(Ran, et al., 2013b; Mali, et al., 2013)。さらにZhangらは、3種の異なる部位を標的するsgRNA(ガイドRNA, gRNA)を用いた実験で、一対のニッカーゼを利用することでオフターゲット(作用)変異誘発の頻度を50倍から1500倍へと劇的に低減できることを示しました。
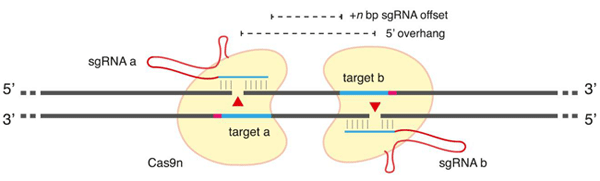
図.3 Cas9ダブルニッカーゼ手法の一般的な概略。Ran, et al.(2013b)より引用
一方で、ダブルニッカーゼのデザインは容易ではありません。お互いのPAM配列が近位に位置するsgRNA(ガイドRNA, gRNA)対("head-to-head"方向)に比べ、お互いのPAM配列が最も遠位に配置されたガイドRNA対("tail-to-tail"方向)では、より高い頻度で挿入欠損が形成されます(Ran, et al., 2013b; Mali, et al., 2013)。さらに、各sgRNA(ガイドRNA, gRNA)の末端同士の間隔(オフセット距離)も重要となります。最も効果の高いニッカーゼペアは、通常オフセット距離が0から20塩基です。ダブルニッカーゼ手法は、オフターゲット作用による期待しない二本鎖切断(DSBs)をほとんど生ずることなく、効率よく二本鎖切断(DSBs)を作製できる一方で、デザインの制約によりアプリケーションによっては適切な部位として使用可能な範囲が限定されます。 最後に、近年の報告では、最短17塩基のsgRNA(ガイドRNA, gRNA)配列は、標準型の20塩基のものと比較して、その挿入欠損形成効果が同程度であることが示されました(Fu, et al., 2014)。さらに、著者らは、これらの「切り詰め型sgRNA」を用いて4種の遺伝子を標的したところ、オフターゲット(作用)変異誘発性が最大5000倍まで低減できたと報告しています。しかし、この切り詰め型sgRNAが一般に効果的であるかどうかは、さらなる検討が必要でしょう。
以上より、sgRNA(ガイドRNA, gRNA)デザインと方策の改良、改変型Cas9ヌクレアーゼ(ニッカーゼ型)との併用などにより、当初考えられていたほどオフターゲット(作用)変異誘発性を過剰に憂慮することなく、遺伝子治療をはじめとする数々のアプリケーションにCRISPR-Cas9を活用できることは明白といえます。