Total RNAには多量のリボソームRNA(rRNA)が含まれ、酵母サンプルから抽出したtotal RNAでは、リボソームRNAは最大で全体の97%を占めます。Lexogen社RiboCop rRNA Depletion Kit for Yeastは、分解の進んだtotal RNAやインタクトなtotal RNAから、細胞質rRNA (25S, 18S, 5.8S, 5S, 35S) やミトコンドリアrRNA (21S, 15S, ACI60_gr01, ACI60_gr02) を効率良く除去します。rRNA除去後のRNAは、次世代シークエンス解析を含む各種RNA解析にご使用いただけます。遺伝子発現解析には、Lexogen社CORALL RNA-Seq V2 Library Preparation Kitをおすすめしていますが、ランダムプライミングを利用した他のtotal RNAライブラリ調製キットにも対応しています。
RNA-Seqのサンプル調製に、酵母用リボソームRNA除去キット RiboCop rRNA Depletion Kit for Yeast
- カテゴリから探す > 分子生物 > DNAシーケンシング > 次世代シーケンシングキット
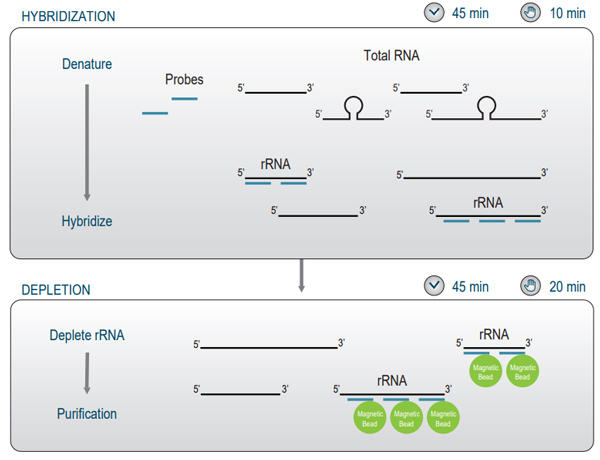
ワークフロー
プローブとターゲット配列のアクセス向上のため、アフィニティプローブとtotal RNAを混ぜて変性させます。その後、昇温によりRNAとプローブをハイブリダイゼーションします。この間、ハイブリダイズしたRNAを除去するために使用する除去ビーズを調製します。残ったRNAを磁気ビーズを用いて精製し、終了です。精製に磁気ビーズを用いる事でプロトコルをオートメーション化し、また容易で短時間のプロトコルとなっています。
RiboCop で調製した RNA サンプルはランダムプライミング法の RNA-Seqライブラリ調製キットと共にご使用いただけます。
パフォーマンス
幅広いインプット量で効率よくrRNAを除去
RiboCop rRNA Depletion Kit for Yeastを用いて、幅広いインプット量 (10 ng〜1 µg) のSaccharomyces cerevisiae total RNAからrRNAを除去したところ、rRNA除去により、rRNAに対するリードを〜1%まで抑えることができました(図1)。
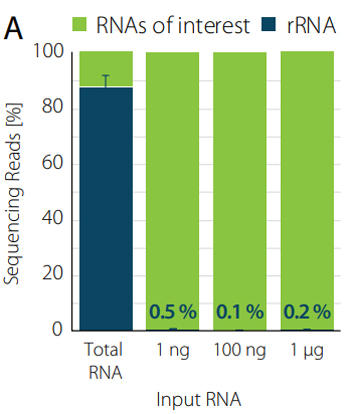
図1. 幅広いインプット量で効率よくrRNAを除去
Saccharomyces cerevisiaeのtotal RNAからRiboCop for Yeastを用いてrRNAを除去した。得られたRNAについて、Lexogen社のCORALL Total RNA-Seq V2ライブラリ調製キットを用いてライブラリを調製し、NextSeq500(1×50 bp)にてシークエンス解析を行った。rRNAに対するリードのパーセンテージを青色で示す。
RiboCop for Yeastはバイアスなく、発現プロファイルを維持
RiboCop for Yeastは効率良くrRNAを除去する一方で、転写産物の発現プロファイルを維持します。RiboCop for Yeastで処理していないサンプルとRiboCop for YeastでrRNAを除去したサンプルでは、転写産物は高い相関を示しました(図2)。また、インプット量に関わらず、rRNAを除去することにより、エクソンに対するリードが非常に増えました(図3)。
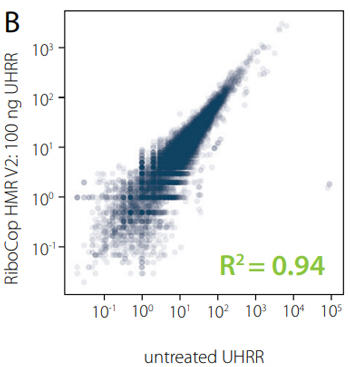
図2. RiboCop for Yeastはバイアスなく、発現プロファイルを維持
RiboCop for Yeastで処理していないS. cerevisiaeのRNAとRiboCop for Yeast でrRNAを除去したS. cerevisiaeのRNAについて、Lexogen社のCORALL Total RNA-Seq V2ライブラリ調製キットを用いてライブラリを調製し、NextSeq500(1×50 bp)にてシークエンス解析を行った。得られたリードはSTAR aligner を用いてS. cerevisiaeのリファレンスゲノムにマップし、FeatureCountsを用いてカウントした。
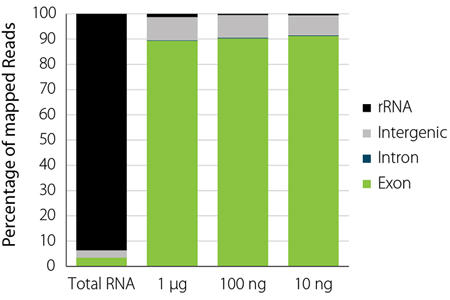
図3. マップされたリードの内訳
RiboCop for Yeastで処理していないS. cerevisiaeのRNAとRiboCop for Yeast でrRNAを除去したS. cerevisiaeのRNAについてリードの割合を比較した。rRNAを除去することにより、エクソンに対するリードが非常に増えた。
rRNA除去により検出遺伝子数が上昇
RiboCop for Yeast でrRNAを除去することにより、検出遺伝子数が上昇しました (図4)。
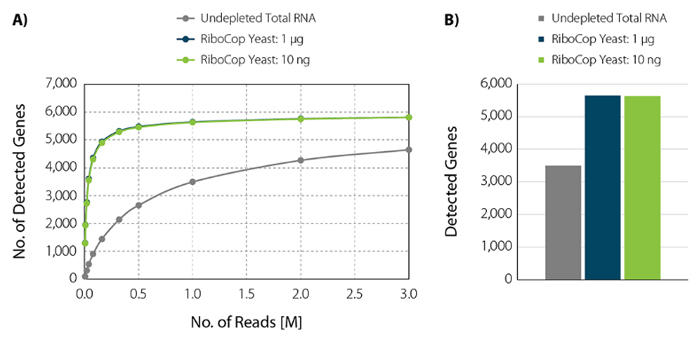
図4. rRNA除去により検出遺伝子数が上昇
A) 検出遺伝子数とFeatureCountsでカウントしたエクソンに対するユニークリード数をプロット。
B) 1Mリードにおける、サンプルあたりの検出遺伝子数を示す。
RiboCop for Yeastを用いて、1 µgまたは10 ngのS. cerevisiaeのtotal RNAからrRNAを除去した。Lexogen社のCORALL Total RNA-Seq V2ライブラリ調製キットを用いてライブラリを調製し、NextSeq500(1×50 bp)にてシークエンス解析を行った。得られたリードはSTAR aligner を用いてS. cerevisiaeのリファレンスゲノムにマップし、FeatureCountsを用いてカウントした。
優れた再現性
RiboCopによるrRNA除去は再現性が高いのも特長です。幅広いインプット量で高い再現性が得られています(図5)。
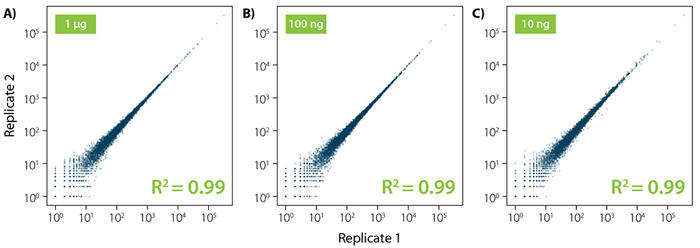
図5. 優れた再現性
10 ng, 100 ng, 1 µg のS. cerevisiaeのtotal RNAを出発材料として、独立した2回のrRNA除去を行った。Lexogen社のCORALL Total RNA-Seq V2ライブラリ調製キットを用いてライブラリを調製し、NextSeq500(1×50 bp)にてシークエンス解析を行った。得られたリードはSTAR aligner を用いてS. cerevisiaeのリファレンスゲノムにマップし、FeatureCountsを用いてカウントした。どのインプット量でも、転写産物の高い相関が見られた。
RiboCop for Yeastは様々なインプット量で発現プロファイルを維持
RiboCopは幅広いインプット量で再現性良くrRNAを除去可能です。10ng〜1gのインプット量で、転写産物の高い相関が見られました (図6)
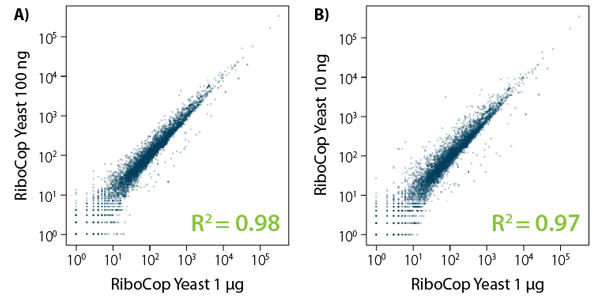
図6. 様々なインプット量で発現プロファイルを維持
RiboCop for Yeastを用いて、10 ng, 100 ng, 1 µg のS. cerevisiaeのtotal RNAからrRNAを除去した。Lexogen社のCORALL Total RNA-Seq V2ライブラリ調製キットを用いてライブラリを調製し、NextSeq500(1×50 bp)にてシークエンス解析を行った。得られたリードはSTAR aligner を用いてS. cerevisiaeのリファレンスゲノムにマップし、FeatureCountsを用いてカウントした。10 ng〜1 µgのインプット量で、転写産物の高い相関が見られた。
酵母用リボソームRNA除去キット
| 品名 | メーカー | 品番 | 包装 | 希望販売価格 |
|---|---|---|---|---|
RiboCop rRNA Depletion Kit for Yeast, Yeast |
LEX | 190.24 | 24 PREP. |
¥290,000 |
RiboCop rRNA Depletion Kit for Yeast, Yeast |
LEX | 190.96 | 96 PREP. |
お問い合わせ |
商品は「研究用試薬」です。人や動物の医療用・臨床診断用・食品用としては使用しないように、十分ご注意ください。
※ 表示価格について
- 「RiboCop rRNA Depletion Kit for Yeast」は、下記のカテゴリーに属しています。
-
- カテゴリから探す > 分子生物 > DNAシーケンシング > 次世代シーケンシングキット























 このページを印刷する
このページを印刷する