タンパク質の一次構造すなわちアミノ酸配列を知ることは、対象としているタンパク質の研究を進める上で必須の情報で、研究を進めるうえで大きな武器となります。本稿の初版では国立遺伝学研究所が開設している日本DNAデータバンク(DDBJ)の諸機能を利用する方法を紹介しましたが、現在はほとんどがゲノムに関する機能の提供となっています。そこで、National Center for Biotechnology Information (NCBI)のサイトを利用することを前提として話を進めます。
図11-1. NCBIのサイト(参照2022.1.1)
ラクトフェリンに限らず既知のタンパク質のアミノ酸配列を求めるには、NCBIのサイトで最上段にある[All Databases]とある欄、あるいは右側にある[Popular Resources]欄でProtein を選択します(図11-1)。さらにキーワード欄にタンパク質名、例えばlactoferrinと入力しますが、ウシラクトフェリンのアミノ酸配列を知りたい場合、AND/ORなどの検索式を使わないで単にスペースで区切ってcowと入力すると右欄中段にどのような検索式で検索されたかが表示されます。この場合はlactoferrin[All Fields] AND ("Bos taurus"[Organism] OR cow[All Fields])という 検索式で検索されたことが分かります。
この検索でヒットした多数のアミノ酸配列の中から、目的に合致するデータを選択しなければなりません。これらのアミノ酸配列データのリストには、ラクトフェリンの全配列だけではなく部分配列もあることが構成アミノ酸数を見て判断できます。他方、ヒットした配列データのほとんどは遺伝子の塩基配列からのもので、それらにはMetから始まるシグナルペプチドの部分も含まれています。しかし、実際に分泌されたタンパク質ではシグナルペプチドの部分は切断・除去されています。アミノ酸配列を使用する目的によりますが、分泌タンパク質のN末端部分の配列を知るためには、DNA塩基配列からのデータではなくペプチドシークエンサーやX線結晶解析から得られた配列データを探すことになります。図11-2にその検索結果の一例を示しました。アクセッション番号1BLF_AがX線結晶解析によって得られたアミノ酸配列データで、689残基となっています。また、ヒットした配列データをクリックして開くと、アミノ酸配列の各部分についての説明もあり、たとえば1-20がシグナルペプチドであることも記載されています。
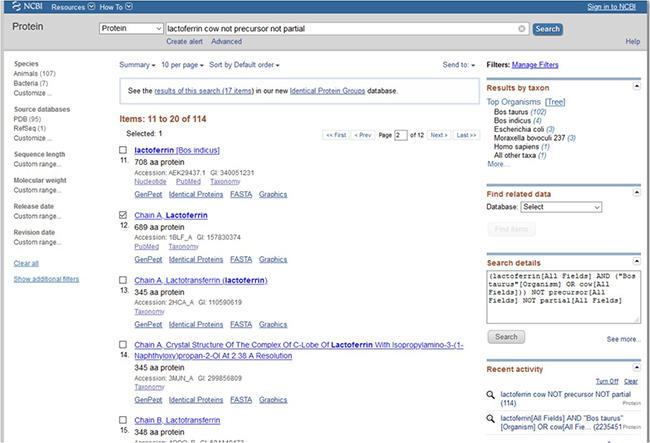
図11-2. NCBIのサイトでキーワード欄にlactoferrin cow not precursor not partialと入力して検索した例
アクセッション番号1BLF_Aにチェックを入れている。リンクを開くと3種類の表示(GenPept、Grapics、FASTA)が選択できる。図11-3にGraphicsを図11-4にFASTA形式を選択した例を示す。
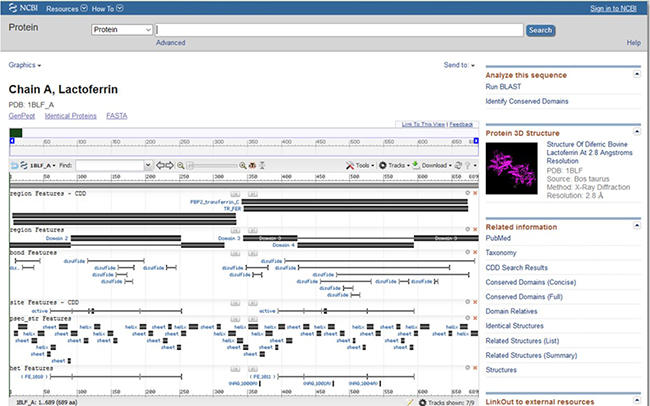
図11-3. Graphicsを選択した画面
APRKNVRWCTISQPEWFKCRRWQWRMKKLGAPSITCVRRAFALECIRAIAEKKADAVTLDGGMVFEAGRD PYKLRPVAAEIYGTKESPQTHYYAVAVVKKGSNFQLDQLQGRKSCHTGLGRSAGWIIPMGILRPYLSWTE SLEPLQGAVAKFFSASCVPCIDRQAYPNLCQLCKGEGENQCACSSREPYFGYSGAFKCLQDGAGDVAFVK ETTVFENLPEKADRDQYELLCLNNSRAPVDAFKECHLAQVPSHAVVARSVDGKEDLIWKLLSKAQEKFGK NKSRSFQLFGSPPGQRDLLFKDSALGFLRIPSKVDSALYLGSRYLTTLKNLRETAEEVKARYTRVVWCAV GPEEQKKCQQWSQQSGQNVTCATASTTDDCIVLVLKGEADALNLDGGYIYTAGKCGLVPVLAENRKSSKH SSLDCVLRPTEGYLAVAVVKKANEGLTWNSLKDKKSCHTAVDRTAGWNIPMGLIVNQTGSCAFDEFFSQS CAPGADPKSRLCALCAGDDQGLDKCVPNSKEKYYGYTGAFRCLAEDVGDVAFVKNDTVWENTNGESTADW AKNLNREDFRLLCLDGTRKPVTEAQSCHLAVAPNHAVVSRSDRAAHVKQVLLHQQALFGKNGKNCPDKFC LFKSETKNLLFNDNTECLAKLGGRPTYEEYLGTEYVTAIANLKKCSTSPLLEACAFLTR
図11-4. FASTAを選択した場合のウシラクトフェリン(1BLF_A)のアミノ酸配列
>で始まる行はタイトルあるいはコメント。1行当り60カラムのデータでアミノ酸は1文字表記。
以上の操作で得られたラクトフェリンのアミノ酸配列データと似た配列を持つタンパク質が他にあるかどうかを検索してみます。この方法は「相同性(ホモロジー)検索」といい、対象としているタンパク質のアミノ酸配列(クエリー配列といいます)と同じあるいは類似の配列を持つタンパク質が検索できます。図11-1の左欄でHomologyを選択し、Toolsを見るといくつものツールがあります。Basic Local Alignment Search Tool (BLAST)を使ってみます。BLASTの入力画面でblastpになっていれば、タンパク質のアミノ酸配列をタンパク質データベースから検索することとなります(表11-1)。実際の検索は、[Enter Query Sequence]でアクセッション番号かgi(補足欄参照)の入力、あるいはFASTA配列をコピー・ペースト、または配列ファイルを入力します。この画面で検索対象とするデータベースや検索プログラム(アルゴリズム)も変更できます。その他にも設定を変更して検索できますが、本稿ではデフォルト設定のままで行っています。
最後に画面の一番下にあるBLASTボタンをクリックします。結果はヒットした配列のグラフィックでの比較、相同性が認められるタンパク質のリスト(相同性の高い順に表示)、配列アラインメントで構成されています。アラインメントはクエリー配列とヒットしたそれぞれのタンパク質とのアミノ酸配列を比較した一覧がそれぞれの対について表示されます。上段にクエリー配列が、3段目に対象タンパク質の配列が、中段に両者で同一となっているアミノ酸などが表示されます。
図11-1で左側に表示されている[Resource List]でProteinを選択すると様々なメニューが表示されます。その中にBlast (Stand-alone)があり、自分のパソコンに検索プログラムをインストールして使うことができます。| 対象(クエリー)配列 | 検索するデータベース | 用いるプログラム | |
|---|---|---|---|
| アミノ酸配列 | アミノ酸配列 | blastp | |
| 塩基配列 | tblastn* | ||
| 塩基配列 | アミノ酸配列 | blastx | |
| 塩基配列 | blastn | ||
| 塩基配列 | tblastx** | ||
*塩基配列データベースを用い、アミノ酸配列に翻訳して検索
**クエリー配列をアミノ酸に翻訳し、塩基配列データベースもアミノ酸配列に翻訳して検索
何らかのペプチドのアミノ酸配列が分かっていて、それが既に知られているペプチドなのか、あるいは何というタンパク質から得られた断片なのかを知りたい場合はよくあります。そのような場合にも上述のBLASTで、一文字記号でのアミノ酸配列を入力して検索します。得られた結果は上述した「相同性の検索」と同じ構成の画面となります(図11-5)。なお、BLASTは図11-1の画面右側のメニュー(Popular Resources)から選択できます。
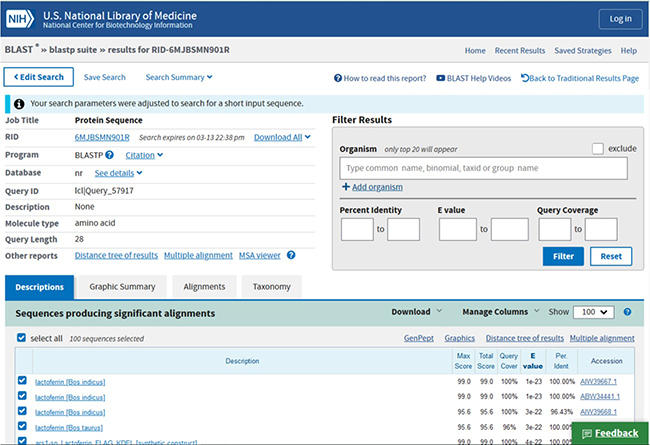
図11-5. BLASTでウシラクトフェリシンのアミノ酸配列(FKCRRWQWRMKKLGAP SITCVRRAFALE)を入力して検索した結果
また、ウシラクトフェリンのアミノ酸配列をヒトやその他の動物のラクトフェリンのアミノ酸配列と比較する場合には、これらの配列を並べて比較しますが、これをアラインメントといい、複数の配列で行う場合は特にマルチプルアラインメント(多重整列)といいます。この操作で、対象としているアミノ酸配列に共通なパターンがあるかどうかを探すことが出来ます。いくつかのタンパク質の間で特徴的な配列部分(配列モチーフ)が見つかれば、タンパク質の機能だけでなく遺伝子構造に関する示唆も得ることができます。
これを行うための手段(プログラム)としてClustalがあります。幾つかのバージョンがありますが、ここではEMBL-EBIのClustal Omegaを使ってみます。比較する複数のアミノ酸配列をFASTA形式やその他の形式で、空白行は入れずに入力し実行(submit)します。
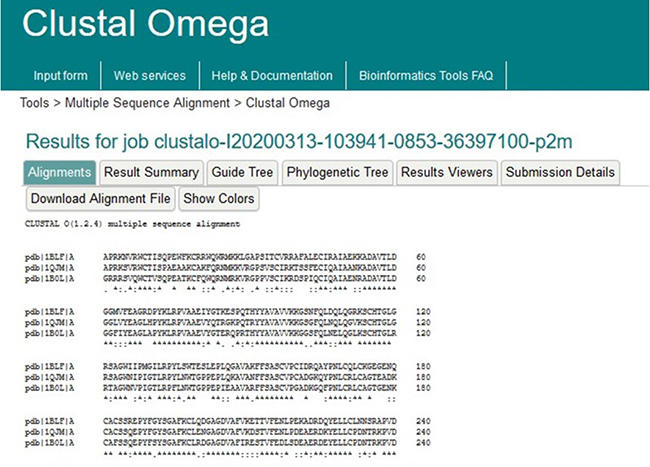
図11-6. ウシ、ウマ、ヒトのラクトフェリン配列をClustal Omegaを用いて比較した画面の一部
(https://www.ebi.ac.uk/Tools/msa/clustalo/)























