合成したペプチドやヌクレオチドの質量を測定して目的物を確認したり、あるいは精製した試料をSDS-PAGEや分析レベルのクロマトグラフィーと同様に、目的成分以外のものの混在の有無をみること、すなわち純度を検定することは、最もシンプルなMALDI-TOF MSの利用法です。その他に以降に述べるようなさまざまな応用例が報告されており、プロテオーム解析・プロテオミクス解析などの主力となっています。新規の応用例は今後も増えてゆくと期待されます。
混合物中に含まれる特定の物質を対象としてその質量を測定する方法も開発されています。SELDI-affinity mass spectrometryといわれる方法で、目的物質と特異的あるいは群特異的(group specific)に相互作用する物質をサンプルプレートに担体として固定化したものを用います。混合物中の特定の目的物質だけがこのサンプルプレート上に捕捉され、それにパルスレーザー光を当ててイオン化して質量分析を行います。たとえばラクトフェリンおよびその半分子であるNローブの測定にはn-ブチル基が用いられています。この方法を、経口摂取されたラクトフェリンが消化管内でどのように分解され、さらに吸収され消失するかを追跡するのに用いてみました。
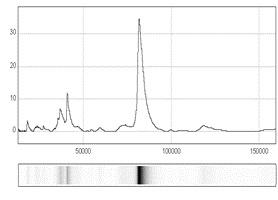
図8-1.ラクトフェリンを経口投与したラットの胃内容物のSELDI-TOF MSパターン1) N末端を有するフラグメントだけがアフィニティー担体によって補足され、それらの質量スペクトルが得られました。
図8-1には未分解のラクトフェリンのピークの他に、分解産物中の特定のフラグメントのピークが観察されています。このフラグメントは、ブチル基やCM基と親和性を示すNローブの特定の部位を有するペプチドです。各ローブに対するモノクローナル抗体を用いたウエスタンブロット法など、免疫化学的方法も同じ試料について、併用したところ、CローブよりもNローブの方が消化・吸収されやすい傾向が観察されました。
このアフィニティー質量分析法は、疾病マーカーの探索やモニタリングなど、特定のタンパク質の発現解析およびイムノアッセイやリガンドアッセイ、その他の相互作用解析に有効でしたが、すでに製造・販売終了となっています(補足aを参照)。
MALDI-TOF MSによって質量を精度良く測定できることは、質量分析の応用範囲の拡大をもたらしました。その一つがタンパク質の同定です。ペプチドマスフィンガープリント法(PMF法)と呼ばれる方法は以下のように行います。まず、精製した目的タンパク質を第5章でも触れたプロテアーゼ消化の操作を行います。次いで、この分解混合物の質量スペクトルを測定して、生成したフラグメントの各質量を決定します。最後にこれらの質量値を既知タンパク質での値と突き合わせます。アミノ酸配列が既知のタンパク質については、各種のプロテアーゼを用いた場合に生成するフラグメントとそれらの質量を計算することは容易なことです。現在、膨大な数のタンパク質についてオンラインで利用できるデータベース(ペプチドマスデータベース)があり、対象タンパク質と一致するフラグメント分布、すなわち同じペプチドマップを表すものがあるかどうかを調べることができます。測定した質量の値が不正確であったり、検索範囲や検索条件が不適であった場合には、信憑性の高い結果は得られませんので注意が必要です。また十分に信頼の置けるデータを適切な検索条件で検索しても該当するタンパク質が無い場合には、まだデータベースに登録されていない新規のタンパク質である可能性があります。
ゲノム解析に対して、タンパク質レベルでの解析をプロテオーム解析(Proteomics)といっています。タンパク質を同定することがその最初の段階です。この時、試料は電気泳動あるいは各種クロマトグラフィーによって精製します。二次元電気泳動法も一般的です。二次元電気泳動とは、異なる条件での電気的分離法の2種類を組み合せて分離する方法で、一次元目に等電点電気泳動法を、二次元目にSDS-電気泳動法を用いる方法が一般的です。タンパク質はバンド(帯)ではなくスポット(点)として分離され、色素染色法や銀染色法で可視化します。その他に膜に転写して検出する場合もあります。
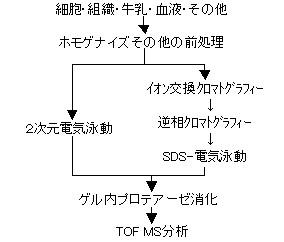
図8-2.プロテオーム解析に用いられるPMF法のフローチャート。2種類のクロマトグラフィーを行う2次元クロマトグラフィーと電気泳動法の組み合わせを3次元分離ということもあります。
タンパク質の解析を行う場合、図8-2に示したように、アプローチの仕方は一通りではなく、試料の量や性質によって選択します。たとえば試料量が多く、かつ対象タンパク質の分子量が大きい(200 kDa以上)あるいは小さい(10 kDa以下)場合、または塩基性タンパク質や疎水性タンパク質の場合は、イオン交換クロマトグラフィーをまず行い、さらに逆相クロマトグラフィーで目的画分を分取した後、SDS-電気泳動で目的のタンパク質バンドを切り取ってペプチドマスフィンガープリント(PMF)法を行う方法が現実的です。最終的にポリアクリルアミドゲル電気泳動で分離したタンパク質やペプチドの場合は、そのバンドを切り出し、ゲル内でプロテアーゼによって分解してペプチド断片を得(in-gel digestion法、第5章を参照)、このペプチド集団に対してTOF- MSによる解析を行います。
アミノ酸配列を決定する方法として、次章で述べるN末端アミノ酸を化学的に逐次分解して決定する方法と、前章で触れたMALDI-TOF MSによるPost- Source Decay MS/MS測定法(PSD解析法)とがあります。これはイオン化して加速され飛行中の分子イオンに崩壊を起こさせ(メタステイブル分解)、生じた分子断片(フラグメント)を精度良く分離して検出します。しかし、質量が全く同じであるLeuとIleは判別できません。これらを識別するには、分子イオンを不活性ガスに衝突させてさらに小さなフラグメントを生成して検出するCollision Induced Dissociation (CID)法を用います。リン酸化など翻訳後修飾されたタンパク質もこの方法で解析することができます。なお、この方法ではペプチドのアミノ酸配列情報を得ることが出来るだけでなく、オリゴヌクレオチドやオリゴ糖の配列情報の解析にも応用できます。
エドマン法(第9章参照)を応用したり、あるいはN末端かC末端から切断する酵素を用いてアミノ酸配列を行う方法(ラダーシーケンス法)もあります。前者の方法はフェニルイソチオシアネート(PITC)に数%のフェニルイソシアネート(PTC)を加えてN末端の切れ方の異なる混合物を生成させます。また後者の方法では、たとえばカルボキシペプチドダーゼを僅かに異なる条件で反応させて、アミノ酸残基の切れ方が異なるペプチドを作成します。こうして作成した試料中のペプチドの質量の差を比較することによって、N末端アミノ酸配列あるいはC末端アミノ酸配列を決めることができます。
これらアミノ酸配列の決定については、PSD法およびCID法の併用、さらには酵素を用いる方法や次章で解説するエドマン法と照らし合わせて結果を検討することが必要です。

























