タンパク質の原子座標データと表示用のソフトウエアがあれば、タンパク質の形すなわち立体的な構造をパーソナルコンピュータの画面上に表示して、かつ様々な操作をすることができます。 立体構造データベースであるRCSBのProtein Data Base (PDB)にアクセスして目的タンパク質のX線結晶構造解析による座標データを入手します。ここで得られる立体構造データ形式は、Brookhaven PDB フォーマットといわれる形式です。ここではPDB IDが不明の場合はタンパク質名で検索が行えます。lactoferrinと入力した場合の画面を図14-1に示します。非常に多くのデータがヒットするので、さらに絞り込みを行わなければなりません。目的とするpdbデータが見つかったならばダウンロードして、分子モデル表示のためのソフトウエアにこのデータを読み込ませることとなります。
図14-1. RCSB PDBのトップサイトで検索欄にlactoferrinと入力した画面。ウシラクトフェリンならばBos taurusを選択する。X線結晶解析によるデータ、あるいはNMRデータの選択もこの画面で行える。タンパク質によっては、ごく最近発達したクライオ(低温)電子顕微鏡法(Cryo electron microscopy)による解析データも増えてきている。 (参照2022.1.4)
分子構造を表示するソフトウエアは有料のものから無料で使えるもの、Windows、iOS 、Linuxさらには各種タブレットやスマートフォンで動作するものまで見つかります。本講座では個人で気軽に使えるフリーソフトの使用を前提とします。各ソフトウエアによって表現が異なることがありますが、針金モデル (wireframe)、骨格モデル (backborn)、空間充填モデル(spacefill)、リボンモデル(ribbons) シリンダーモデル (cylinders)などでタンパク質の立体構造を表示することができ、かつ自由に分子を回転させたり、あるいは分子内の任意の部分を色分けして表示することも出来ます(図14-2)。さらに一部を空間充填モデルで表示し、他の部分をリボンモデルなどという異なるモデルによる組み合せでの表示も可能です。
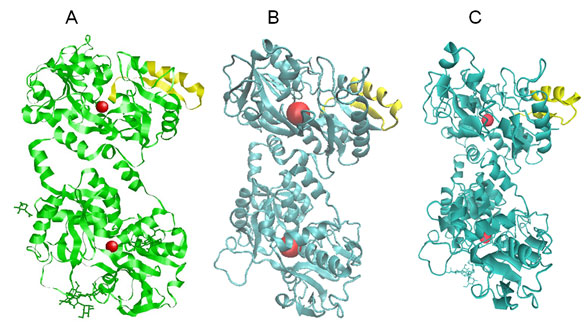
図14-2. 3種類のソフトウエアでウシラクトフェリン分子 (1BLF.pdb1) )を表示した。黄色で識別した部分はNローブに見いだされた抗菌性ペプチドのラクトフェリシンで、2個の●(赤丸)は鉄イオン。用いたソフトウエアは(A) RasMol(PICT形式で保存した後にjpeg形式に変換、貼付け)、(B) VMD(スクリーンショットを直に貼付け)、(C) CMol (iPad、スクリーンショットに保存し、パソコンに送ったpng形式のファイルを貼付け)で、RasMolとCMolではcartoon、VMDはnew cartoonで表示した。
第3章で作成したウシラクトフェリンCローブに対するモノクローナル抗体の反応性を調べると、興味ある結果が得られました。第10章で決定した抗体結合部位の配列が、ヒトラクトフェリンにも存在しています。しかし、ELISA法やドットブロット法による検定では、ウシラクトフェリンCローブに対するモノクローナル抗体に対する反応性は認められませんでした。そこでポリペプチド主鎖の折りたたみ構造をウシとヒトのラクトフェリンについて比較したところ、図14-3に示したようにその重なりの度合いが非常に高いことが分かりました。これはモノクローナル抗体との反応が主鎖ではなく側鎖との相互作用であること、またそれらの配向が微妙に異なっていることが示唆されましたが、ここで示した抗体結合部位とは離れたペプチド鎖の影響を受けている可能性もあります。そこでラクトフェリンを変性させてからELISAプレートに吸着させ、抗ウシラクトフェリンCローブモノクローナル抗体、次いでペルオキシダーゼ標識抗体で反応させたところ、ヒトラクトフェリンにもはっきりとした反応が認められました。抗体結合部位近傍の主鎖、側鎖の状態を分子モデルで詳細に調べたところ、未変性のヒトラクトフェリンでは667Gln, 670Ala, 671Gly, 675Leuが抗体との結合を妨げているものと結論されました2)。なお、図14-4に示したものは、図14-3で示したペプチド部分を含むドメインの主鎖の構造モデルで、該当する配列AGWNIPMLIはα5に含まれています。

図14-3. ProteinAdviser(富士通九州システムエンジニアリング)によって示した、ウシとヒトラクトフェリンの抗体結合部位のポリペプチド主鎖の重なり具合2)。アミノ酸配列AGWNIPMLIはウシラクトフェリンでは465-474、ヒトラクトフェリンでは468-477に存在。
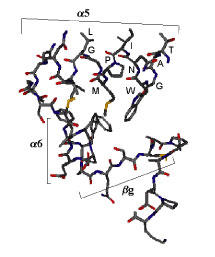
図14-4.ウシラクトフェリンのCローブのドメインの一つ(464-498)は、抗Cローブモノクローナル抗体との反応部位を含んでいます2)。このドメインはα5(463-479)とα6(482-486)の二つのヘリックス、βg(488-492)の一本のβストランドを含んでいます。(ProteinAdviserで表示)
ラクトフェリン分子の立体的なモデルを、3Dプリンターを用いて作製できます。実際に表面モデル(surface model)と、分子の骨格構造を示すリボンモデルで作製し図14-5に示しました。その詳細については省略しますので、作成過程を紹介したレポート(ラクトフェリン学会ニュースレター)3)を参照して下さい。

図14-5.3Dプリンターで作ったラクトフェリン分子のリボンモデル。赤い球は鉄イオン(分かりやすくするために大きな球で示した)。

























