

FtsZ: 新たな抗生剤の標的となるチュ−ブリンホモログ
チューブリンホモログであるFtsZ (Filamenting temperature- sensitive mutant Z)は細菌の細胞分裂に密接に関与しており、新たな理想的な抗生剤の標的と考えられています。このタンパク質を欠損している変異体は細胞分裂することができず、フィラメント状に伸長し続けます。FtsZはGTPaseの一種でヌクレオチド依存性に一本鎖のフィラメントを形成し、頭尾重合により伸縮性のあるリングを形成します。このリングはZ-ringと呼ばれ、細菌が分裂する際に隔壁が形成される細胞膜の内側に形成されます。FtsZ重合反応は速やかに定常状態に到達しますが、FtsZのGTP加水分解により供給されているかのような継続的で迅速な補充により細胞分裂中も動的に維持されています(1-3)。FtsZは分裂部位に最初に出現し、細菌の分裂に関与する他のタンパクを誘導します。他の分裂関連タンパクの足場として働く一方で、FtsZ自身も細胞分裂作用を発揮し、細胞分裂を誘導します(1-6)。
FtsZは真核生物におけるチューブリンの原核生物におけるホモログと考えられています(5-11)。チューブリンとFtsZはいずれもGTP結合ドメイン(7-11)とGTPase活性(1-3, 12)を有しており、原繊維、2次元シート、原繊維リングを構築し、構造的に一致しています(5-11)。このような類似点があるにもかかわらず、これら2分子間での塩基配列のホモロジーは10-18%と低く(1, 13, 14)、FtsZの基礎となるサブユニットは単量体であるのに対してチューブリンのサブユニットはαとβのヘテロ2量体で構成されています(1-3)。また、in vitroにおけるFtsZ会合のためのGTPの加水分解の必要性は実験条件によって様々です;会合はしますがGTPase活性を欠損したFtsZ変異体はin vivoにおいて異なる細胞の表現形や機能を示します(17)。
最近になって、FtsZが新しい抗菌薬の新規標的として抗生物質研究において注目されています(18-23)。薬剤耐性菌の顕著な増加と新しい抗生物質開発の遅れから、細菌の分裂に関連するタンパクといった新規の標的に作用する抗菌剤が注目の的となっています(20, 24)。異なる種のFtsZ間のアミノ酸配列のホモロジーは35-99%で、その多くは40-60%の中に含まれます(www.cytoskeleton.com/new-products参照)。このような一致のレベルは創薬研究に主に以下に示す2通りの影響を与えます。1. 広い薬効範囲を持つ細菌FtsZに対するリガンドを同定することが難しくなります。2. 1種類のFtsZを標的として使用した場合、その種に特異性の高い薬剤を作製することができます。実際、HaydonらはFtsZ阻害剤であるPC190723は70%のホモロジーを持つStaphylococcus aureusとBacillus subtilisには作用しますが、Escherichia coli (51%; 対S. aureus、47%; 対B. subtilis)には作用しないことを報告しています(Figure 1)(25, 26)。
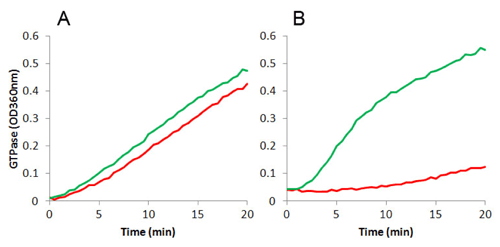
図1: 15uM PC190723(阻害剤:赤線)およびHEPES250バッファー(緑線)存在下でのE.coli由来FtsZ (0.5 mg/ml:A)およびS. aureus由来FtsZ (0.5 mg/ml:B)の重合。GTPase活性はMESG-based assay(カタログ番号 BK052)を用いて測定した。詳細は文献(25-27)参照。
GTPase活性測定や蛍光消光法、FRET解析法、沈降法、光散乱法といった方法がFtsZ重合の測定に用いられています。薬剤スクリーニングにはGTPase活性測定と蛍光消光法が適しています(詳細はwww.cytoskeleton.com/new-products参照)。タンパク相互作用の測定には、沈降法とGTPase活性測定が適しています。
これまでFtsZに関する多くの知見を得てきてはいるものの、以下のような多くの疑問がいまだ残されています。(i) Z-ringは何らかの力を発生する構造なのか、もしくは分裂に関連するタンパクが局在するための単なる足場なのか、(ii) 分裂においてヌクレオチド加水分解がどのような役割を果たしているのか、(iii)フィラメント中のどこで加水分解が起こっているのか、(v) ヌクレオチドの変換は単量体内、重合体内、もしくは両方で起こっているのか、(v) FtsZフィラメント間の側方相互作用はどのような性質を示すのか(3)。加えて、FtsZ重合におけるトレッドミル現象に関する報告や力学的不安定性に関してはさらに検討する必要があります。
FtsZおよびFtsZに関連した解析、異なるFtsZタンパク間のホモロジーに関する詳細な情報は サイトスケルトン社ウェブサイト「New Products」 ![]() をご参照ください。
をご参照ください。
上部イメージ:FtsZ重合体(Aylett, et al. (2010). PNAS, 107, 19766-19771.
【参考文献】
1. Romberg and Levin (2003). Assembly dynamics of the bacterial cell division protein FtsZ: Poised at the edge of stability. Annu. Rev. Microbiol. 57, 125-154.
2. Erickson et al (2010). FtsZ in bacterial cytokinesis: cytoskeleton and force generator all in one. Microbiol. Mol. Biol. Rev. 74, 504-528.
3. Mingorance et al (2010). Strong FtsZ is with the force: mechanisms to constrict bacteria. Trends Microbiol. 18, 348–56.
4. Osawa and Erickson (2006). FtsZ from divergent foreign bacteria can function for cell division in Escherichia coli. J. Bacteriol. 188, 7132-7140.
5. Erickson et al (1996). Bacterial cell division protein FtsZ assembles into protofilament sheets and minirings, structural homologs of tubulin polymers. Proc. Natl. Acad. Sci. USA. 93, 519-523.
6. Erickson (1997). FtsZ, a tubulin homologue in prokaryotic cell division. Trends Cell Biol. 7, 362-367.
7. Erickson (1998). Atomic structures of tubulin and FtsZ. Trends Cell Biol. 8, 133-137.
8. Nogales et al (1998). Structure of the alpha/beta tubulin dimer by electron crystallography. Nature. 391, 199-202.
9. Lowe and Amos (1998). Crystal structure of the bacterial cell-division protein FtsZ. Nature. 391, 203-206.
10. Lowe (1998). Crystal structure determination of FtsZ from Methanococcus jannaschii. J. Struct. Biol. 124, 235-243.
11. Erickson (1995). FtsZ, a prokaryotic homolog of tubulin? Cell. 80, 367-370.
12. de Boer et al (1992). The essential bacterial cell-division protein FtsZ is a GTPase. Nature. 359, 254-256.
13. de Pereda et al (1996). Tubulin secondary structure analysis, limited proteolysis sites, and homology to FtsZ. Biochemistry. 35, 14203-14215.
14. Mukherjee and Lutkenhaus (1994). Guanine nucleotide-dependent assembly of FtsZ into filaments. J. Bacteriol. 176, 2754-2758.
15. Mukherjee and Lutkenhaus (1998). Dynamic assembly of FtsZ regulated by GTP hydrolysis. EMBO J. 17, 462-469.
16. Mukherjee et al (2001). Assembly of an FtsZ mutant deficient in GTPase activity has implications for FtsZ assembly and the role of the Z ring in cell division. J. Bacteriol. 183, 7190-7197.
17. Bi and Lutkenhaus (1990). Analysis of ftsZ mutations that confer resistance to the cell division inhibitor SulA (SfiA). J. Bacteriol. 172, 5602-5610.
18. Margalit et al (2004). Targeting cell division: small-molecule inhibitors of FtsZ GTPase perturb cytokinetic ring assembly and induce bacterial lethality. Proc. Natl. Acad. Sci. USA. 101, 11821-11826.
19. Andreu et al (2010). The antibacterial cell division inhibitor PC190723 is an FtsZ polymer-stabilizing agent that induces filament assembly and condensation. J. Biol. Chem. 285, 14239-14246.
20. Kumar et al (2010). Discovery of anti-TB agents that target the cell-division protein FtsZ. Future Med. Chem. 2, 1305-1323.
21. Sass et al (2011). Antibiotic acyldepsipeptides activate ClpP peptidase to degrade the cell division protein FtsZ. Proc. Natl. Acad. Sci. USA. 108, 17474-17479.
22. Mathew et al (2011). Novel pyridopyrazine and pyrimidothiazine derivatives as FtsZ inhibitors. Bioorg. Med. Chem. 19, 7120-7128.
23. Schaffner-Barbero et al (2011). Targeting the assembly of bacterial cell division protein FtsZ with small molecules. ACS Chem. Biol. DOI: 10.1021/cb2003626.
24. Mintz (2011). Why the antibiotic discovery business needs to be revamped. Life Science Leader. http://www.lifescienceleader.com. October 2011.
25. Haydon et al (2008). An inhibitor of FtsZ with potent and selective anti-Staphyloccocal aureus activity. Science. 321, 1673-1675.
26. Haydon et al (2010). Creating an antibacterial with in vivo efficacy: synthesis and characterization of potent inhibitors of the bacterial cell division protein FtsZ with improved pharmaceutical properties. J. Med. Chem. 53, 3927-3936.
27. Chen and Erickson (2005). Rapid in vitro assembly dynamics and subunit turnover of FtsZ demonstrated by fluorescence resonance energy transfer. J. Biol. Chem. 280, 22549-22554.
28. Popp et al (2010). Suprastructures and dynamic properties of Mycobacterium tuberculosis FtsZ. J. Biol. Chem. 15,
新規FtsZタンパクツール
FtsZは細菌におけるチューブリンホモログで、細菌の分裂に不可欠なタンパク質です。FtsZの不活化により分裂が抑制されることから新しい抗菌薬の標的として注目されています。
FtsZタンパク質はsるていどのホモロジーを有しているものの、これらのタンパク質に対する阻害剤は異なる親和性、効果を示します。したがって、標的精度を向上するには異なる種由来のタンパク質をスクリーニングする必要があります。
種選択的抗菌薬のスクリーニング精度を向上させるために、Cytoskeleton社ではStaphylococcus aureus、Streptococcus pneumoniae、Enterococcus faecalis由来のFtsZタンパク質を提供しています。他の種由来のFtsZタンパク質もご要望によりご提供可能です。
| FtsZタンパク質の結晶構造 Low and Armos (1998). Nature, 391, 203-206. |
 |
| FtsZ種由来 | %ホモロジー(vs E. coli) | 品番 |
|---|---|---|
| Escherichia coli | 100 | FTZ05 |
| Salmonella enteritica | 99 | NA |
| Staphylococcus aureus | 47 | FTZ02 |
| Streptococcus pneumoniae | 50 | FTZ03 |
| Enterococcus Faecalis | 49 | FTZ04 |
| 品名 | メーカー | 品番 | 包装 | 希望販売価格 |
|---|---|---|---|---|
FtsZ Protein, Staphylococcus aureus  |
CYT | FTZ02-A | 1*1 MG |
¥130,000 |
FtsZ Protein, Staphylococcus aureus  |
CYT | FTZ02-B | 5*1 MG |
お問い合わせ |
FtsZ Protein |
CYT | FTZ03-A | 1*1 MG |
¥130,000 |
FtsZ Protein |
CYT | FTZ03-B | 5*1 MG |
お問い合わせ |
FtsZ Protein, Streptococcus pneumoniae  |
CYT | FTZ04-A | 1*1 MG |
¥130,000 |
FtsZ Protein, Streptococcus pneumoniae  |
CYT | FTZ04-B | 5*1 MG |
お問い合わせ |
FtsZ protein, Escherichia coli  |
CYT | FTZ05-A | 1*1 MG |
¥130,000 |
FtsZ protein, Escherichia coli  |
CYT | FTZ05-B | 5*1 MG |
お問い合わせ |
 FtsZタンパク質(サイトスケルトン社;CYT)
FtsZタンパク質(サイトスケルトン社;CYT)
■ CYTOSKELETON NEWS バックナンバー
- 2020年10月号 紡錘体 - 可視化に向けた新規ツール
- 2020年8月号 細胞膜染色用蛍光プローブ
- 2020年4月号 生細胞におけるF-アクチンプローブ
- 2020年3月号 コロナウイルスと細胞骨格
- 2020年2月号 タウ(Tau)の将来性をMapping
- 2020年1月号 Rho GTPaseによる細胞遊走制御
- 2019年12月号 表現型プロファイリング:アクチンに焦点を当てたがん治療
- 2019年11月号 チューブリンの過剰グルタミル化、ミトコンドリア、神経変性
- 2019年9月号 細胞運動性を制御するために相互作用する膜張力とアクチン細胞骨格
- 2019年8月号 Rac1B、がん、およびRac1
- 2019年7月号 Rhoファミリー GTPases、神経可塑性、およびうつ状態
- 2019年6月号 アクチンメチオニン酸化: 動的制御の次の段階
- 2019年5月号 ミクログリアと神経変性疾患
- 2019年2月号 生細胞画像化に対するCNS疾患や障害
2018年
- 2018年12月号 アクチン細胞骨格とメカノトランスダクション(機械的シグナル伝達)
- 2018年11月号 軸索再生と細胞骨格
- 2018年10月号 ニューロンにおける微小管と極性
- 2018年8月号 Rab GTPase と 神経変性
- 2018年7月号 SUMO レスリング: バランスが全て
- 2018年6月号 なぜ K-Ras は発がん特異性を示すのか?
- 2018年5月号 治療標的としてのユビキチンプロテアソームシステム:チューブリンは関与するか?
- 2018年4月号 RhoファミリーGEFと樹状突起スパインの構造的可塑性
- 2018年3月号 βカテニンとTFC/LEF-1の翻訳後修飾による標準的なWntシグナル制御
- 2018年2月号 がん抑制遺伝子p53の翻訳後修飾による機能の調整
- 2018年1月号 自閉スペクトラム症におけるGEF Trioの役割
2017年
- 2017年12月号 プロフィリン: アクチン結合タンパク質の多機能な役割
- 2017年11月号 ミトコンドリアにおけるアセチル化:新たな考え方と治療への応用の可能性
- 2017年9月号 翻訳後修飾のアセチル化による微小管の安定化
- 2017年8月号 神経軸索におけるアクチンリングを基盤とした周期的膜骨格(PMS)
- 2017年7月号 E3ユビキチンリガーゼMdm2によるがん抑制遺伝子p53の翻訳後制御
- 2017年6月号 多能性幹細胞(PSC)での転写因子による翻訳後制御
- 2017年5月号 Arf6 GEF と癌細胞の浸潤・転移
- 2017年4月号 PTEN(Phosphatase and Tensin Homolog)による翻訳後制御
- 2017年3月号 Tau の翻訳後修飾: アルツハイマー病の治療標的
- 2017年2月号 樹状細胞の移動におけるアクチン結合タンパク質とF-アクチン
2016年
- 2016年11月/12月号 GEF を介した GTPase シグナル伝達の低分子阻害剤
- 2016年9月号 FtsZ タンパク質: 抗菌薬の新規ターゲット
- 2016年7月号 翻訳後修飾(PTM)は心臓病において細胞骨格タンパク質を調節する
- 2016年6月号 モータータンパク質キネシンと神経変性
- 2016年5月号 チロシンリン酸化は Rhoファミリー GTPase 活性を調節する
- 2016年4月号 Rac1と糖尿病: ポジティブな役割とネガティブな役割
- 2016年3月号 SUMO化: 細胞骨格タンパク質の機能を調節するレギュレーター
- 2016年1月/2月号 ビメンチン中間径フィラメント: リン酸化による調節
2015年
- 2015年8月号 タンパク質調節に不可欠な翻訳後修飾
- 2015年7月号 アクチン細胞骨格のライブセルイメージング
- 2015年6月号 有糸分裂に関わるタンパク質のSUMO化: 局在と機能
- 2015年5月号 Ras 癌の治療: 5つの有望なターゲット
- 2015年4月号 Ras 依存性の癌で注目される YAP1
- 2015年3月号 増刊号 統合失調症において遺伝子変異により誘導されるアクチン依存のシナプスの変化
- 2015年3月号 Ral GTPase を調節する翻訳後修飾
- 2015年1月/2月号 RhoA のリン酸化はシグナル伝達を調節する
- 2015年1月号 増刊号 微小管を不安定化する suprafenacine: 新規抗癌剤のリード化合物としての可能性
2014年
- 2014年12月号 増刊号 RhoA は心筋細胞におけるアクチン細胞骨格の再構成とグルコース取り込みを仲介する
- 2014年11月号 増刊号 樹状突起の形態形成: ドーパミンD1受容体 および Rho ファミリー GTPase による制御
- 2014年11月/12月号 GTPase 活性化アッセイ: アイソフォームの検出
- 2014年10月号 アルギニンの正電荷を消失させるシトルリン化
- 2014年9月号 キネシンサブドメインの探索
- 2014年9月号 増刊号 アクチン結合タンパク質コフィリンの S-ニトロシル化: 細胞移動に対する影響
- 2014年8月号 増刊号 原発性硬化性胆管炎における N-Ras 発現および活性
- 2014年8月号 SUMO化: 細胞骨格タンパク質を標的とした翻訳後修飾
- 2014年7月号 Sos/K-Ras 結合を介して Ras シグナル伝達を制御する新しい低分子阻害剤
- 2014年6月号 増刊号 頭頸部扁平上皮癌における microRNA-138 による RhoC のダウンレギュレーション
- 2014年6月号 Rho GTPase と活性酸素種: クロストークとフィードバック
- 2014年5月号 ミオシンのアセチル化はサルコメアの構造と機能を調節する
- 2014年4月号 リジンのアセチル化 - 多様な細胞プロセスの制御因子
- 2014年3月号 インテグリンを介したβ-アクチンの酸化還元制御: PDIの出現
- 2014年1/2月号 ダイニン: 一つのモーターが関わる複数の神経変性疾患
2013年
- 2013年11/12月号 ダイニン:チームとして強力に作用するモータータンパク質
- 2013年10月号 神経変性:Rhes、SUMO化、ハンチントン病
- 2013年9月号 モノユビキチン化:タンパク質調節のダイナミックなタグ
- 2013年8月号 Ras及びRhoのプレニル化による翻訳後修飾:癌創薬における役割
- 2013年7月号 アクチンが引き起こす膜突起による浸潤:コルタクチン
- 2013年6月号 アクチン修飾と細胞骨格
- 2013年5月号 微小管内部の実体
- 2013年4月号 神経変性におけるTauの多面性
- 2013年3月号 蛍光フィブロネクチンタンパク質を用いた特発性肺線維症の創薬
- 2013年1/2月号 樹状突起棘:発生におけるArf6の役割
2012年
- 2012年11/12月号 ミオシンの小分子モジュレーター
- 2012年10月号 Rhoファミリーパスウェイのユビキチン化と制御
- 2012年9月号 神経変性におけるRac1 GTPaseの機能
- 2012年8月号 上皮間葉転換(EMT)とRhoファミリー低分子量G-タンパク質の関与
- 2012年7月号 チューブリンの多重修飾:グルタミル化とグリシル化
- 2012年6月号 細胞接着のフィブロネクチン制御と原線維形成
- 2012年5月号 アクチン酸化サイクルの機能
- 2012年4月号 トラフィッキング:ArfとCdc42/Racの結合
- 2012年3月号 G-LISAを用いた心臓研究: 糖尿病性心筋症におけるRho経路に関する研究
- 2012年1月/2月号 FtsZ: 新たな抗生剤の標的となるチュ−ブリンホモログ
商品は「研究用試薬」です。人や動物の医療用・臨床診断用・食品用としては使用しないように、十分ご注意ください。
※ 表示価格について























 このページを印刷する
このページを印刷する












