記事ID : 13304
SUMO化: 細胞骨格タンパク質を標的とした翻訳後修飾 CYTOSKELETON NEWS 2014年8月号

SUMO化: 細胞骨格タンパク質を標的とした翻訳後修飾
タンパク質は、翻訳が行われると細胞内の適切な位置に輸送され、それぞれの生理的機能を遂行できるようになります。タンパク質の正確な局在化や機能に関与するメカニズムの一つに、翻訳後修飾(PTMs)があります。PTM には、プロテアーゼによる切断、タンパク質フォールディング、生化学官能基(例: 酢酸、リン酸)や低分子量タンパク質(例: ユビキチン、SUMO[Small Ubiquitin-like Modifier: 低分子ユビキチン様修飾因子])の付加、などがあります。このように、PTM は、タンパク質の機能やシグナル伝達および寿命を、二次的に調節しているといえます1,2。PTM を介した制御により、タンパク質の結合モチーフに変化が生じる場合があり、他のタンパク質や核酸、および様々な小分子との結合に影響します。また、酵素を介した可逆的な PTM は、タンパク質が正確な制御下で多様な役割を果たすことができる、特に強力な調節方法であると言えます1,2。
SUMO タンパク質は、それぞれ独立した4つの研究グループ4-8 によって18年前に発見された 12kDa のタンパク質で3、発見されて以来、細胞プロセスの制御における役割が重点的に研究されてきました9,10。SUMO タンパク質は、酵母から哺乳類細胞まで高度に保存されています11。出芽酵母(Saccharomyces cerevisiae)では、一つの SUMO タンパク質 (Smt3) しか存在しませんが、脊椎動物では、3つの主要な SUMO アイソフォーム (SUMO-1、SUMO-2、SUMO-3) が存在し、全ての組織で発現しています12。成熟型 SUMO-2 と SUMO-3 はアミノ酸配列が 97% 同一(SUMO-1 とは 48% 同一)であり、機能的な差が確認されていないことから、まとめて SUMO-2/3 と呼ばれています13。SUMO タンパク質(SUMO-1 および SUMO-2/3)は、それぞれ細胞内局在が異なり、SUMO-2/3 は SUMO-1 より高い発現レベルを示します14-16。SUMO-1 は1分子のみが標的タンパク質に結合しますが、SUMO-2/3 は、複数が標的タンパク質のリジン残基に結合してポリ SUMO-2/3 鎖を形成し、SUMO-1 のライゲーションによって重合が終了します17,18。
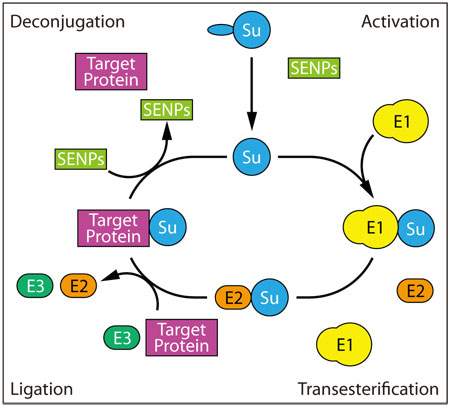
図1 SUMO化/脱SUMO化のサイクル
SUMO化(SUMO のイソペプチド結合を介した標的タンパク質への結合)は、一連の酵素反応からなります(図1)19。プロセシング酵素(SENPs)は、新たに合成された SUMO のC末端を切断し、ヘテロ二量体である E1 SUMO 活性化酵素(SAE1/2 サブユニット)は、 SUMO の露出したジグリシンと SAE2 の保存されたシステイン残基をチオエステル結合することにより、 SUMO を活性化します19,20。活性化された SUMO は、E2 結合酵素 (Ubc9) に運ばれます。ここで、SUMO は、2つの経路を介して標的タンパク質に結合します。一つは、SUMO-Ubc9 複合体が SUMO 結合モチーフ(SUMO interacting motif: SIM)を介して標的タンパク質に結合し、Ubc9 が SUMO の C末端と標的タンパク質リジン残基のεアミノ基とのイソペプチド結合を触媒する、E3 酵素非依存的な経路です22。もう一つは、PIAS ファミリーのメンバー23、RanBP224、Pc225 などの様々な E3 リガーゼが関与するメカニズムです24-28。SUMO-Ubc9 と E3 リガーゼが複合体を形成すると、E3 リガーゼは標的タンパク質に結合し、SUMO の標的タンパク質への結合を促進します29。多くの SUMO化反応は、コンセンサス配列 Ψ-K-x-D/E (Ψ; 疎水性アミノ酸、K; リジン、x; 任意のアミノ酸、 D/E; アスパラギン酸/グルタミン酸の酸性アミノ酸)上で起こりますが30、コンセンサス配列に合致しない部位でのSUMO化も報告されています31,32。
SUMO化は、機能上、Rac1 GTPアーゼおよび細胞骨格を構成するタンパク質であるアクチンとチューブリンなど、様々なタンパク質が標的となります33,34 (図2)。Rho ファミリー GTP アーゼである Rac1 の SUMO化は、SUMO E3 リガーゼ PIAS3 によって仲介され、様々な成長因子に応答した Rac1 の活性化、続いて起こる Rac-1 を介した葉状仮足/膜ラフリング(細胞移動や浸潤に関与)の形成に必要です34。ある研究グループは、SUMO化は Rac1 の活性化に必須ではありませんが、活性化の時間が延長されることを示唆しています。Rac1 の SUMO化は、in vitro および in vivo の両方で起こります。また、GTP 結合型および GDP 結合型 Rac1 のどちらも SUMO化されますが、活性化型 Rac1 が優先的に標的となります34。質量分析により、Rac1 の in vitro における SUMO化の 95% は、C末端の PBR (polybasic region)内にあるリジン 188、183、184、186 残基で起こることがわかりました34。また、in vivo においても、同じリジン残基がSUMO化の標的となります34。Rho ファミリー GTPアーゼの上流には、細胞骨格タンパク質であるアクチンが存在します。In vitro では、アクチンの SUMO2 および SUMO3 による SUMO化は核移行に関与し19,35,36、核アクチンは、アミノ酸 K68 と K284 の相互作用を介して、SUMO2 および SUMO3 によって修飾されます36。
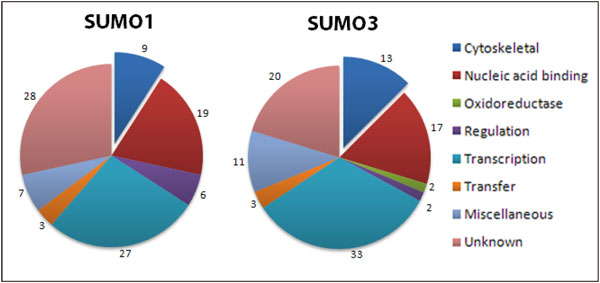
図2 SUMO化の標的となるタンパク質タイプの割合を示すチャート(プロテオーム解析による)
参考文献19より修正改変。
もう一つの主要な細胞骨格タンパク質であるチューブリンも、SUMO の標的となります。α-チューブリンの SUMO化は in vitro において確認されており、β-チューブリンは、SUMO-1 および SUMO-3 の標的であると推定されています19。ある研究グループは19、チューブリンの SUMO化には、多量体タンパク質複合体を組み立てるために、他の SUMO化タンパク質を隔離する役割があると仮定しています。
SUMOが介するタンパク質の局在化および機能の制御は、複雑なだけではなく、細胞構造や輸送の主要な調節因子として重要な役割を担っています。SUMO化によるタンパク質の制御のさらなる理解を目指して、Cytoskeleton 社は PTM に非常に特異的で高品質な抗体の開発を行っています。
参考文献
1. Yang X.J. 2005. Multisite protein modification and intramolecular signaling. Oncogene. 24, 1653-1662.
2. Flotho A. & Melchior F. 2013. Sumoylation: a regulatory protein modification in health and disease. Annu. Rev. Biochem. 82, 357-385.
3. Bayer P. et al. 1998. Structure determination of the small ubiquitin-related modifier SUMO-1. J. Mol. Biol. 280, 275-286.
4. Boddy M.N. et al. 1996. PIC 1, a novel ubiquitin-like protein which interacts with the PML component of a multiprotein complex that is disrupted in acute promyelocytic leukaemia. Oncogene. 13, 971-982.
5. Shen Z. et al. 1996. UBL1, a human ubiquitin-like protein associating with human RAD51/RAD52 proteins.Genomics. 36, 271-279.
6. Okura T. et al. 1996. Protection against Fas/APO-1- and tumor necrosis factor-mediated cell death by a novel protein, sentrin. J. Immunol. 157, 4277-4281.
7. Matunis M.J. et al. 1996. A novel ubiquitin-like modification modulates the partitioning of the Ran-GTPase-activating protein RanGAP1 between the cytosol and the nuclear pore complex. J. Cell. Biol. 135, 1457-1470.
8. Mahajan R. et al. 1997. A small ubiquitin-related polypeptide involved in targeting Ran-GAP1 to nuclear pore complex protein RanBP2. Cell. 88, 97-107.
9. Gareau J.R. & Lima C.D. 2010. The SUMO pathway: emerging mechanisms that shape specificity, conjugation and recognition. Nat. Rev. Mol. Cell Biol. 11, 861-871.
10. Jentsch S. & Psakhye I. 2013. Control of nuclear activities by substrate-selective and protein-group SUMOylation.Annu. Rev. Genet. 47, 167-186.
11. Chen A. et al. 1998. Characterization of mouse ubiquitin-like SMT3A and SMT3B cDNAs and gene/pseudogenes.Biochem. Mol. Biol. Int. 46, 1161-1174.
12. Huang W.C. et al. 2004. Crystal structures of the human SUMO-2 protein at 1.6 A and 1.2 A resolution: implication on the functional differences of SUMO proteins. Eur. J. Biochem. 271, 4114-4122.
13. Kamitani T. et al. 1998. Characterization of a second member of the sentrin family of ubiquitin-like proteins. J. Biol. Chem. 273, 11349-11353.
14. Saitoh H. & Hinchey J. 2000. Functional heterogeneity of small ubiquitin-related protein modifiers SUMO-1 versus SUMO-2/3. J. Biol. Chem. 275, 6252-6258.
15. Manza L.L. et al. 2004. Global shifts in protein sumoylation in response to electrophile and oxidative stress. Chem. Res. Toxicol. 17, 1706-1715.
16. Ayaydin F. & Dasso M. 2004. Distinct in vivo dynamics of vertebrate SUMO paralogues. Mol. Biol. Cell. 15, 5208-5218.
17. Bohren K.M. et al. 2004. A M55V polymorphism in a novel SUMO gene (SUMO-4) differentially activates heat shock transcription factors and is associated with susceptibility to type I diabetes mellitus. J. Biol. Chem. 279, 27233-27238.
18. Tatham M.H. et al. 2001. Polymeric chains of SUMO-2 and SUMO-3 are conjugated to protein substrates by SAE1/SAE2 and Ubc9. J. Biol. Chem. 276, 35368-35374.
19. Rosas-Acosta G. et al. 2005. A universal strategy for proteomic studies of SUMO and other ubiquitin-like modifiers. Mol. Cell. Proteomics. 4, 56-72.
20. Johnson E.S. et al. 1997. The ubiquitin-like protein Smt3p is activated for conjugation to other proteins by an Aos1p/Uba2p heterodimer. EMBO J. 16, 5509-5519.
21. Lois L.M. & Lima C.D. 2005. Structures of the SUMO E1 provide mechanistic insights into SUMO activation and E2 recruitment to E1. EMBO J. 24, 439-451.
22. Melchior F. 2000. SUMO--nonclassical ubiquitin. Annu. Rev. Cell Dev. Biol. 16, 591-626.
23. Johnson E.S. & Gupta A.A. 2001. An E3-like factor that promotes SUMO conjugation to the yeast septins. Cell. 106, 735-744.
24. Pichler A. et al. 2002. The nucleoporin RanBP2 has SUMO1 E3 ligase activity. Cell. 108, 109-120.
25. Kagey M.H. et al. 2003. The polycomb protein Pc2 is a SUMO E3. Cell. 113, 127-137.
26. Schmidt D. & Muller S. 2002. Members of the PIAS family act as SUMO ligases for c-Jun and p53 and repress p53 activity. Proc. Natl. Acad. Sci. U.S.A. 99, 2872-2877.
27. Ungureanu D. et al. 2003. PIAS proteins promote SUMO-1 conjugation to STAT1. Blood. 102, 3311-3313.
28. Sachdev S. et al. 2001. PIASy, a nuclear matrix-associated SUMO E3 ligase, represses LEF1 activity by sequestration into nuclear bodies. Genes Dev. 15, 3088-3103.
29. Lin D. et al. 2002. Identification of a substrate recognition site on Ubc9. J. Biol. Chem. 277, 21740-21748.
30. Hay R.T. 2005. SUMO: a history of modification. Mol. Cell. 18, 1-12.
31. Kamitani T. et al. 1998. Identification of three major sentrinization sites in PML. J. Biol. Chem. 273, 26675-26682.
32. Rangasamy D. et al. 2000. SUMO-1 modification of bovine papillomavirus E1 protein is required for intranuclear accumulation. J. Biol. Chem. 275, 37999-38004.
33. Panse V.G. et al. 2004. A proteome-wide approach identifies sumolyated substrate proteins in yeast. J. Biol. Chem. 279, 41346–41351.
34. Castillo-Lluva S. et al. 2010. SUMOylation of the GTPase Rac1 is required for optimal cell migration. Nat. Cell Biol. 12, 1078-1085.
35. Vertegaal A.C. et al. 2004. A proteomic study of SUMO-2 target proteins. J. Biol. Chem. 279, 33791-33798.
36. Hofmann W.A. et al. 2009. SUMOylation of nuclear actin. J. Cell Biol. 186, 193-200.
| 品名 | メーカー | 品番 | 包装 | 希望販売価格 |
|---|---|---|---|---|
Anti Acetyl Lysine, (Mouse) , 3C6.08.20 |
CYT | AAC01 | 200 UL [2 x 100 μl] |
¥125,000 |
Chemically Acetylated Serum Albumin (BSA), Bovine |
CYT | AACX1 | 1*500 UG |
CYT社 AACX1 1000 を参照 |
Tricostatin A (TSA) treated and untreated cell lysates |
CYT | AACX2 | 200 UG [2 x 100 μg] |
¥34,000 |
RhoA Pulldown Activation Assay Kit |
CYT | BK036 | 1 KIT [80 assays] |
¥269,000 |
RhoA Pulldown Activation Assay Kit |
CYT | BK036-S | 1 KIT [20 assays] |
¥124,000 |
RhoA G-LISA(R) Activation Assay (absorbance) |
CYT | BK124 | 96 ASSAY |
¥302,000 |
【関連商品】
- 抗アセチルリジン マウスモノクローナル抗体
-翻訳後修飾されたアセチル化リジンを幅広く検出 - 低分子量タンパク質活性アッセイ Biochem Kit™
-低分子量Gタンパク質活性化を、プルダウンアッセイで簡単に測定! - G-LISA® 低分子量Gタンパク質活性アッセイキット
-少量のサンプルから、3時間以内で低分子量Gタンパク質の活性を測定!
■ CYTOSKELETON NEWS バックナンバー
- 2020年10月号 紡錘体 - 可視化に向けた新規ツール
- 2020年8月号 細胞膜染色用蛍光プローブ
- 2020年4月号 生細胞におけるF-アクチンプローブ
- 2020年3月号 コロナウイルスと細胞骨格
- 2020年2月号 タウ(Tau)の将来性をMapping
- 2020年1月号 Rho GTPaseによる細胞遊走制御
- 2019年12月号 表現型プロファイリング:アクチンに焦点を当てたがん治療
- 2019年11月号 チューブリンの過剰グルタミル化、ミトコンドリア、神経変性
- 2019年9月号 細胞運動性を制御するために相互作用する膜張力とアクチン細胞骨格
- 2019年8月号 Rac1B、がん、およびRac1
- 2019年7月号 Rhoファミリー GTPases、神経可塑性、およびうつ状態
- 2019年6月号 アクチンメチオニン酸化: 動的制御の次の段階
- 2019年5月号 ミクログリアと神経変性疾患
- 2019年2月号 生細胞画像化に対するCNS疾患や障害
2018年
- 2018年12月号 アクチン細胞骨格とメカノトランスダクション(機械的シグナル伝達)
- 2018年11月号 軸索再生と細胞骨格
- 2018年10月号 ニューロンにおける微小管と極性
- 2018年8月号 Rab GTPase と 神経変性
- 2018年7月号 SUMO レスリング: バランスが全て
- 2018年6月号 なぜ K-Ras は発がん特異性を示すのか?
- 2018年5月号 治療標的としてのユビキチンプロテアソームシステム:チューブリンは関与するか?
- 2018年4月号 RhoファミリーGEFと樹状突起スパインの構造的可塑性
- 2018年3月号 βカテニンとTFC/LEF-1の翻訳後修飾による標準的なWntシグナル制御
- 2018年2月号 がん抑制遺伝子p53の翻訳後修飾による機能の調整
- 2018年1月号 自閉スペクトラム症におけるGEF Trioの役割
2017年
- 2017年12月号 プロフィリン: アクチン結合タンパク質の多機能な役割
- 2017年11月号 ミトコンドリアにおけるアセチル化:新たな考え方と治療への応用の可能性
- 2017年9月号 翻訳後修飾のアセチル化による微小管の安定化
- 2017年8月号 神経軸索におけるアクチンリングを基盤とした周期的膜骨格(PMS)
- 2017年7月号 E3ユビキチンリガーゼMdm2によるがん抑制遺伝子p53の翻訳後制御
- 2017年6月号 多能性幹細胞(PSC)での転写因子による翻訳後制御
- 2017年5月号 Arf6 GEF と癌細胞の浸潤・転移
- 2017年4月号 PTEN(Phosphatase and Tensin Homolog)による翻訳後制御
- 2017年3月号 Tau の翻訳後修飾: アルツハイマー病の治療標的
- 2017年2月号 樹状細胞の移動におけるアクチン結合タンパク質とF-アクチン
2016年
- 2016年11月/12月号 GEF を介した GTPase シグナル伝達の低分子阻害剤
- 2016年9月号 FtsZ タンパク質: 抗菌薬の新規ターゲット
- 2016年7月号 翻訳後修飾(PTM)は心臓病において細胞骨格タンパク質を調節する
- 2016年6月号 モータータンパク質キネシンと神経変性
- 2016年5月号 チロシンリン酸化は Rhoファミリー GTPase 活性を調節する
- 2016年4月号 Rac1と糖尿病: ポジティブな役割とネガティブな役割
- 2016年3月号 SUMO化: 細胞骨格タンパク質の機能を調節するレギュレーター
- 2016年1月/2月号 ビメンチン中間径フィラメント: リン酸化による調節
2015年
- 2015年8月号 タンパク質調節に不可欠な翻訳後修飾
- 2015年7月号 アクチン細胞骨格のライブセルイメージング
- 2015年6月号 有糸分裂に関わるタンパク質のSUMO化: 局在と機能
- 2015年5月号 Ras 癌の治療: 5つの有望なターゲット
- 2015年4月号 Ras 依存性の癌で注目される YAP1
- 2015年3月号 増刊号 統合失調症において遺伝子変異により誘導されるアクチン依存のシナプスの変化
- 2015年3月号 Ral GTPase を調節する翻訳後修飾
- 2015年1月/2月号 RhoA のリン酸化はシグナル伝達を調節する
- 2015年1月号 増刊号 微小管を不安定化する suprafenacine: 新規抗癌剤のリード化合物としての可能性
2014年
- 2014年12月号 増刊号 RhoA は心筋細胞におけるアクチン細胞骨格の再構成とグルコース取り込みを仲介する
- 2014年11月号 増刊号 樹状突起の形態形成: ドーパミンD1受容体 および Rho ファミリー GTPase による制御
- 2014年11月/12月号 GTPase 活性化アッセイ: アイソフォームの検出
- 2014年10月号 アルギニンの正電荷を消失させるシトルリン化
- 2014年9月号 キネシンサブドメインの探索
- 2014年9月号 増刊号 アクチン結合タンパク質コフィリンの S-ニトロシル化: 細胞移動に対する影響
- 2014年8月号 増刊号 原発性硬化性胆管炎における N-Ras 発現および活性
- 2014年8月号 SUMO化: 細胞骨格タンパク質を標的とした翻訳後修飾
- 2014年7月号 Sos/K-Ras 結合を介して Ras シグナル伝達を制御する新しい低分子阻害剤
- 2014年6月号 増刊号 頭頸部扁平上皮癌における microRNA-138 による RhoC のダウンレギュレーション
- 2014年6月号 Rho GTPase と活性酸素種: クロストークとフィードバック
- 2014年5月号 ミオシンのアセチル化はサルコメアの構造と機能を調節する
- 2014年4月号 リジンのアセチル化 - 多様な細胞プロセスの制御因子
- 2014年3月号 インテグリンを介したβ-アクチンの酸化還元制御: PDIの出現
- 2014年1/2月号 ダイニン: 一つのモーターが関わる複数の神経変性疾患
2013年
- 2013年11/12月号 ダイニン:チームとして強力に作用するモータータンパク質
- 2013年10月号 神経変性:Rhes、SUMO化、ハンチントン病
- 2013年9月号 モノユビキチン化:タンパク質調節のダイナミックなタグ
- 2013年8月号 Ras及びRhoのプレニル化による翻訳後修飾:癌創薬における役割
- 2013年7月号 アクチンが引き起こす膜突起による浸潤:コルタクチン
- 2013年6月号 アクチン修飾と細胞骨格
- 2013年5月号 微小管内部の実体
- 2013年4月号 神経変性におけるTauの多面性
- 2013年3月号 蛍光フィブロネクチンタンパク質を用いた特発性肺線維症の創薬
- 2013年1/2月号 樹状突起棘:発生におけるArf6の役割
2012年
- 2012年11/12月号 ミオシンの小分子モジュレーター
- 2012年10月号 Rhoファミリーパスウェイのユビキチン化と制御
- 2012年9月号 神経変性におけるRac1 GTPaseの機能
- 2012年8月号 上皮間葉転換(EMT)とRhoファミリー低分子量G-タンパク質の関与
- 2012年7月号 チューブリンの多重修飾:グルタミル化とグリシル化
- 2012年6月号 細胞接着のフィブロネクチン制御と原線維形成
- 2012年5月号 アクチン酸化サイクルの機能
- 2012年4月号 トラフィッキング:ArfとCdc42/Racの結合
- 2012年3月号 G-LISAを用いた心臓研究: 糖尿病性心筋症におけるRho経路に関する研究
- 2012年1月/2月号 FtsZ: 新たな抗生剤の標的となるチュ−ブリンホモログ
商品は「研究用試薬」です。人や動物の医療用・臨床診断用・食品用としては使用しないように、十分ご注意ください。
※ 表示価格について























 このページを印刷する
このページを印刷する













